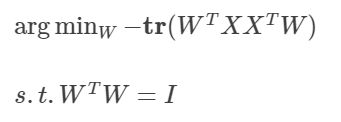(系列笔记)27.主成分分析——PCA(下)
文章目录
- PCA——用 SVD 实现 PCA
- PCA 优化算法
- 算法一,拉格朗日乘子法:
- 算法二
- PCA 的作用
- 奇异值分解(Singular Value Decomposition, SVD)
- SVD的三个矩阵
- 三个矩阵间的关系
- SVD的计算
- 用 SVD 实现 PCA
- 直接用 SVD 降维
- SVD & PCA 实例
- SVD压缩图片
- PCA压缩图片
PCA——用 SVD 实现 PCA
PCA 优化算法
算法一,拉格朗日乘子法:
令:
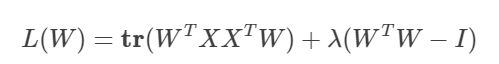
然后对W求导,并令导函数为0可得:
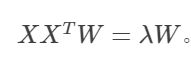
这是一个标准的特征方程求解问题,只需要对协方差矩阵 X X T XX^T XXT进行特征值分解,将求得的特征值排序: λ 1 ≥ λ 2 ≥ . . . ≥ λ d \lambda_1\ge\lambda_2\ge...\ge\lambda_d λ1≥λ2≥...≥λd,再取前 d ′ d' d′个特征对应的特征向量构成 W = ( w 1 , w 2 , . . . , w d ′ ) W=(w_1,w_2,...,w_{d'}) W=(w1,w2,...,wd′)即可。这样就求出了W,即为主成分分析的解。
算法描述:
【输入】:
- d维空间中n个样本数据的集和D={ x ( 1 ) , x ( 2 ) , . . . , x ( n ) x^{(1)},x^{(2)},...,x^{(n)} x(1),x(2),...,x(n)}
- 低维空间的维数 d ′ d' d′,这个数值通常由用户指定。
【过程】:
- 对所有原始样本做中心话: x ( i ) : = x ( i ) − 1 n ∑ i = 1 n x ( i ) x^{(i)}:=x^{(i)}-\frac{1}{n}\sum_{i=1}^{n}{x^{(i)}} x(i):=x(i)−n1∑i=1nx(i)
- 计算样本的协方差矩阵: X X T XX^T XXT
- 协方差矩阵 X X T XX^T XXT进行特征值分解;
- 取最大的 d ′ d' d′个特征值对应的特征向量 w ( 1 ) , w ( 2 ) , . . . , w ( d ′ ) w^{(1)},w^{(2)},...,w^{(d')} w(1),w(2),...,w(d′)
【输出】:
W = ( w ( 1 ) , w ( 2 ) , . . . , w ( d ′ ) ) W=(w^{(1)},w^{(2)},...,w^{(d')}) W=(w(1),w(2),...,w(d′))
算法二
上述求解过程可以换一个角度来看。
先对协方差矩阵 ∑ i = 1 n x ( i ) x ( i ) T \sum_{i=1}^nx^{(i)}{x^{(i)}}^T ∑i=1nx(i)x(i)T做特征值分解,取最大特征值对应的特征向量 w 1 w_1 w1; 再对 ∑ i = 1 n x ( i ) x ( i ) T − λ 1 w 1 w 1 T \sum_{i=1}^nx^{(i)}{x^{(i)}}^T-\lambda_1w_1w_1^T ∑i=1nx(i)x(i)T−λ1w1w1T做特征值分解,取其最大特征值对应的特征向量 w 2 w_2 w2;以此类推。因为 W 的各个分量正交,因此 ∑ i = 1 n x ( i ) x ( i ) T = ∑ j = 1 d λ 1 w 1 w 1 T \sum_{i=1}^nx^{(i)}{x^{(i)}}^T=\sum_{j=1}^d\lambda_1w_1w_1^T ∑i=1nx(i)x(i)T=∑j=1dλ1w1w1T
故而解法二和解法一等价。
PCA 的作用
PCA 将 d 维的原始空间数据转换成了d′ 维的新空间数据,无疑丧失了部分数据。
根据上面讲的算法我们知道,经过 PCA 后,原样本集协方差矩阵进行特征值分解后,倒数(d−d′) 个特征值对应的特征向量被舍弃了。
因为舍弃了这部分信息,导致的结果是:
- 样本的采样密度增大——这是降维的首要动机;]
- 最小的那部分特征值所对应的特征向量往往与噪声有关,舍弃它们有降噪的效果。
奇异值分解(Singular Value Decomposition, SVD)
SVD 是线性代数中一种重要的矩阵分解方法,在信号处理、统计学等领域有重要应用。
SVD的三个矩阵
我们先来看看 SVD 方法本身。假设 M 是一个 m×n 阶实矩阵,则存在一个分解使得:
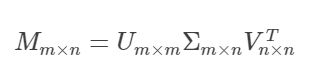
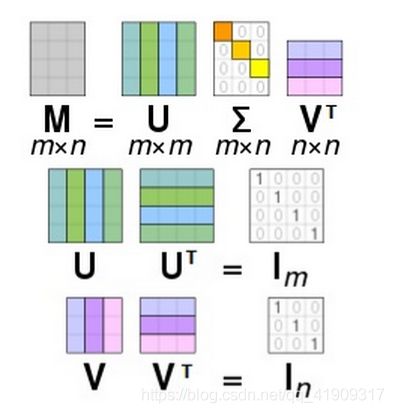
其中,Σ 是一个m×n 的非负实数对角矩阵,Σ 对角线上的元素是矩阵 M 的奇异值:
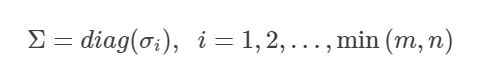
注意:对于一个非负实数 σ 而言,仅当存在 m 维的单位向量 u 和 n 维的单位向量v,它们和 M 及 σ 有如下关系时: M v = σ u Mv=σu Mv=σu 且 M T u = σ v M^Tu=σv MTu=σv
我们说 σ 是 M 矩阵的奇异值,向量 u 和 v 分别称为 σ 的左奇异向量和右奇异向量。
一个 m×n 的矩阵至多有 min(m,n) 个不同的奇异值
U是一个m×m的酉矩阵,它是一组由 M 的左奇异向量组成的正交基: U = ( u 1 , u 2 , . . . , u m ) U=(u_1,u_2,...,u_m) U=(u1,u2,...,um)。
它的每一列 u i u_i ui都是 ∑ \sum ∑中对应序号的对角值 σ i \sigma_i σi关于M的左奇异向量。
V是一个n×n的酉矩阵,它是一组由 M 的右奇异向量组成的正交基: V = ( v 1 , v 2 , . . . , v n ) V=(v_1,v_2,...,v_n) V=(v1,v2,...,vn)。
它的每一列 u i u_i ui都是 ∑ \sum ∑中对应序号的对角值 σ i \sigma_i σi关于M的右奇异向量。
酉矩阵:若一个n×n的实数方阵U满足 U T U = U U T = I n U^TU=UU^T=I_n UTU=UUT=In,则称为酉矩阵。
三个矩阵间的关系
我们这样看来:
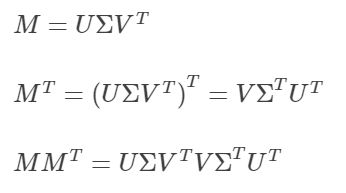
又因为U和V都是酉矩阵,所以:
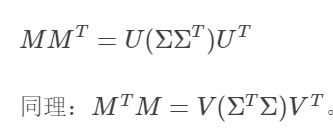
也就是说U的列向量是 M M T MM^T MMT的特征向量,V的列向量是 M T M M^TM MTM的特征向量;而 ∑ \sum ∑的对角元素,是M的奇异值,也是 M M T MM^T MMT或者 M T M M^TM MTM的非零特征值的平方根。
SVD的计算
手动计算过程:
- 计算 M M T MM^T MMT和 M T M M^TM MTM;
- 分别计算 M M T MM^T MMT和 M T M M^TM MTM的特征向量及其特征值;
- 用 M M T MM^T MMT的特征向量组成U, M T M M^TM MTM的特征向量组成V;
- 对 M M T MM^T MMT和 M T M M^TM MTM的非零特征值求平方根,对应上述特征向量的位置,填入 ∑ \sum ∑的对角元。
更详细的过程可以看:http://web.mit.edu/be.400/www/SVD/Singular_Value_Decomposition.htm
用 SVD 实现 PCA
所谓降维度,就是按照重要性排列现有特征,舍弃不重要的,保留重要的。
上面讲了 PCA 的算法,很关键的一步就是对协方差矩阵进行特征值分解,不过其实在实践当中,我们通常用对样本矩阵 X 进行奇异值分解来代替这一步。
X 是原空间的样本矩阵,W 是投影矩阵,而 T 是降维后的新样本矩阵,有 T = X W T=XW T=XW。
我们直接对X做SVD,得到: T = X W = U ∑ W T W T=XW=U\sum W^TW T=XW=U∑WTW
因为W是标准正交基组成的矩阵,因此: T = U ∑ W T W = U ∑ T=U\sum W^TW=U\sum T=U∑WTW=U∑。
我们选矩阵U前d’列,和 ∑ \sum ∑左上角前d’×d’区域内的对角值,也就是前d’打的奇异值,然后直接降维: T d ′ = U d ′ ∑ d ′ T_{d'}=U_{d'}\sum_{d'} Td′=Ud′∑d′
这样做很容易解释其物理意义:样本数据的特征重要性程度既可以用特征值来表征,也可以用奇异值来表征。
动机也很清楚,当然是成本。直接做特征值分解需要先求出协方差矩阵,当样本或特征量大的时候,计算量很大。更遑论对这样复杂的矩阵做特征值分解的难度了。
而对矩阵M进行SVD时,直接对 M M T MM^T MMT做特征值分解,要简单得多。
当然SVD算法本身也是一个接近 O ( m 3 ) O(m^3) O(m3)(假设m>n)时间复杂度的运算,不过现在SVD的并行运算已经被实现,效率也因此提高不少。
直接用 SVD 降维
除了可以用于 PCA 的实现,SVD 还可以直接用来降维。
在现实应用中,SVD 也确实被作为降维算法大量使用。
有一些应用,直接用眼睛就能看得见。比如:用 SVD 处理图像,减少图片信息量,而又尽量不损失关键信息。
图片是由像素(Pixels)构成的,一般彩色图片的单个像素用三种颜色(Red、Green、Blue)描述,每一个像素点对应一个 RGB 三元值。一张图片可以看作是像素的二维点阵,正好可以对应一个矩阵。那么我们用分别对应 RGB 三种颜色的三个实数矩阵,就可以定义一张图片。
设 X R , X G , X B X_R,X_G,X_B XR,XG,XB是用来表示一张图片的RGB数值矩阵,我们对其做SVD:
X R = U R ∑ R V R T X_R=U_R\sum_RV_R^T XR=UR∑RVRT
然后我们制定一个参数: k , U R k,U_R k,UR和 V R V_R VR取前k列,形成新的矩阵 U R k U_R^k URk和 V R k V_R^k VRk, ∑ R \sum_R ∑R取左上k×k的区域,形成新矩阵 ∑ R k \sum_R^k ∑Rk,然后用它们生成新的矩阵:
X R ′ = U R k ∑ R k ( V R k ) T X_R^{'}=U_R^k\sum_R^k{(V_R^k)}^T XR′=URk∑Rk(VRk)T
对 X G , X B X_G,X_B XG,XB做同样的事情,最后形成的 X R ′ , X G ′ , X B ′ X'_R,X'_G,X'_B XR′,XG′,XB′定义的图片,就是压缩了信息量后的图片。
如此处理后的图片尺寸未变,也就是说 X R ′ , X G ′ , X B ′ X'_R,X'_G,X'_B XR′,XG′,XB′与原本的 X R , X G , X B X_R,X_G,X_B XR,XG,XB行列数一致,只不过矩阵承载的信息量变小了。
比如, X R X_R XR是一个m×n矩阵,那么 U R U_R UR是一个m×m矩阵, ∑ R \sum_R ∑R是一个m×n矩阵,而 V R T V^T_R VRT是一个n×n矩阵, U R k U^k_R URk是一个m×k矩阵, ∑ R k \sum^k_R ∑Rk是一个k×k矩阵,而 ( V R k ) T {(V^k_R)}^T (VRk)T是一个k×n矩阵,它们相乘的矩阵仍然是m×n矩阵。从数学上讲,经过 SVD 重构后的新矩阵,相对于原矩阵秩(Rank)下降了。
SVD & PCA 实例
SVD压缩图片
我们用 SVD 分解上面这张图片,设不同的 k 值,来看分解后的结果。
下面几个结果的 k 值分别是:100、50、20和5。很明显,k 取 100 的时候,损失很小,取 50 的时候还能看清大致内容,到了 20 就模糊得只能看轮廓了,到了 5 则是一片条纹。

code
import os
import threading
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.image as mpimg
import sys
def svdImageMatrix(om, k):
U, S, Vt = np.linalg.svd(om)
cmping = np.matrix(U[:, :k]) * np.diag(S[:k]) * np.matrix(Vt[:k,:])
return cmping
def compressImage(image, k):
redChannel = image[..., 0]
greenChannel = image[..., 1]
blueChannel = image[..., 2]
cmpRed = svdImageMatrix(redChannel, k)
cmpGreen = svdImageMatrix(greenChannel, k)
cmpBlue = svdImageMatrix(blueChannel, k)
newImage = np.zeros((image.shape[0], image.shape[1], 3), 'uint8')
newImage[..., 0] = cmpRed
newImage[..., 1] = cmpGreen
newImage[..., 2] = cmpBlue
return newImage
path = 'liye.jpg'
img = mpimg.imread(path)
title = "Original Image"
plt.title(title)
plt.imshow(img)
plt.show()
weights = [100, 50, 20, 5]
for k in weights:
newImg = compressImage(img, k)
title = " Image after = %s" %k
plt.title(title)
plt.imshow(newImg)
plt.show()
newname = os.path.splitext(path)[0] + '_comp_' + str(k) + '.jpg'
mpimg.imsave(newname, newImg)
PCA压缩图片
使用from sklearn.decomposition import PCA
过程部分,只要将上面代码中的svdImageMatrix() 替换为如下 pca() 即可:
def pca(om, cn):
ipca = PCA(cn).fit(om)
img_c = ipca.transform(om)
print img_c.shape
print np.sum(ipca.explained_variance_ratio_)
temp = ipca.inverse_transform(img_c)
print temp.shape
return temp
cn 对应 sklearn.decomposition.PCA 的 n_components 参数,指的是 PCA 算法中所要保留的主成分个数 n,也就是保留下来的特征个数 n。
我们仍然压缩四次,具体的 cn 值还是 [100, 50, 20, 5]。
从运行的结果来看,和 SVD 差不多。

其实好好看看 sklearn.decomposition.PCA 的代码,不难发现,它其实就是用 SVD 实现的。