数据预处理,降维,特征提取,聚类
数据预处理,降维,特征提取,聚类
数据预处理,使用StandardScaler进行数据的预处理
# 导入numpy
import numpy
# 导入绘图工具
import matplotlib.pyplot as plt
%matplotlib inline
# 导入数据集生成工具
from sklearn.datasets import make_blobs
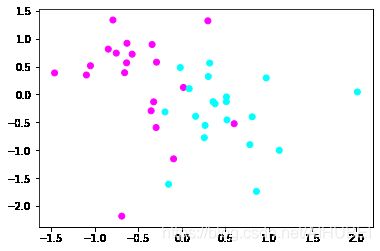
X, y = make_blobs(n_samples=40, centers=2, random_state=50, cluster_std=2)
# 使用散点图绘制数据
plt.scatter(X[:,0], X[:,1], c = y, cmap = plt.cm.cool)
plt.show()
样本数为40,分类centers为2,随机状态random_state为50,标准差cluster_std为2的数据。
使用sklearn的preprocessing模块对这个手工生成的数据集进行处理
# 导入StandardScaler
from sklearn.preprocessing import StandardScaler
# 使用StandardScaler进行数据预处理
X_1 = StandardScaler().fit_transform(X)
# 使用散点图绘制经过数据预处理的数据点
plt.scatter(X_1[:,0], X_1[:,1], c = y, cmap=plt.cm.cool)
# 显示图像
plt.show()
似乎没有什么变化,但是这里的 x x x和 y y y轴发生了变化,现在的数据所有的特征1的数值都在-2到3之间,特征2的数值都在-3到2之间。因为StandardScaler的原理就是,把所有的数据的特征值转化为;均值为零,方差为一的状态。这样一来就可以确保数据的大小都是一致的,更有利于模型的训练。
使用MinMaxScaler进行数据预处理
# 导入MinMaxScaler
from sklearn.preprocessing import MinMaxScaler
# 使用MinMaxScaler进行数据预处理
X_2 = MinMaxScaler().fit_transform(X)
# 绘制散点图
plt.scatter(X_2[:,0],X_2[:,1],c=y,cmap=plt.cm.cool)
plt.show()
这样一来,所有的数据每个特征值都被转换到0和1之间。这会使得模型的训练速度加快,同时准确率也会得以提高。
使用RobustScaler进行数据预处理
# 导入RobustScaler
from sklearn.preprocessing import RobustScaler
# 使用RobustScaler进行数据预处理
X_3 = RobustScaler().fit_transform(X)
# 绘制散点图
plt.scatter(X_3[:,0], X_3[:,1], c=y, cmap=plt.cm.cool)
plt.show()
这种方法和StandarScaler比较近似,但是他使用的是中位数和四分位数。他会直接把一些异常值剔除,有点类似于“去掉一个最高分,去掉一个最低分”
使用Normalizer进行数据预处理
# 导入Normalizer
from sklearn.preprocessing import Normalizer
# 使用Normalizer进行数据预处理
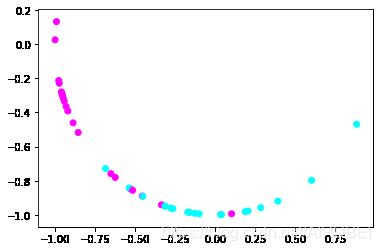
X_4 = Normalizer().fit_transform(X)
# 绘制散点图
plt.scatter(X_4[:,0], X_4[:,1], c=y, cmap=plt.cm.cool)
plt.show()
Normalizer方法讲数据或者说所有样本的特征向量转化为欧几里得距离为1.也就是把数据的分布变成一个半径为1的圆, 或者是一个球。通常在,我们只想保留数据的特征向量的方向,而忽略其数值的时候使用。
使用scale函数,对单个类似数组的数据集执行操作
import sklearn.preprocessing
import numpy as np
X_train = np.array([[1, -1, 2],
[2, 0, 0],
[0, 1, -1]])
X_scaled = sklearn.preprocessing.scale(X_train)
plt.scatter(X_train[:2,:],X_train[1:3,:])
plt.show()
X_scaled
array([[ 0. , -1.22474487, 1.33630621],
[ 1.22474487, 0. , -0.26726124],
[-1.22474487, 1.22474487, -1.06904497]])
plt.scatter(X_scaled[:2,:],X_scaled[1:3,:])
plt.show()
print("均值:",X_scaled.mean(axis = 0))
print("单位方差:",X_scaled.std(axis = 0))
均值: [0. 0. 0.]
单位方差: [1. 1. 1.]
标度数据的均值和单位方差为零
使用MaxAbsScaler进行数据预处理
# 导入MaxAbsScaler
from sklearn.preprocessing import MaxAbsScaler
# 使用Normalizer进行数据预处理
X_5 = MaxAbsScaler().fit_transform(X)
# 绘制散点图
plt.scatter(X_5[:,0], X_5[:,1], c=y, cmap=plt.cm.cool)
plt.show()
通过其最大绝对值缩放每个特征。分别缩放和转换每个特征,以使训练集中每个特征的最大绝对值为1.0。它不会移动/居中数据,因此不会破坏任何稀疏性。
该缩放器也可以应用于稀疏CSR或CSC矩阵。
print(X_5.mean())
print(X_5.std())
-0.3163704548637977
0.306489301692821
处理类别特征 使用OrdinalEncoder
通常,特征不是连续值而是类别型的。比如一个人的特征,这样的特征可以有效的编码为整数。使用OrdinalEncoder,实现讲类别特征编码到:(0 to n_categories -1)
# 导入OrdinalEncoder
from sklearn.preprocessing import OrdinalEncoder
# 使用OrdinalEncoder编码数据
Y = [['male', 'from US', 'uses Safari'],
['female', 'from Europe', 'uses Firefox']]
Y_ordinal = OrdinalEncoder().fit_transform(Y)
Y_ordinal
array([[1., 1., 1.],
[0., 0., 0.]])
但是,此类整数表示不能直接与所有scikit-learn估计器一起使用,因为它们期望连续输入,并且会将类别解释为有序的,这通常是不希望的(即,浏览器集是任意定序的)。
处理类别特征 使用独热码OneHotEncoder
# 导入OneHotEncoder
from sklearn.preprocessing import OneHotEncoder
# 使用OneHotEncoder处理数据
Y = [['male', 'from US', 'uses Safari'],
['female', 'from Europe', 'uses Firefox']]
Y_OneHot = OneHotEncoder().fit_transform(Y)
Y_OneHot
<2x6 sparse matrix of type ''
with 6 stored elements in Compressed Sparse Row format>
Y_OneHot.toarray()
array([[0., 1., 0., 1., 0., 1.],
[1., 0., 1., 0., 1., 0.]])
OneHotEncoder().fit(Y)
OneHotEncoder(categories='auto', drop=None, dtype=,
handle_unknown='error', sparse=True)
OneHotEncoder().categories
'auto'
它将带有n_categories可能值的每个分类特征 转换为n_categories二进制特征,其中一个为1,所有其他为0
# 可以使用参数明确指定categories
genders = ['female', 'male']
locations = ['from Africa', 'from Asia', 'from Europe', 'from US']
browsers = ['uses Chrome', 'uses Firefox', 'uses IE', 'uses Safari']
# 特征
X = [['male', 'from US', 'uses Safari'], ['female', 'from Europe', 'uses Firefox']]
X_fit = OneHotEncoder(categories=[genders, locations, browsers]).fit_transform([['female', 'from Asia', 'uses Chrome']]).toarray()
X_fit
array([[1., 0., 0., 1., 0., 0., 1., 0., 0., 0.]])
X_fit = OneHotEncoder(categories=[genders, locations, browsers]).fit_transform(X).toarray()
X_fit
array([[0., 1., 0., 0., 0., 1., 0., 0., 0., 1.],
[1., 0., 0., 0., 1., 0., 0., 1., 0., 0.]])
如果训练数据有可能缺少分类特征,则通常最好指定handle_unknown='ignore’而不是categories如上所述手动设置。如果 handle_unknown='ignore’指定且在转换过程中遇到未知类别,则不会引发错误,但是此功能生成的一键编码列将全为零(handle_unknown='ignore’仅一键编码支持):
# 数据/特征
X = [['male', 'from US', 'uses Safari'], ['female', 'from Europe', 'uses Firefox']]
X_fit = OneHotEncoder(handle_unknown='ignore').fit(X)
X_fit
OneHotEncoder(categories='auto', drop=None, dtype=,
handle_unknown='ignore', sparse=True)
X_fit = OneHotEncoder(handle_unknown='ignore').fit_transform(X).toarray()
X_fit
array([[0., 1., 0., 1., 0., 1.],
[1., 0., 1., 0., 1., 0.]])
使用Binarizer进行特征二值化,特征二值化是阈值化数字特征以获得布尔值的过程
# 在文本处理中使用二值化特征很常见,即使在实践归一化数据的表现要好一些。
X = [[ 1., -1., 2.],
[ 2., 0., 0.],
[ 0., 1., -1.]]
# 导入Binarizer
from sklearn.preprocessing import Binarizer
# 使用Binarizer处理数据
X_Bina = Binarizer().fit_transform(X)
X_Bina
array([[1., 0., 1.],
[1., 0., 0.],
[0., 1., 0.]])
# 可以调整二值化器的阈值 将数据进行二值化,threshold表示大于阈值的数据为1,小于阈值的数据为0
# 使用Binarizer处理数据
X_Bina = Binarizer(threshold=1).fit_transform(X)
X_Bina
array([[0., 0., 1.],
[1., 0., 0.],
[0., 0., 0.]])
缺失值的估算
由于各种原因,许多现实世界的数据集包含缺失值,通常将其编码为空白,NaN或其他占位符。但是,此类数据集与scikit-learn估计器不兼容,后者假定数组中的所有值都是数字,并且都具有并具有含义。使用不完整数据集的基本策略是丢弃包含缺失值的整个行和/或列。但是,这是以丢失有价值的数据为代价的(即使数据不完整)。更好的策略是估算缺失值,即从数据的已知部分推断出缺失值。参见 插补的通用术语表和API元素条目。
单变量特征插补
该SimpleImputer课程提供了估算缺失值的基本策略。可以使用提供的恒定值或使用缺失值所在各列的统计信息(平均值,中位数或最频繁)来估算缺失值。此类还允许使用不同的缺失值编码。
# 使用np.nan编码为缺失值,使用各列的均值来替换缺失值
import numpy as np
from sklearn.impute import SimpleImputer
imp = SimpleImputer(missing_values=np.nan, strategy='mean')
imp.fit([[1,2], [np.nan, 3], [7,6]])
SimpleImputer(add_indicator=False, copy=True, fill_value=None,
missing_values=nan, strategy='mean', verbose=0)
X = [[np.nan, 2],[6, np.nan],[7, 6]]
print(imp.transform(X))
[[4. 2. ]
[6. 3.66666667]
[7. 6. ]]
X = [[np.nan, 2],[6, np.nan],[7, 6]]
X_impute = SimpleImputer().fit_transform(X)
X_impute
array([[6.5, 2. ],
[6. , 4. ],
[7. , 6. ]])
SimpleImputer同时也支持稀疏矩阵
import scipy.sparse as sp
X = sp.csc_matrix([[1,2],[0,-1],[8,4]])
X
<3x2 sparse matrix of type ''
with 5 stored elements in Compressed Sparse Column format>
X_sparse = SimpleImputer(missing_values = -1, strategy='mean').fit_transform(X)
X_sparse
<3x2 sparse matrix of type ''
with 5 stored elements in Compressed Sparse Column format>
print(X_sparse.toarray())
[[1. 2.]
[0. 3.]
[8. 4.]]
SimpleImputer同时也可以用于处理分类型的数据
import pandas as pd
df = pd.DataFrame([["a", "x"],
[np.nan, "y"],
["a", np.nan],
["b", "y"]], dtype="category")
# 使用出现频率最高的数据进行填补空值
df_IMputer = SimpleImputer(strategy="most_frequent").fit_transform(df)
print(df_IMputer)
[['a' 'x']
['a' 'y']
['a' 'y']
['b' 'y']]
多元特征插补
import numpy as np
from sklearn.experimental import enable_iterative_imputer
from sklearn.impute import IterativeImputer
imp = IterativeImputer(max_iter=10, random_state=0)
imp.fit([[1, 2], [3, 6], [4, 8], [np.nan, 3], [7, np.nan]])
X_test = [[np.nan, 2], [6, np.nan], [np.nan, 6]]
# the model learns that the second feature is double the first
print(np.round(imp.transform(X_test)))
[[ 1. 2.]
[ 6. 12.]
[ 3. 6.]]
# 显示的导入enable_iterative_imputer后
# 再从sklearn.impute 导入IterativeImputer
# from sklearn.experimental import enable_iterative_imputer
# from sklearn.impute import IterativeImputer
# from sklearn.ensemble import RandomForestRegressor
# import pandas as pd
# # 载入数据
# titanic = pd.read_csv("titanic.csv")
# titanic = titanic.loc[:, ['Pclass', 'Age', 'SibSp', 'Parch', 'Fare']]
# # 使用随机森林估计器
# imp = IterativeImputer(RandomForestRegressor(), max_iter=10, random_state=0)
# titanic = pd.DataFrame(imp.fit_transform(titanic), columns=titanic.columns)
实例1
# 导入红酒数据集
from sklearn.datasets import load_wine
# 导入MLP神经网络
from sklearn.neural_network import MLPClassifier
# 导入数据集拆分工具
from sklearn.model_selection import train_test_split
# 建立训练集和测试集
wine = load_wine()
X_train, X_test, y_train, y_test = train_test_split(wine.data, wine.target, random_state=62)
# 打印数据集
print(X_train.shape, X_test.shape)
(133, 13) (45, 13)
import warnings
warnings.filterwarnings("ignore")
# 设定MLP神经网络的参数
mlp = MLPClassifier(hidden_layer_sizes=[100,100],max_iter=400,random_state=62)
# 使用MLP拟合数据
mlp.fit(X_train, y_train)
print("未经处理的数据,模型得分为:{:.2f}".format(mlp.score(X_test, y_test)))
未经处理的数据,模型得分为:0.93
设定MLP的隐藏层为2, 每一层100各节点,最大迭代次数400,同时指定random_state,从而保证,重复使用该模型的时候,训练的结果是一致的。
# 使用MinMaxScaler进行数据预处理
scaler = MinMaxScaler()
scaler.fit(X_train)
X_train_pp = scaler.transform(X_train)
X_test_pp = scaler.transform(X_test)
# 再次训练模型
mlp.fit(X_train_pp, y_train)
# 打印模型表现
print("经处理的数据,模型得分为:{:.2f}".format(mlp.score(X_test_pp, y_test)))
经处理的数据,模型得分为:1.00
数据降维-----主成分分析
# 导入红酒数据集
from sklearn.preprocessing import StandardScaler
# 对红酒数据集进行预处理
scaler = StandardScaler()
X = wine.data
y = wine.target
# 处理之后的数据
X_scaled = scaler.fit_transform(X)
print(X_scaled.shape)
(178, 13)
现在的数据集中,样本数量为178,特征数量为13.
# 导入PCA
from sklearn.decomposition import PCA
# 设置主成份数量为6
pca = PCA(n_components=2)
pca.fit(X_scaled)
X_pca = pca.transform(X_scaled)
# 打印主成分提取之后的数据形态
print(X_pca.shape)
(178, 2)
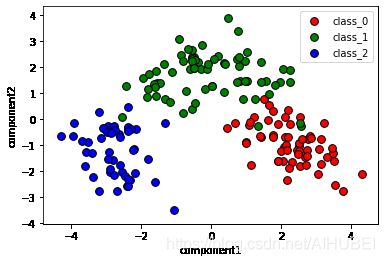
# 把三个分类中的主成分提取出来
X0 = X_pca[wine.target==0]
X1 = X_pca[wine.target==1]
X2 = X_pca[wine.target==2]
# 散点图
plt.scatter(X0[:,0], X0[:,1],c='r', s=60,edgecolor='k')
plt.scatter(X1[:,0], X1[:,1],c='g', s=60,edgecolor='k')
plt.scatter(X2[:,0], X2[:,1],c='b', s=60,edgecolor='k')
# 设置图注
plt.legend(wine.target_names,loc='best')
plt.xlabel('component1')
plt.ylabel('component2')
# 显示
plt.show()
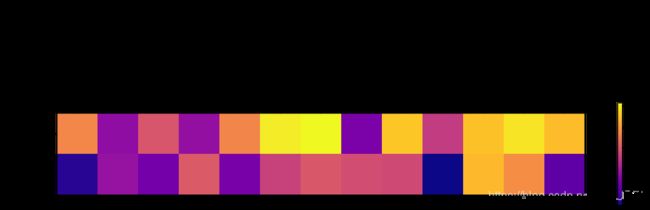
原始特征与PCA主成分之间的关系
# 使用主成分绘制热力图
plt.matshow(pca.components_, cmap='plasma')
# 纵轴为主成分数量
plt.yticks([0,1],["component1","component2"])
plt.colorbar()
# 横轴为原始特征数量
plt.xticks(range(len(wine.feature_names)), wine.feature_names, rotation=60, ha='left')
# 显示图像
plt.show()
特征提取
特征提取,是构造一个新的特征空间,并将原始特征投影在新的空间中得到新的表示。以线性投影为例,令 X ∈ R D \boldsymbol{X}\in \mathcal{R}^D X∈RD为原始特征空间, X ′ ∈ R K \boldsymbol{X}^{\prime}\in \mathcal{R}^K X′∈RK是经过线性投影后得到的在新空间中的特征向量,如下:
X ′ = W X \boldsymbol{X}^{\prime}=\boldsymbol{W}\boldsymbol{X} X′=WX
其中, W ∈ R K x D \boldsymbol{W}\in{\mathcal{R}}^{KxD} W∈RKxD为映射矩阵。
PCA主成分分析用于特征提取
# 导入数据集获取工具
from sklearn.datasets import fetch_lfw_people
# 载入人脸数据集
faces = fetch_lfw_people(min_faces_per_person=20, resize=0.8)
# 将照片打印出来
fig, axes = plt.subplots(3,4,figsize=(12,9),subplot_kw={'xticks':(), 'yticks':()})
for target, image,ax in zip(faces.target, faces.images, axes.ravel()):
ax.imshow(image, cmap=plt.cm.gray)
ax.set_title(faces.target_names[target])
plt.show()
# 导入神经网络
from sklearn.neural_network import MLPClassifier
X_train, X_test, y_train, y_test = train_test_split(faces.data/255, faces.target, random_state=60)
mlp=MLPClassifier(hidden_layer_sizes=[100,100],random_state=62,max_iter=500)
mlp.fit(X_train, y_train)
print("模型识别准确率:{:.2f}".format(mlp.score(X_test, y_test)))
模型识别准确率:0.59
使用白化功能,白化的目的就是:降低冗余性。白化的过程会让样本之间的相关性降低,且所有特征具有相同的方差
# 使用白化功能处理数据
pca = PCA(whiten=True, n_components=0.9, random_state=60).fit(X_train)
X_train_whiten = pca.transform(X_train)
X_test_whiten = pca.transform(X_test)
X_train_whiten.shape
(1740, 101)
# 使用白化之后的数据训练神经网络
mlp.fit(X_train_whiten, y_train)
mlp.score(X_test_whiten, y_test)
0.6327586206896552
K均值聚类
from sklearn.datasets import make_blobs
# 生成分类数为1的数据集
blobs = make_blobs(random_state=1, centers=1)
X_blobs = blobs[0]
plt.scatter(X_blobs[:,0],X_blobs[:,1],c='r',edgecolor='k')
plt.show()
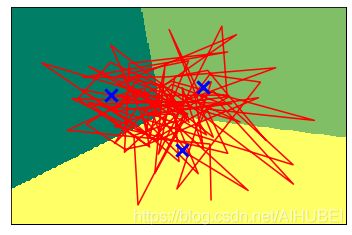
使用K均值进行聚类
# 导入KMeans
from sklearn.cluster import KMeans
# 设定为3类
kmeans = KMeans(n_clusters=3)
# 拟合数据
kmeans.fit(X_blobs)
x_min, x_max = X_blobs[:,0].min()-0.5, X_blobs[:,0].max()+0.5
y_min, y_max = X_blobs[:,1].min()-0.5, X_blobs[:,1].max()+0.5
xx, yy = np.meshgrid(np.arange(x_min, x_max, .02),np.arange(y_min, y_max, .02))
z = kmeans.predict(np.c_[xx.ravel(), yy.ravel()])
z = z.reshape(xx.shape)
plt.figure(1)
plt.clf()
plt.imshow(z, interpolation='nearest', extent=(xx.min(), xx.max(), yy.min(),yy.max()),cmap=plt.cm.summer, aspect='auto', origin='lower')
plt.plot(X_blobs[:, 0], X_blobs[:, 1], 'r', markersize=5)
# 使用蓝色的❌代表据类的中心
centroids = kmeans.cluster_centers_
plt.scatter(centroids[:,0], centroids[:,1],
marker = 'x',s = 150, linewidth=3,
color = 'b', zorder=10)
plt.xlim(x_min,x_max)
plt.ylim(y_min,y_max)
plt.xticks(())
plt.yticks(())
plt.show()
# 打印Kmeans进行聚类的标签
print("K均值的聚类标签:\n{}".format(kmeans.labels_))
K均值的聚类标签:
[2 2 0 1 1 1 2 2 0 1 2 1 2 0 2 1 1 2 0 0 1 0 2 2 2 2 1 2 2 2 0 0 2 2 1 0 1
0 2 0 1 2 0 0 1 1 1 2 0 2 0 2 1 0 1 1 0 1 1 2 1 0 1 2 0 1 0 0 2 1 1 2 1 1
1 2 1 2 2 0 1 0 1 1 0 2 1 2 0 0 1 2 0 0 1 1 2 1 1 2]
凝聚聚类算法
# 导入dendrogram和ward工具
from scipy.cluster.hierarchy import dendrogram, ward
# 使用连线的方式进行可视化
linkage = ward(X_blobs)
dendrogram(linkage)
ax = plt.gca()
plt.xlabel("sample index")
plt.ylabel("Cluster distance")
plt.show()