Science Bulletin:绝对丰度的植物根际微生物群落“扩增-选择”组装模型
Science Bulletin: 基于绝对丰度的植物根际微生物群落“扩增-选择”组装模型
2020年3月7日,Science Bulletin在线发表中科院分子植物科学卓越创新中心/植物生理与生态研究所王二涛课题组及其合作团队完成的题为 “An amplification-selection model for quantified rhizosphere microbiota assembly” 的研究论文,该研究基于微生物绝对丰度提出了植物根际微生物群落 “扩增-选择” 组装的新模型。
根际微生物群落“扩增-选择”组装新模型:左图为基于微生物相对量的根际微生物群落“两步选择”组装模型(two-step selection model),右图为根际微生物群落“扩增-选择”组装新模型(Amplification-selection model)。
基于绝对丰度的植物根际微生物群落“扩增-选择”组装模型
An amplification-selection model for quantified rhizosphere microbiota assembly
Impact Factor:6.28(预计9.61)
DOI:https://doi.org/10.1016/j.scib.2020.03.005
发表日期:2020-03-07
第一作者:Xiaolin Wang(王孝林)1,2
通讯作者:Xuebin Zhang(张学斌)4([email protected]), Nan Yu(于楠)2([email protected]), Ertao Wang(王二涛)1* ( [email protected])
合作作者:Mingxing Wang(王明星)1,3, Xingguang Xie(谢星光)1, Siyi Guo(郭思义)4, Yun Zhou(周云)4
主要单位:
1中国科学院分子植物科学卓越创新中心(Center for Excellence in Molecular Plant Sciences, Institute of Plant Physiology and Ecology, Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences)
2上海师范大学生命科学学院(College of Life Sciences, Shanghai Normal University)
3中国科学院大学(University of Chinese Academy of Sciences,)
4河南大学生命科学学院(Department of Biology, Henan University)
背景介绍
在自然界中,植物根系与大量的微生物互作,这些微生物定殖在根际土中,或附着于根系表面,或定殖于根内,统称为植物根际微生物组。植物根际微生物对于植物的生长发育和环境适应具有重要作用。基于传统的微生物相对丰度(the relative abundance,通过高通量测序16S/18S rRNA基因获得环境样本中各微生物群落的相对组成)数据,研究人员提出了根际微生物群落的两步或多步筛选组装模型(two-step selection model 或者multi-step selection model),该模型认为:微生物依次在根外土(bulk soil)、根际土 (rhizosphere soil) 和根内 (root) 逐步被筛选,形成植物根际特异的微生物群落(图1g)。其中变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)是根际富集的主要菌群,而酸酐菌门(Acidobacteria)是根际排斥的主要菌群(图1g)。
基于微生物相对丰度的研究描述了不同植物物种根际微生物的群落组成及多样性,但忽略了单位质量或体积中不同微生物群落的绝对丰度(the absolute abundance)。另外基于16S/18S rRNA基因的相对丰度研究忽视了不同细菌/真菌的16S/18S rRNA基因的拷贝数差异。因此,相对丰度的研究结果很难与定量的植物生理生态指标进行联系,阻碍了我们对于根际微生物生态功能的理解。
研究方法
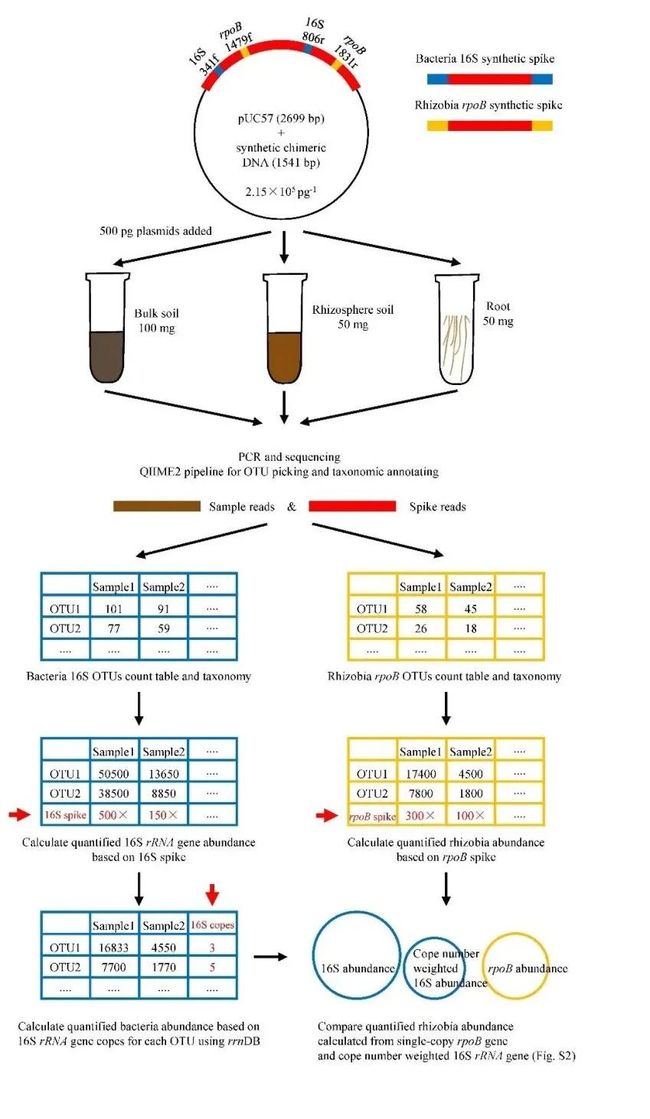
为了克服常规16S rRNA基因高通量测序研究的局限性,我们采用了以下组合方法 (图S1):(1)我们先向根际样本中添加了已知含量的人工合成的spike-in质粒(spike-in质粒包含随机排列的碱基序列,随机序列两端是16S rRNA基因测序的正反向引物对应序列,随机序列长度及碱基GC含量与16S rRNA基因测序片段一致),用来定量检测单位质量根际样本中细菌16S rRNA基因的总量 (quantified 16S abundance);(2)我们进一步通过rrnDB数据库获取了每种细菌可操作分类单元(Operational Taxonomic Units,OTU)的16S rRNA基因平均拷贝数,然后通过加权平均获得了每种细菌OTU的绝对细胞数目(quantified bacterial abundance);(3)我们通过对单拷贝的rpoB基因测序,比较并评估基于16S rRNA基因定量测序计算出的绝对细菌数目的准确性。采用该组合方法,我们定量了蒺藜苜蓿和水稻的根外土、根际土及根内微生物群落的绝对丰度。
图S1. 一种定量并评估根际微生物绝对丰度的组合方法
结果与讨论
通过定量蒺藜苜蓿的根际微生物组,我们发现平均每克根外土、根际土和根系中细菌16S rRNA 基因的含量为1.1 × 109,1.51 × 1010和3.28 × 109,根际土中细菌16S rRNA 基因的含量是根外土的13.7倍。当对16S rRNA 基因的拷贝数进行加权后,我们发现平均每克根外土、根际土和根系中细菌数目为6.91 × 108,6.44 × 109和1.24 × 109,根际土中细菌细胞数目的含量是根外土的9.3倍。表明相对于根外土,细菌的绝对丰度在根际土中是显著升高的。
植物来源的根际养分被认为可以促进化能有机营养细菌在根际的生长,我们推测根际土细菌绝对丰度的显著升高可能归因于部分快速生长细菌的根际扩增。我们进一步分析了主要微生物菌门在根外土和根际中的丰度,发现与根外土相比,主要菌门在根际土中的绝对丰度都显著扩增,包括变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、酸杆菌门(Acidobacteria)、疣微菌门(Verrucomicrobia)、绿弯菌门(Chloroflexi)、厚壁菌门(Firmicutes)和 芽单胞菌门(Gemmatimonadete)等(图1a, b),扩增幅度从2倍到30倍不等,最低的是芽单胞菌门(2.44倍),最高的是α-变形菌门(29.86倍)。
图1:绝对定量苜蓿根际微生物群落揭示植物根际微生物群落“扩增-选择”组装模型
我们进一步在OTU水平分析了细菌类群在根际的扩增情况,发现基于相对丰度根际显著排斥的OTU中,其中63.9%的OTU在根际土中实际情况显著扩增(图1d-f)。另外,我们发现与根外土比,在根际土中绝对丰度降低的OTU的测序reads数都很低,表明这种变化很可能是测序深度不足导致的。因而,我们按绝对丰度的比例对根外土、根际土和根内微生物分别进行抽样,然后计算三组样本中细菌的α-多样性。发现细菌的α-多样性在根际土中最高,其次是根外土,在根内最低 (图S5a)。但是如果按照传统的基于相对丰度的研究方法,不考虑不同样本中微生物的绝对丰度差异,根外土中细菌的α-多样性最高,其次是根际土。基于以上结果,我们认为对于大部分细菌类群来说,其在根际土中都显著扩增(图S5b)。
图S5:忽视不同样本细菌的绝对丰度可能导致α-多样性的不准确估计
对于植物根内细菌群落,我们发现变形菌门、拟杆菌门和厚壁菌门的绝对丰度显著高于根外土。根内微生物主要来自于根际,我们进一步分析了根内微生物群落相对于根际微生物群落的比例,发现放线菌门、γ-变形菌门及Δ-变形菌门从根际土进入根内的比例最大,分别为63%、59%和59%。对于水稻根际细菌群落的分析得出了与苜蓿类似的结论。
基于这些研究结果,我们提出根际微生物群落组装 “扩增-选择”的新模型,该模型认为,与根外土相比,主要菌门在根际土中的绝对丰度都被显著扩增,经根际土扩增的微生物进一步被根筛选,形成特异的根内微生物群落。我们把根外土比作“乡村”或者“沙漠”,把根际土比作“大都市”或者“绿洲”,相对于营养较为贫乏的“乡村”,“大都市”可以为微生物提供更多的工作机会,大部分微生物能在“大都市”找到岗位并繁衍扩增。不同微生物扩增倍数可能由微生物自身扩增速率,微生物与微生物互作,植物与微生物互作等因素决定。根际微生物群落组装的“扩增-选择”新模型,将指导我们定量追踪植物不同生长周期下其根际微生物的绝对丰度的变化,将根际微生物与植物定量性状关联起来,更好利用根际微生物提升农业的可持续发展。
该工作是中科院分子植物科学卓越创新中心/植物生理与生态研究所王二涛课题组与上海师范大学于楠课题组和河南大学张学斌课题组合作完成的,王孝林博士为第一作者。王二涛研究员,于楠副教授和张学斌教授为论文共同通讯作者。该研究受中国科学院和国家自然基金委的资助。
作者简介
王二涛
王二涛,博士,研究员,博士生导师,“国家杰出青年基金”获得者。2003年于河南大学获学士学位;2008年于中国科学院研究生院获博士学位;2008-2012年在英国John Innes Centre从事博士后研究;2013年至今,任中国科学院上海植物生理生态研究所研究员。主要从事豆科植物-根瘤共生固氮,植物-丛枝菌根真菌共生方面的研究。建立植物-丛枝菌根真菌共生营养交换的新理论模型;克隆菌根因子受体复合,并阐述了菌根因子信号转导途径等。研究成果以通讯作者发表在Science, Nature系列, Molecular Plant, The Plant Cell和Cell Research等国际主流学术期刊上,对植物-微生物共生领域有重要影响。
于楠
于楠,博士,副教授。2003于上海交通大学获学士学位;2010年于中国科学院研究生院获博士学位;2010-2013年在英国John Innes Centre从事博士后研究;2015至今在上海师范大学担任副教授。现主要从事植物根部干细胞层命运决定分子机制及豆科植物根瘤发育分子机理的研究。近年主要研究成果以第一作者或通讯作者发表在Plant Cell, Nucleic Acids Research, Cell Research, Molecular Plant等国际知名期刊。
张学斌
张学斌,博士,教授,博士生导师。2005于山西农业大学获学士学位;2008年于英国诺丁汉大学获理学博士学位;2008-2011年在英国Rothamsted Research从事博士后工作;2011-2018就职于美国Brookhaven National Laboratory。现任河南大学生命科学学院教授。主要从事植物苯丙素类化合物合成途径调控和F-box基因家族功能解析方面的研究。在Nature Communications, Plant Biotechnology, The Plant Cell,Plant Physiology,Molecular plant, Biochemical Journal,FEBS letter等国际知名学术期刊发表研究论文。
王孝林
王孝林,博士。2013年于江南大学生物工程学院获学士学位;2018年于中国农业大学生物学院获博士学位;2018年至今在中科院分子植物科学卓越创新中心从事博士后工作。目前主要研究方向为微生物生态和植物微生物组。近年在Nature、The ISME Journal、Soil Biology and Biochemistry、Science Bulletin等杂志发表过论文。
Reference
原文链接:https://www.sciencedirect.com/science/article/pii/S2095927320301341
Xiaolin Wang, Mingxing Wang, Xingguang Xie, Siyi Guo, Yun Zhou, Xuebin Zhang, Nan Yu & Ertao Wang. An amplification-selection model for quantified rhizosphere microbiota assembly. Science Bulletin. 2020. doi:https://doi.org/10.1016/j.scib.2020.03.005
杂志简介
官网:http://engine.scichina.com/publisher/scp/journal/SB
海外发行官方:https://www.journals.elsevier.com/science-bulletin
Science Bulletin是由中国科学院和国家自然科学基金委员会共同主办、《中国科学》杂志社出版的自然科学综合性学术刊物, 致力于快速报道自然科学各学科基础理论和应用研究的最新研究动态、消息、进展, 点评研究动态和学科发展趋势。要求文章短小精悍, 可读性强, 能在比较宽泛的学术领域产生影响。
2018年影响因子:6.277
预计2019年影响因子 > 9.61
历史:Science Bulletin原名CHINESE SCIENCE BULLETIN,历史悠久,自1997即获得首个影响因子0.3至2016年止影响因子1.649,年发稿量在600篇左右。
当今:2015年杂志起更名为Science Bulletin,对稿件质量有了更高的要求,年发文量下降至200篇以内,2016年获得首个影响因子4.902,至2018年跨越式发展,影响因子达6.277。在中国科技期刊国际影响力提升计划、中国科研全面发展和更严格的文章质量控制的背景下,该杂志今年预计影响因子可达9.61,甚至突破10,值得同行关注。
相关文章
Plant Com:中科院遗传发育所白洋组开发定量检测宿主微生物组的HA-QAP技术(王二涛点评)
植物学报:根际微生物促进水稻氮利用的机制(王孝林, 王二涛)
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]() 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读