如何画热力图
热图绘制函数heatmap.2()的参数解析
在进行数据分析的时候,你一定见过或用过热图(heatmap)。它适合二维数据表的可视化,数值大小可以用不同颜色梯度进行展示。事实上,经过高通量测序生成的物种丰度表以及差异分析得到的差异物种丰度表,只有进一步生成直观图,才能更好地进行结果展示,热图就是我们常用的图片类型之一。然而,你会不会在很多时候觉得绘制出来的热图让你很不满意,想修改却无从下手?给大家介绍一个用于绘制热图的R函数——来自gplots包的heatmap.2(),看看如何利用其修改参数让热图更称你的心意。
一、基础绘图
首先,加载程序包,输入原始数据。
> library(gplots)
> data=read.table("for_plot_filter.txt",header=TRUE,row.names=1,sep="t",check.names=F,quote="")
输入的数据格式是这个样子的:
然后,R画图。
> pdf("heatmap.pdf",12,16)
> heatmap.2(as.matrix(data))
> dev.off()
如果不对参数进行任何修改,仅用默认参数,得到的图形是这个样子的:
得到这么原始的图片,我想你的内心一定是崩溃的。那么,接下来就来看看如何通过调整不同参数,让我们的热图改头换面吧。
二、调整参数
2.1 修改整体布局
> pdf("heatmap.pdf",12,16)
> par(oma=c(0,0,0,20)) #调整边界
> lmat = rbind(c(4,4),c(0,3),c(2,1),c(0,5)) #安排热图每个部分的位置
> lhei = c(0.5,0.8,4,0.5) #每个部分的高度比例
> lwid = c(1,3) #页面宽度方向的比例
> cexRow=2 #行名,即物种名的字体大小
> cexCol=2 #列名,即样品名的字体大小
> margin=c(2,2) #边界宽度
> heatmap.2(as.matrix(data),margins=margin,lmat = lmat,lhei=lhei,lwid=lwid,cexRow=cexRow,cexCol=cexCol)
> dev.off()
这样一来,各部分的安置包括字体的大小都有了很大的改观:
2.2 调整色块及颜色
> note=matrix(0,nrow(data),ncol(data))
> for(i in 1:nrow(data)){
+ for (j in 1:ncol(data)){
+ if(data[i,j]>0.1) note[i,j]="*" else if(data[i,j]>0.01) note[i,j]="+" else note[i,j]=""
+ }
+ } #对样品中相对丰度大于0.01和0.1的物种丰度进行标记,也可以不要
> pdf("heatmap.pdf",12,16)
> par(oma=c(0,0,0,20))
> library(RColorBrewer) #加载调色板
> col = colorRampPalette(c("lightblue", "yellow", "orange", "red"),bias=3)(3000) #设置颜色梯度
> heatmap.2(as.matrix(data),margins=margin,lmat = lmat,lhei=lhei,lwid=lwid,cexRow=cexRow,cexCol=cexCol,
col=col,scale='none',trace="none",
cellnote=note, notecol='black',notecex=1.0, #色块标记设置及标记的颜色和大小
colsep=c(1:ncol(data)),rowsep=c(1:nrow(data)),sepcolor="black",sepwidth=c(0.01, 0.01), #设置色块之间的间隔及颜色
key.title=NA, keysize=0.06,key.xlab="relative abundance") #图片最上方键的设置,包括大小及名称
> dev.off()
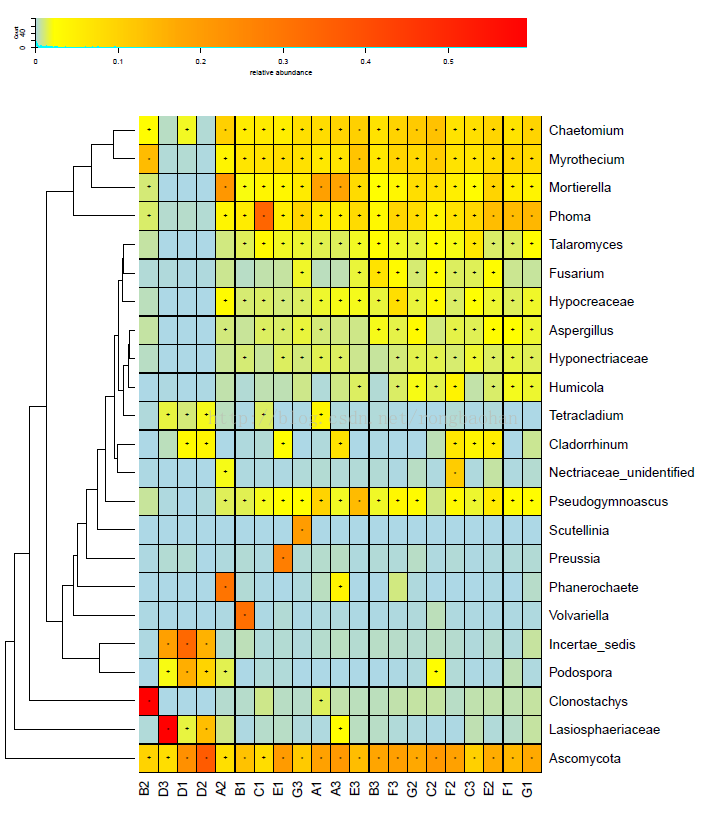
调整色块和颜色后,热图看着清爽多了:
2.3 个性化取消或添加某部分
在上面这张热图中,有行和列两个方向的聚类,即物种和样品的聚类,如果你只想要其中一个方向的聚类树,或者两个都不想要,通过调整dendrogram的值即可。为了更美观,各部分高度和宽度的比例值也可以进行相应修改。
> pdf("heatmap.pdf",12,16)
> par(oma=c(0,0,10,20))
> lmat = rbind(c(4,4),c(0,3),c(2,1),c(0,5))
> lhei = c(0.5,0.2,4,0.5) #修改高度比例
> lwid = c(1,3)
> heatmap.2(as.matrix(data),margins=c(2,2),lmat = lmat,lhei=lhei,lwid=lwid,
+ cexRow=cexRow,cexCol=cexCol,col=col,
+ scale='none',trace="none",
+ cellnote=note, notecol='black',notecex=1.0,
+ colsep=c(1:ncol(data)),rowsep=c(1:nrow(data)),sepcolor="black",sepwidth=c(0.01, 0.01),
+ key.title=NA, keysize=0.06,
+ key.xlab="relative abundance",
+ dendrogram = "row") #只保留行上的聚类树 dendrogram=c("both","row","column","none")
> dev.off()
文章出处:http://www.realbio.cn/index.php?c=msg&id=1151&