RNA-seq分析流程
提前申明:该贴参照于https://www.jianshu.com/p/e8cd62ba14fe
标题1. 用conda安装RNA-seq所需要
#启动conda自设环境
conda activate python3
或者用
source activate python3
#安装所需软件(conda可以同时安装多个软件,但是建议初学者还是选择逐一安装,避免出现错误)
conda install bowtie14 samtools fastqc
conda install hisat2 samtools sratoolkit
conda install trimmomatics htseq(要求在python2的环境下运行,所以安装时记得退出python3,激活python2环境安装)
#查看当前环境安装软件是否可用
samtools
which samtools
#退出当前环境
source deactivate#退出python2
source deactivate#退出base
标题2. 获取原始数据
原始数据的获得最直接的是测序公司测序得到,但是有时候需要我们从网上挖掘已有研究的数据,这就涉及到原始数据的下载问题。从NCBI下载的原始数据一般为SRA文件,需要使用sratoolkit中的fastq-dump和sam-dump命令将sra格式转为fastq格式。
Aspera connect是IBM的商业化高速文件下载软件,但可以免费下载NCBI和EBI的数据。速度可达200-500Mbps,几乎所有站点都超过10Mbps,如果Aspera connect不能下载,则推荐sratoolkit的prefetch功能;尽量不要使用wget或curl命令来下载。
fastq-dump /path/to/xxx.sra
默认情况下fastq-dump不对reads进行拆分, 对于单端测序选择默认参数即可,但是对于双端测序而言,默认就会把原本的两条reads合并成一个,后续分析必然会出错:
–split-spot: 将双端测序分为两份,但是都放在同一个文件中
–split-files: 将双端测序分为两份,放在不同的文件,但是对于一方有而一方没有的reads直接丢弃
–split-3 : 将双端测序分为两份,放在不同的文件,但是对于一方有而一方没有的reads会单独放在一个文件夹里。如果你不知道到底是单端还是双端的SRA文件,一律用–split-3.
另外,–gzip就能输出gz格式, 能够节省空间的同时也不会给后续比对软件造成压力, 比对软件都支持,就是时间要多一点。
标题3. 用fastqc进行质量控制
用法:
fastqc [-o output dir] [–(no)extract] [-f fastq|bam|sam] [-c contaminant file] seqfile1 … seqfileN
参数:
-o 输出目录,需自己创建目录
–(no)extract 是否解压输出文件,默认是自动解压缩zip文件。加上–noextract不解压文件。
-f 指定输入文件的类型,支持fastq|bam|sam三种格式的文件,默认自动识别。
-t 同时处理的文件数目。
-c 是contaminant 文件,会从中搜索overpresent 序列。
#将所有的数据进行质控,得到zip的压缩文件和html文件
fastqc -o . *.fastq.gz
注意:-o后面有空格,表示输出到当前文件夹,之后的.后也有空格
标题 4. 质控结果批量查看工具:multiQC
如果文件不多可以一个个查看fastqc质控结果,但如果文件很多,可以进行合并查看:
#conda安装
conda install -c bioconda multiqc # install multiqc
multiqc . #Run
安装好后,进入你要分析的测序文件所在的文件夹,直接输入multiqc加要扫描的目录即可运行,如果要扫描当前文件夹,直接输入"multiqc ."即可
multiqc .
multiqc /data/mydir/
multiqc /data/*fastqc.zip
multiqc /data/sanple_1*
使用“–ignore”参数忽略某些文件
multiqc . --ingore *_R2*
multiqc . --ignore run_two/
multiqc . --ignore */run_three/*/fastqc/*.zip
使用文本指定要分析的文件的路径
multiqc --file-list_my_file_list.txt
分析结果默认命名为“multiqc_report.html”,相关的以tab风格的data file保存在“multiqc_data”文件夹下。可以用“-n”参数改变结果文件的名字,用“-o”改变输出文件的位置,添加参数“-f”,输出结果时会自动覆盖同名文件,添加参数“-t”或者“–template”可以选择不同风格的报告模板,具体内容请查看帮助文档“multiqc --help”。同时,MultiQC也支持自行创作结果文件的模板。
除了直接输出html文件外,Multiqc还可以直接保存图片文件。用以下参数进行保存:
multiqc -p/--export
默认设置下,图片会保存在“multiqc_plots”文件夹中,以.png/.svg或者pdf格式保存。 同时,也可以直接在html文件中使用“toolbox”中的Export 保存图片。
标题5. 下载参考基因组及基因注释
测序得到的是几百bp的短read, 相当于把拼图打散了给你。如果没有参考基因组,从头(de novo)组装等于是重走人类基因组计划的老路,也就是打散了拼图,却不告诉你原来是什么样子,那么任务将会及其艰巨。还好许多动物的基因组已经组装好了,我们只需要把我们测得序列回贴(mapping)回去即可。因此第一步就是要去UCSC(http://genome.ucsc.edu/index.html)或NCBI/ENSEML下载你需要的参考基因组。
然而参考基因组是一部无字天书,要想解读书中的内容,需要额外的注释信息协助。因此第二步,就是去gencode数据库(http://www.gencodegenes.org/)下载基因组注释文件
标题6. 序列比对:Hisat2
RNA-Seq数据分析分为很多种,比如说找差异表达基因或寻找新的可变剪切。如果找差异表达基因单纯只需要确定不同的read计数就行的话,我们可以用bowtie, bwa这类比对工具,或者是salmon这类align-free工具,并且后者的速度更快。
但是如果你需要找到新的isoform,或者RNA的可变剪切,看看外显子使用差异的话,你就需要TopHat, HISAT2或者是STAR这类工具用于找到剪切位点。因为RNA-Seq不同于DNA-Seq,DNA在转录成mRNA的时候会把内含子部分去掉。所以mRNA反转的cDNA如果比对不到参考序列,会被分开,重新比对一次,判断中间是否有内含子。
a. 建立索引
有时候没有现成的index,我们就需要自己用HISAT2重新构建索引;包括外显子、剪切位点及SNP索引的建立:
# 其实hisat2-buld在运行的时候也会自己寻找exons和splice_sites,但是先做的目的是为了提高运行效率
extract_exons.py gencode.v26lift37.annotation.sorted.gtf > hg19.exons.gtf &
extract_splice_sites.py gencode.v26lift37.annotation.gtf > hg19.splice_sites.gtf &
# 建立index, 必须选项是基因组所在文件路径和输出的前缀
hisat2-build --ss hg19.splice_sites.gtf --exon hg19.exons.gtf genome/hg19/hg19.fa hg19
b.开始比对:用hisat2,得到SAM文件(参考帮助文件)
c.SAM文件转换为BAM文件
sam文件转换为bam文件,并对bam文件进行sorted(其中有两种排序方式N和P),最后建立索引:
第一种方式
# 首先将比对后的sam文件转换成bam文件
# 利用的是samtools的view选项,参数-S 输入sam文件;参数-b 指定输出的文件为bam;最后重定向写入bam文件
$ cd mnt/f/rna_seq/aligned
$ for ((i=56;i<=62;i++));do samtools view -S SRR35899${i}.sam -b > SRR35899${i}.bam;done
# 将所有的bam文件按默认的染色体位置进行排序
$ for ((i=56;i<=62;i++));do samtools sort SRR35899${i}.bam -o SRR35899${i}_sorted.bam;done
# 将所有的排序文件建立索引,索引文件.bai后缀
$ for ((i=56;i<=62;i++));do samtools index SRR35899${i}_sorted.bam;done
第二种方式
for i in `seq 56 62`
do
samtools view -S SRR35899${i}.sam -b > SRR35899${i}.bam
samtools sort SRR35899${i}.bam -o SRR35899${i}_sorted.bam
samtools index SRR35899${i}_sorted.bam
done
标题7. 对BAM进行质控QC
Picard https://broadinstitute.github.io/picard/ RSeQC
http://rseqc.sourceforge.net/ Qualimap
http://qualimap.bioinfo.cipf.es/
标题8. 加载IGV,可视化比对结果
载入参考序列,注释和BAM文件
标题9. HTseq-count进行reads计数
定量分为三个水平:
基因水平(gene-level)
转录本水平(transcript-level)
外显子使用水平(exon-usage-level)。
在基因水平上,常用的软件为HTSeq-count,featureCounts,BEDTools, Qualimap, Rsubread, GenomicRanges等。以常用的HTSeq-count为例,这些工具要解决的问题就是根据read和基因位置的overlap判断这个read到底是谁家的孩子。值得注意的是不同工具对multimapping reads处理方式也是不同的,例如HTSeq-count就直接当它们不存在。而Qualimpa则是一人一份,平均分配。
对每个基因计数之后得到的count matrix再后续的分析中,要注意标准化的问题。如果你要比较同一个样本(within-sample)不同基因之间的表达情况,你就需要考虑到转录本长度,因为转录本越长,那么检测的片段也会更多,直接比较等于让小孩和大人进行赛跑。如果你是比较不同样本(across sample)同一个基因的表达情况,虽然不必在意转录本长度,但是你要考虑到测序深度(sequence depth),毕竟测序深度越高,检测到的概率越大。除了这两个因素外,你还需要考虑GC%所导致的偏差,以及测序仪器的系统偏差。目前对read count标准化的算法有RPKM(SE), FPKM(PE),TPM, TMM等,不同算法之间的差异与换算方法已经有文章进行整理和吐槽了。但是,有一些下游分析的软件会要求是输入的count matrix是原始数据,未经标准化,比如说DESeq2,这个时候你需要注意你上一步所用软件会不会进行标准化。
在转录本水平上,一般常用工具为Cufflinks和它的继任者StringTie, eXpress。这些软件要处理的难题就时转录本亚型(isoforms)之间通常是有重叠的,当二代测序读长低于转录本长度时,如何进行区分?这些工具大多采用的都是expectation maximization(EM)。好在我们有三代测序。上述软件都是alignment-based,目前许多alignment-free软件,如kallisto, silfish, salmon,能够省去比对这一步,直接得到read count,在运行效率上更高。不过最近一篇文献[1]指出这类方法在估计丰度时存在样本特异性和读长偏差。
在外显子使用水平上,其实和基因水平的统计类似。但是值得注意的是为了更好的计数,我们需要提供无重叠的外显子区域的gtf文件[2]。用于分析差异外显子使用的DEXSeq提供了一个Python脚本(dexseq_prepare_annotation.py)执行这个任务。
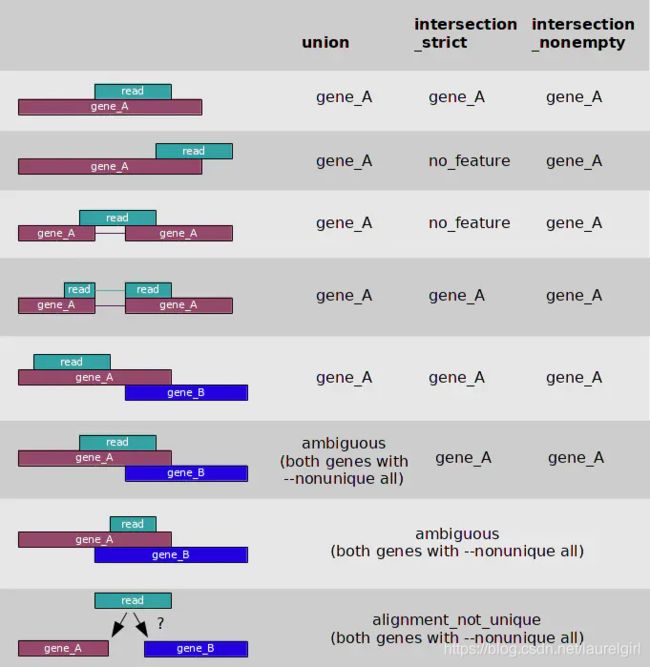
如何判断一个 reads 属于某个基因, htseq-count 提供了 union, intersection_strict,intersection_nonempty 3 种模型,如图(大多数情况下作者推荐用 union模型),如果这三种模型还是不和你心意,可以通过htseq-count 自定义模型,方法详见 A tour through HTSeq 。
a.数据准备
输入为sam格式的文件,如果是paired-end(双端测序),必须对SAM文件按照reads名称排序(sort by name,not position)。对read name或 位置 进行排序皆可,通过 -r 可以指定您的数据是以什么方式排序的: 可以是 name 或 pos , 默认是name.命令为
samtools sort -n accepted_hits_unique.bam -o accepted_hits_unique_name_sorted.bam # -n 按read name 排序 ,如果不指定则按染色体位置(pos)排序
具体来说:先用samtools先对bam文件排序,再转换为sam(这一步可以不要,不必进行转换)。
b.reads计数,得到表达矩阵
数据准备已经完成,接下来要使用htseq-count对数据进行reads 计数。
htseq支持SAM和BAM文件,所以就没必要把name-sorted BAM文件转成SAM文件,那样会用去很多时间和空间。这个通过-f bam解决。另外双端测序数据必须进行排序,看-r选项,即支持染色体位置排序(pos),又支持reads name排序,但name排序会更好。前面已经按name排序完成
for ((i=59;i<=62;i++));do htseq-count -r name -f bam /mnt/f/rna_seq/aligned/SRR35899${i}_nsorted.bam /mnt/f/rna_seq/data/reference/annotation/mm10/gencode.vM10.chr_patch_hapl_scaff.annotation.gtf > /mnt/f/rna_seq/data/matrix/SRR35899${i}.count;done
或这样处理
for i in `seq 59 62`
do
htseq-count -r name -f bam /mnt/f/rna_seq/aligned/SRR35899${i}_nsorted.bam /mnt/f/rna_seq/data/reference/annotation/mm10/gencode.vM10.chr_patch_hapl_scaff.annotation.gtf > /mnt/f/rna_seq/data/matrix/SRR35899${i}.count 2> /mnt/f/rna_seq/data/matrix/SRR35899${i}.log
done
c.对结果进行统计
$ wc -l *.count
48825 SRR3589959.count
48825 SRR3589960.count
48825 SRR3589961.count
48825 SRR3589962.count
195300 total
看下每个文件的格式,查看前4行,第一列ensembl_gene_id,第二列read_count计数
$ head -n 4 *.count
==> SRR3589959.count <==
ENSMUSG00000000001.4 1648
ENSMUSG00000000003.15 0
ENSMUSG00000000028.14 835
ENSMUSG00000000031.15 65
==> SRR3589960.count <==
ENSMUSG00000000001.4 2941
ENSMUSG00000000003.15 0
ENSMUSG00000000028.14 1366
ENSMUSG00000000031.15 52
==> SRR3589961.count <==
ENSMUSG00000000001.4 2306
ENSMUSG00000000003.15 0
ENSMUSG00000000028.14 950
ENSMUSG00000000031.15 83
==> SRR3589962.count <==
ENSMUSG00000000001.4 2780
ENSMUSG00000000003.15 0
ENSMUSG00000000028.14 1051
ENSMUSG00000000031.15 53
$ tail -n 4 *.count
==> SRR3589959.count <==
__ambiguous 295498
__too_low_aQual 1107674
__not_aligned 1025857
__alignment_not_unique 11278964
==> SRR3589960.count <==
__ambiguous 498973
__too_low_aQual 1799176
__not_aligned 1675660
__alignment_not_unique 18440618
==> SRR3589961.count <==
__ambiguous 425708
__too_low_aQual 1303141
__not_aligned 1067038
__alignment_not_unique 14080481
==> SRR3589962.count <==
__ambiguous 381237
__too_low_aQual 1542845
__not_aligned 1529309
__alignment_not_unique 14495860
- 合并表达矩阵并进行注释(R中进行)
(1) 载入数据,添加列名
再看下原始数据,可见59和61和control,60和62是实验
> options(stringsAsFactors = FALSE)
> control1<-read.table("SRR3589959.count",sep = "\t",col.names = c("gene_id","control1"))
> head(control1)
gene_id control1
1 ENSMUSG00000000001.4 1648
2 ENSMUSG00000000003.15 0
3 ENSMUSG00000000028.14 835
4 ENSMUSG00000000031.15 65
5 ENSMUSG00000000037.16 70
6 ENSMUSG00000000049.11 0
> control2<-read.table("SRR3589961.count",sep = "\t",col.names = c("gene_id","control2"))
> treat1<-read.table("SRR3589960.count",sep = "\t",col.names = c("gene_id","treat1"))
> treat2<-read.table("SRR3589962.count",sep = "\t",col.names = c("gene_id","treat2"))
(2)merge进行数据整合
gencode的注释文件中的gene_id(如ENSMUSG00000105298.13_3)在EBI是不能搜索到的,所以用gsub功能只保留ENSMUSG00000105298这部分,处理之前先看一下,也就是最后5行是我们不需要的,可以删除
> tail(control1)
gene_id control1
48820 ENSMUSG00000110424.1 26
48821 __no_feature 12642161
48822 __ambiguous 295498
48823 __too_low_aQual 1107674
48824 __not_aligned 1025857
48825 __alignment_not_unique 11278964
进行整合
raw_count <- merge(merge(control1, control2, by=“gene_id”), merge(treat1, treat2, by=“gene_id”))
head(raw_count)
gene_id control1 control2 treat1 treat2
1 __alignment_not_unique 11278964 14080481 18440618 14495860
2 __ambiguous 295498 425708 498973 381237
3 __no_feature 12642161 15042888 22357626 18675857
4 __not_aligned 1025857 1067038 1675660 1529309
5 __too_low_aQual 1107674 1303141 1799176 1542845
6 ENSMUSG00000000001.4 1648 2306 2941 2780
tail(raw_count)
gene_id control1 control2 treat1 treat2
48820 ENSMUSG00000110419.1 46 39 68 58
48821 ENSMUSG00000110420.1 0 0 0 0
48822 ENSMUSG00000110421.1 0 0 0 1
48823 ENSMUSG00000110422.1 1 0 1 2
48824 ENSMUSG00000110423.1 0 0 0 0
48825 ENSMUSG00000110424.1 26 20 48 24
删除前5行
#这里要注意,我读入之后顺序变了,删除的时候看下删除的是哪些行
raw_count_filt <- raw_count[-1:-5,]
gene_id control1 control2 treat1 treat2
6 ENSMUSG00000000001.4 1648 2306 2941 2780
7 ENSMUSG00000000003.15 0 0 0 0
8 ENSMUSG00000000028.14 835 950 1366 1051
9 ENSMUSG00000000031.15 65 83 52 53
10 ENSMUSG00000000037.16 70 53 94 66
11 ENSMUSG00000000049.11 0 3 4 5
因为我们无法在EBI数据库上直接搜索找到ENSMUSG00000024045.5这样的基因,只能是ENSMUSG00000024045的整数,没有小数点,所以需要进一步替换为整数的形式。
# 第一步将匹配到的.以及后面的数字连续匹配并替换为空,并赋值给ENSEMBL
>ENSEMBL <- gsub("\\.\\d*", "", raw_count_filt$gene_id)
# 将ENSEMBL重新添加到raw_count_filt1矩阵
>row.names(raw_count_filt) <- ENSEMBL
>head(raw_count_filt)
gene_id control1 control2 treat1 treat2
ENSMUSG00000000001 ENSMUSG00000000001.4 1648 2306 2941 2780
ENSMUSG00000000003 ENSMUSG00000000003.15 0 0 0 0
ENSMUSG00000000028 ENSMUSG00000000028.14 835 950 1366 1051
ENSMUSG00000000031 ENSMUSG00000000031.15 65 83 52 53
ENSMUSG00000000037 ENSMUSG00000000037.16 70 53 94 66
ENSMUSG00000000049 ENSMUSG00000000049.11 0 3 4 5
对获得的差异基因进行注释
第二:用bioMart对ensembl_id转换成gene_symbol
> library('biomaRt')
> library("curl")
Warning message:
package ‘curl’ was built under R version 3.4.4
> mart <- useDataset("mmusculus_gene_ensembl", useMart("ensembl"))
> my_ensembl_gene_id<-row.names(raw_count_filt)
> #Sys.setenv(https_proxy="http://proxy:8000")
> options(timeout = 4000000)
> my_ensembl_gene_id<-row.names(raw_count_filt)
> mms_symbols<- getBM(attributes=c('ensembl_gene_id','hgnc_symbol',"chromosome_name", "start_position","end_position", "band"),
+ filters = 'ensembl_gene_id', values = my_ensembl_gene_id, mart = mart)
> readcount<-read.csv(file="raw_count_filt.csv",header = TRUE)
> head(readcount)
ensembl_id gene_id gene_symbol control1 control2 treat1 treat2
1 ENSMUSG00000000001 ENSMUSG00000000001.4 Gnai3 1648 2306 2941 2780
2 ENSMUSG00000000003 ENSMUSG00000000003.15 Pbsn 0 0 0 0
3 ENSMUSG00000000028 ENSMUSG00000000028.14 Cdc45 835 950 1366 1051
4 ENSMUSG00000000031 ENSMUSG00000000031.15 H19 65 83 52 53
5 ENSMUSG00000000037 ENSMUSG00000000037.16 Scml2 70 53 94 66
6 ENSMUSG00000000049 ENSMUSG00000000049.11 Apoh 0 3 4 5
#查看Akap8(别名AKAP95)表达情况
> Akap8 <- readcount[readcount$gene_symbol=="Akap8",]
> Akap8
ensembl_id gene_id gene_symbol control1 control2 treat1 treat2
4265 ENSMUSG00000024045 ENSMUSG00000024045.5 Akap8 936 1368 3386 4116
#treat中显著升高