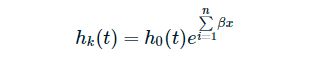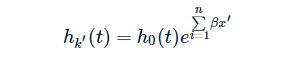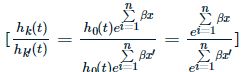翻译自:http://www.sthda.com/english/wiki/cox-proportional-hazards-model
Cox比例风险模型(考克斯,1972年)是常用的统计在医学研究调查的患者和一个或多个预测变量的存活时间之间的关联回归模型。
在上一章 生存分析基础 中,我们描述了生存分析的基本概念以及生存数据的分析和汇总方法,包括:
- 风险和生存函数的定义,
- 不同患者群体的Kaplan-Meier生存曲线的构建
- 对数秩检验(Log-Rank test),用于比较两个或多个生存曲线
上述方法-Kaplan-Meier曲线和logrank检验-是单变量分析的示例。他们根据调查中的一个因素描述了生存情况,但忽略了其他因素的影响。
此外,仅当预测变量为分类变量时(例如:治疗A与治疗B;男性与女性),Kaplan-Meier曲线和对数秩检验才有用。对于定量预测指标(例如基因表达,体重或年龄),它们并不容易工作。
一种替代方法是Cox比例风险回归分析,它既适用于定量预测变量也适用于类别变量。此外,Cox回归模型扩展了生存分析方法,可以同时评估几种风险因素对生存时间的影响。
在本文中,我们将描述Cox回归模型并提供使用R软件的实际示例。
内容
多元统计建模的需求
在临床研究中,有许多情况,其中几个已知量(称为 协变量covariates)可能会影响患者的预后。
例如,假设比较了两组患者:有和没有特定基因型的患者。如果其中一组还包含较年长的个体,则生存率的任何差异都可能归因于基因型或年龄,或两者都有。因此,在调查与任何一个因素相关的生存率时,通常需要针对其他因素的影响进行调整。
统计模型是一种常用工具,可以同时分析多个因素的生存率。此外,统计模型还提供了每个因素的影响大小。
考克斯比例风险模型是用于对生存分析数据进行建模的最重要方法之一。下一节介绍Cox回归模型的基础。
Cox比例风险模型的基础
该模型的目的是同时评估几个因素对生存的影响。换句话说,它允许我们检查特定因素如何影响特定时间点特定事件(例如,感染,死亡)的发生率。该比率通常称为风险比率。预测变量(或因子)在生存分析文献中通常称为协变量 covariates。
Cox模型由 h(t) 表示的风险函数表示。简而言之,危险函数可以解释为在时间t死亡的风险。可以估计如下:
其中:
- t 表示生存时间
- h(t) 是由一组p协变量(x1,x2,…,xp)确定的风险函数
- 系数(b1,b2,…,bp)衡量协变量的影响(即效应大小)(可以理解为风险权重)
- 术语 h0 称为基线危险。如果所有的系数等于零(既exp(0)等于1),则对应于风险的值。h(t)中的“t”提醒我们,危险可能随时间而变化。
Cox模型可以被写为变量x(i)的危险对数的多元线性回归,而基线危险是随时间变化的“截距”项。
系数 bi 称为危险比率(HR,hazard ratio)。bi 值大于零,或相当于风险比率大于1,表明随着第 i 个协变量值的增加,事件风险增加,因此生存时间缩短。
换句话说,风险比大于1表示协变量与事件概率正相关,因此与存活时间负相关。
总之,
HR=1:无影响
HR<1:危害降低
HR>1:危险增加
在癌症研究中:
- 风险比 > 1(即:b > 0)的协变量称为不良预后因素
- 风险比 < 1(即:b < 0)的协变量被称为良好的预后因素
Cox模型的关键假设是观察组(或患者)的危险曲线应成比例,并且不能交叉。
假设两个x值不同的患者k和k'。相应的风险函数可以简单地写成如下:
-
患者k的风险函数:
-
患者k'的风险函数:
-
这两名患者的危险比 与时间t无关。
因此,Cox 模型是一个比例风险模型:任何一组事件的风险都是其他任何一组事件风险的常数倍。这一假设意味着,如上所述,各组的危险曲线应成比例,不能交叉。
换言之,如果一个人在某个初始时间点的死亡风险是另一个人的两倍,那么在以后的任何时候,死亡风险仍然是另一个人的两倍。
这种比例风险的假设应该得到检验。我们将在本系列的下一篇文章中讨论评估比例性的方法:Cox模型假设。
在R中计算Cox模型
安装并加载所需的R包
我们将使用两个R包:
survival 用于计算存活分析
survminer 用于可视化生存分析结果
安装软件包
install.packages(c("survival", "survminer"))
- 加载包
library("survival")
library("survminer")
用于计算Cox模型的R函数:coxph()
函数coxph()[在survival包中]可用于计算R中的Cox比例风险回归模型。
简化格式如下:
coxph(formula, data, method)
- formula:是以生存对象为响应变量的线性模型。使用函数Surv()创建生存对象,如下所示:Surv(time,event)。
- data:包含变量的数据框
- method:用于指定如何处理
tie。默认值为“efron”。其他选项有“breslow”和“exact”。默认的“efron”通常优先于曾经流行的“breslow”方法。“exact”方法的计算量要大得多。
示例数据集
我们将在生存R数据包中使用肺癌数据。
data("lung")
head(lung)
inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
1 3 306 2 74 1 1 90 100 1175 NA
2 3 455 2 68 1 0 90 90 1225 15
3 3 1010 1 56 1 0 90 90 NA 15
4 5 210 2 57 1 1 90 60 1150 11
5 1 883 2 60 1 0 100 90 NA 0
6 12 1022 1 74 1 1 50 80 513 0
- 机构:机构代码
- 时间:以天为单位的生存时间
- 状态:审查状态1 =审查,2 =失效
- 年龄:岁
- 性别:男= 1女= 2
- ph.ecog:ECOG成绩得分(0 =好5 =死)
- ph.karno:医师对Karnofsky绩效评分(不良= 0-良好= 100)
- pat.karno:Karnofsky表现评分,按患者评分
- meal.cal:用餐时消耗的卡路里
- wt.loss:最近六个月的体重减轻
计算考克斯模型
我们将使用以下协变量来拟合Cox回归:年龄,性别,ph.ecog和wt.loss。
我们首先为所有这些变量计算单变量Cox分析。然后我们将使用两个变量来拟合多元Cox分析,以描述这些因素如何共同影响生存。
单变量Cox回归
单变量Cox分析的计算公式如下:
res.cox <- coxph(Surv(time, status) ~ sex, data = lung)
res.cox
Call:
coxph(formula = Surv(time, status) ~ sex, data = lung)
coef exp(coef) se(coef) z p
sex -0.531 0.588 0.167 -3.18 0.0015
Likelihood ratio test=10.6 on 1 df, p=0.00111
n= 228, number of events= 165
Cox模型的功能摘要()产生更完整的报告:
summary(res.cox)
Call:
coxph(formula = Surv(time, status) ~ sex, data = lung)
n= 228, number of events= 165
coef exp(coef) se(coef) z Pr(>|z|)
sex -0.5310 0.5880 0.1672 -3.176 0.00149 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
exp(coef) exp(-coef) lower .95 upper .95
sex 0.588 1.701 0.4237 0.816
Concordance= 0.579 (se = 0.022 )
Rsquare= 0.046 (max possible= 0.999 )
Likelihood ratio test= 10.63 on 1 df, p=0.001111
Wald test = 10.09 on 1 df, p=0.001491
Score (logrank) test = 10.33 on 1 df, p=0.001312
Cox回归结果可以解释为:
统计意义。标记为“ z”的列给出了Wald统计值。它对应于每个回归系数与其标准误差的比率(z = coef / se(coef))。Wald统计量会评估beta给定变量的系数在统计上显着不同于0。从上面的输出中,我们可以得出结论,变量性别具有很高的统计学上显着的系数。
回归系数。Cox模型结果中要注意的第二个特征是回归系数(coef)的符号。正号表示该变量值较高的受试者的危险(死亡风险)较高,因此预后较差。变量sex被编码为数字向量。1:男性,2:女性。Cox模型的 R summary 提供了第二组相对于第一组(即女性与男性)的危险比(HR)。在这些数据中,性别的beta系数= -0.53表示女性比男性具有较低的死亡风险(较低的生存率)。
危险率。指数系数(exp(coef)= exp(-0.53)= 0.59),也称为危险比,给出了协变量的影响大小。例如,女性(性别= 2)可以将危险降低0.59或41%。女性是有良好预后的。
危险比的置信区间。摘要输出还给出了危险比(exp(coef))的上限和下限95%置信区间,下限95%= 0.4237,上限95%= 0.816。
该模型的全局统计意义。最后,输出为模型的总体重要性提供了三个备选检验的p值:似然比检验,Wald检验和得分对数秩统计。这三种方法渐近等效。对于足够大的N,它们将给出相似的结果。对于较小的N,它们可能会有所不同。对于小样本量,似然比测试具有更好的行为,因此通常是首选。
要将单变量coxph函数一次应用于多个协变量,请输入以下命令:
covariates <- c("age", "sex", "ph.karno", "ph.ecog", "wt.loss")
univ_formulas <- sapply(covariates,
function(x) as.formula(paste('Surv(time, status)~', x)))
univ_models <- lapply( univ_formulas, function(x){coxph(x, data = lung)})
# Extract data
univ_results <- lapply(univ_models,
function(x){
x <- summary(x)
p.value<-signif(x$wald["pvalue"], digits=2)
wald.test<-signif(x$wald["test"], digits=2)
beta<-signif(x$coef[1], digits=2);#coeficient beta
HR <-signif(x$coef[2], digits=2);#exp(beta)
HR.confint.lower <- signif(x$conf.int[,"lower .95"], 2)
HR.confint.upper <- signif(x$conf.int[,"upper .95"],2)
HR <- paste0(HR, " (",
HR.confint.lower, "-", HR.confint.upper, ")")
res<-c(beta, HR, wald.test, p.value)
names(res)<-c("beta", "HR (95% CI for HR)", "wald.test",
"p.value")
return(res)
#return(exp(cbind(coef(x),confint(x))))
})
res <- t(as.data.frame(univ_results, check.names = FALSE))
as.data.frame(res)
beta HR (95% CI for HR) wald.test p.value
age 0.019 1 (1-1) 4.1 0.042
sex -0.53 0.59 (0.42-0.82) 10 0.0015
ph.karno -0.016 0.98 (0.97-1) 7.9 0.005
ph.ecog 0.48 1.6 (1.3-2) 18 2.7e-05
wt.loss 0.0013 1 (0.99-1) 0.05 0.83
上面的输出显示了每个变量相对于总生存率的回归beta系数,效应大小(以危险比给出)和统计显着性。通过单独的单变量Cox回归评估每个因素。
从上面的输出中,
性别,年龄和ph.ecog变量具有极高的统计显着性系数,而ph.karno的系数则不显着。
年龄和ph.ecog的β系数为正,而性别的系数为负。因此,年龄更大和phogog较高与较差的生存率有关,而女性(性别= 2)则与较好的生存率有关。
现在,我们要描述这些因素如何共同影响生存。为了回答这个问题,我们将执行多元Cox回归分析。由于变量ph.karno在单变量Cox分析中不重要,因此在多变量分析中将其跳过。我们将3个因素(性别,年龄和ph.ecog)纳入多元模型。
多元Cox回归分析
时间常数协变量的死亡时间的Cox回归指定如下:
res.cox <- coxph(Surv(time, status) ~ age + sex + ph.ecog, data = lung)
summary(res.cox)
Call:
coxph(formula = Surv(time, status) ~ age + sex + ph.ecog, data = lung)
n= 227, number of events= 164
(1 observation deleted due to missingness)
coef exp(coef) se(coef) z Pr(>|z|)
age 0.011067 1.011128 0.009267 1.194 0.232416
sex -0.552612 0.575445 0.167739 -3.294 0.000986 ***
ph.ecog 0.463728 1.589991 0.113577 4.083 4.45e-05 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
exp(coef) exp(-coef) lower .95 upper .95
age 1.0111 0.9890 0.9929 1.0297
sex 0.5754 1.7378 0.4142 0.7994
ph.ecog 1.5900 0.6289 1.2727 1.9864
Concordance= 0.637 (se = 0.026 )
Rsquare= 0.126 (max possible= 0.999 )
Likelihood ratio test= 30.5 on 3 df, p=1.083e-06
Wald test = 29.93 on 3 df, p=1.428e-06
Score (logrank) test = 30.5 on 3 df, p=1.083e-06
所有三个总体测试(似然性,Wald和得分)的p值均显着,表明该模型具有显著性。这些测试评估了所有beta的综合零假设为0。在上面的示例中,检验统计量非常一致,并且完全拒绝了综合零假设。
在多变量Cox分析中,协变量性别和ph.ecog保持显着性(p <0.05)。但是,协变量年龄不显着(p = 0.23,大于0.05)。
性别的p值为0.000986,危险比HR = exp(coef)= 0.58,表明患者的性别与死亡风险降低之间有很强的关系。协变量的危险比可解释为对危险的倍增效应。例如,保持其他协变量不变(女性(性别= 2))可将危险降低0.58或42%。我们得出结论,成为女性与良好的预后相关。
同样,ph.ecog的p值为4.45e-05,危险比HR = 1.59,表明ph.ecog值与死亡风险增加之间有很强的关系。保持其他协变量不变,ph.ecog的值越高,生存率越低。
相比之下,年龄的p值现在为p = 0.23。危险比HR = exp(coef)= 1.01,95%置信区间为0.99至1.03。由于HR的置信区间为1,因此这些结果表明,在调整phog值和患者的性别之后,年龄对HR差异的贡献较小,并且仅趋于显着。例如,在其他协变量保持不变的情况下,再增加一岁会引起每日死亡危险,其系数为expβ= 1.01或1%,这并不是一个重要的贡献。
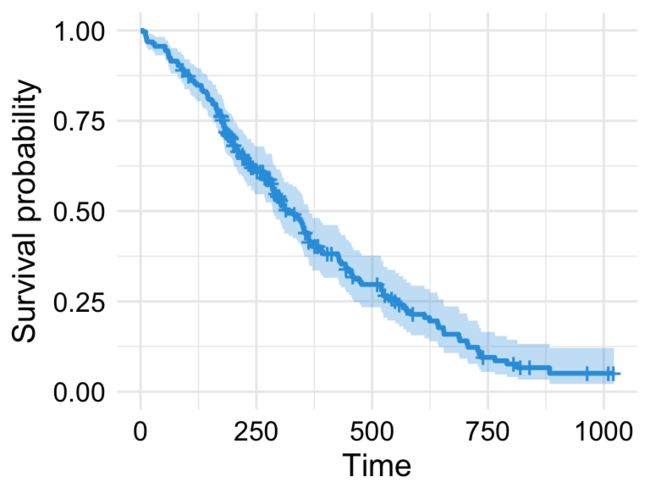
可视化生存时间的估计分布
将Cox模型拟合到数据后,就可以可视化特定风险组在任何给定时间点的预测生存率。函数survfit()估计生存比例,默认情况下为协变量的平均值。
# Plot the baseline survival function
ggsurvplot(survfit(res.cox), color = "#2E9FDF",
ggtheme = theme_minimal())
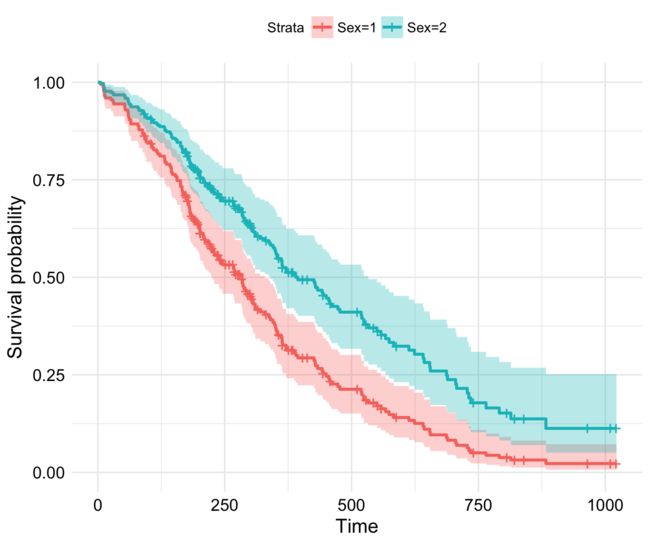
我们不妨展示估计的生存率如何取决于目标协变量的值。
考虑到这一点,我们想评估性别对估计生存率的影响。在这种情况下,我们用两行构造一个新的数据帧,每一行代表性别。其他协变量固定为其平均值(如果是连续变量)或最低水平(如果它们是离散变量)。对于伪协变量,平均值为数据集中编码为1的比例。该数据帧通过newdata参数传递给survfit():
# Create the new data
sex_df <- with(lung,
data.frame(sex = c(1, 2),
age = rep(mean(age, na.rm = TRUE), 2),
ph.ecog = c(1, 1)
)
)
sex_df
sex age ph.ecog
1 1 62.44737 1
2 2 62.44737 1
# Survival curves
fit <- survfit(res.cox, newdata = sex_df)
ggsurvplot(fit, conf.int = TRUE, legend.labs=c("Sex=1", "Sex=2"),
ggtheme = theme_minimal())
概要
在本文中,我们描述了Cox回归模型,用于同时评估多种风险因素与患者生存时间之间的关系。我们演示了如何使用生存包计算Cox模型。此外,我们描述了如何使用survminer软件包来可视化分析结果。
参考文献
- 考克斯博士(1972)。回归模型和寿命表(有讨论)。JR Statist Soc B 34:187–220
- MJ Bradburn,TG Clark,SB Love和DG Altman。生存分析第二部分:多元数据分析–概念和方法介绍。英国癌症杂志(2003)89,431 – 436