mice包--R中数据缺失值的处理
来源 | R友舍
简介
缺失值是一个实际数据处理中常见的问题。其缺失机制大致可以分为完全随机缺失(MCAR),非随机缺失(MNAR)
缺失的例子说明:假设一个数据集有3个变量 X1 , X2 , Y ,假设 X1 , X2 是完全变量, Y存在缺失值,那么
当 Y 以0.5概率缺失,为MCAR
当 X1<0 或者当 Y<0 , Y 发生缺失,为MNAR
对于完全随机缺失,直接剔除一般不会带来偏误,而对于非完全随机缺失,由于缺失值出现的位置可能和其他变量有关联,带来的后果则相对复杂得多。
常见处理方法
删除法
直接删除有缺失值的样本
删除存在大面积缺失值的变量。或完全变量分析:如果研究的问题只涉及到全部变量中的一部分变量,这部分变量是完整的,那么可以只分析这几个完整变量之间的关系
填补法
单变量填补
简单随机填补:对于每一个缺失值,从已有的该变量数据中随机抽样作为填补值,填补进缺失位置。仅仅考虑到了缺失变量本身,而并没有考虑到相关变量的信息。因此,信息量的利用少。
均值/中位数/分位数填补:用存在缺失值的变量的已有值的均值/中位数/分位数,作为填补值。这种方法显然会导致方差偏小。
回归填补:将缺失变量作为因变量,相关变量(其他变量)作为自变量,进行回归拟合,用预测值作为填补值。用于作为自变量的变量最好是具有完全数据(无缺失)。
热平台和冷平台:热平台法又称匹配插补法,思路是在完全数据样本中,找到一个和具有缺失值的样本相似的完全数据样本,用完全数据样本值作为填充值,其过程有点类似于K阶近邻的思想。冷平台法又称条件均值插补法,思路是先将总体分层(聚类),采用样本所在层(类)的完全数据的均值来替代缺失值。
多变量填补
回归插补法:对缺失变量和完全数据变量拟合多元回归模型来预测缺失值。是多重填补法的一种应用。多重填补法(Multiple Imputation Missing Data)的具体技术方法众多且相对复杂,限于篇幅此处不一一展开。
基于mice包的缺失值填补方法
mice即是基于多重填补法构造的。基本思想是对于一个具有缺失值的变量,用其他变量的数据对这个变量进行拟合,再用拟合的预测值对这个变量的缺失值进行填补。
数据准备(可跳过)
set.seed(2016)data <- airqualitydata[sample(nrow(data),7),3] <- NAdata[sample(nrow(data),7),4] <- NAdata <- data[-c(5,6)]采用R自带的airquality数据,其第一和第二列数据已经有很多缺失值,现在我们再人为地在第三个第四列中加入随机的7个缺失值。由于最后两列是月份和日期,不适合作为自变量,所以在缺失值填充中先剔除掉。
一个简单可用的版本
以下代码起到一个初始化的作用,使用时只需要改data为你自己需要填补缺失值的数据集名称
library(mice)init = mice(data, maxit=0)meth = init$methodpredM = init$predictorMatrix如果不想要使全部变量都参与拟合,比如排除掉Ozone变量,加入以下代码:
predM[, c("Ozone")]=0当然,即使你排除了拟合变量,并不意味着简单的将其排除,他只是不作为拟合变量,但仍然会作为被拟合变量,进行缺失值填充。
如果你想要跳过某个变量,如Temp,不对其填补,加入以下代码:
meth[c("Temp")]=""在这种情况下,虽然这个变量不会被作为被拟合变量进行填充,但仍然会作为拟合变量用于拟合其他变量的缺失值。
对于每个变量的拟合,可以指定所用的拟合方法:
meth[c("Ozone")]="norm"meth[c("Solar.R")]="logreg"meth[c("Wind")]="polyreg"=后面的双引号内的即为方法的名字,=前面的双引号中的为所需指定的变量。norm代表贝叶斯线性回归,logreg代表logit回归拟合,polyreg代表多项式拟合。
设定完成后执行填充:
imputed = mice(data, method=meth, predictorMatrix=predM, m=5)## ## iter imp variable## 1 1 Ozone Solar.R Wind Temp## 1 2 Ozone Solar.R Wind Temp## 1 3 Ozone Solar.R Wind Temp## 1 4 Ozone Solar.R Wind Temp## 1 5 Ozone Solar.R Wind Temp...这里我们选择让所有变量都进入拟合,每个变量的方法用默认方法(即不运行前面的设定代码),注意变量拟合时采用方法需要根据不同变量的情况选择,否则可能导致效果不佳或者算法无法计算。这需要事先对数据情况有一定了解。另外作为一个例子,本例仅仅作为代码演示,并不代表正确的参数选择方法。
输出填充结果到imputed:
imputed <- complete(imputed)检查是否存在缺失值
sapply(imputed, function(x) sum(is.na(x)))## Ozone Solar.R Wind Temp ## 0 0 0 0到这里就已经可以解决大部分缺失值问题了,如果想继续深入了解mice包的用法,请看下一节。
进阶版本
对分类变量的填充效果一般不是非常好,不建议对分类变量(categorical variables )做填充,本例中只对连续变量进行填充。
假设数据是MCAR的,一般数据量的5%的样本存在缺失值是相对安全的比例。
计算数据的缺失率
miss <- function(x){sum(is.na(x))/length(x)*100}apply(data,2,miss)## Ozone Solar.R Wind Temp ## 24.183007 4.575163 4.575163 4.575163apply(data,1,miss)## [1] 25 0 0 0 50 25 0 0 25 25 25 0 0 0 0 0 0 25 0 0 25 25 0## [24] 0 25 25 50 25 0 0 0 25 50 25 25 25 25 0 25 0 25 25 25 0 25 25## [47] 0 0 0 0 0 25 25 25 25 25 25 25 50 25 25 0 0 0 25 0 0 0 0## [70] 0 0 50 0 0 25 0 0 0 0 0 0 0 25 25 0 0 0 0 0 0 25 0## [93] 0 0 0 25 25 25 0 0 0 25 25 0 0 0 25 0 0 0 0 0 0 0 25## [116] 0 0 0 25 0 0 0 0 0 0 0 0 25 25 0 0 0 0 0 0 0 25 0## [139] 0 0 0 0 0 0 0 0 0 0 0 25 0 0 0从变量角度看,可以看到Qzone的缺失率高达25%,因此我们可能需要剔除这个变量。
对于样本,由于只有4个变量,缺失一个就有高达25%的缺失率,权衡一下样本量,我们可以选择剔除缺失高达50%和以上的样本。
使用mice判断缺失情况
对于上述缺失率等缺失情况的计算,在mice包中还有更简便的函数:
md.pattern(data)## Solar.R Wind Temp Ozone ## 100 1 1 1 1 0## 32 1 1 1 0 1## 5 0 1 1 1 1## 6 1 0 1 1 1## 5 1 1 0 1 1## 2 0 1 1 0 2## 1 1 0 1 0 2## 2 1 1 0 0 2## 7 7 7 37 58最左边一列是样本数,右边的0-1矩阵的1代表列对应的变量没有缺失,0代表有缺失。最右边一列是对应模式累计缺失值个数,最下面一列是变量累积存在的缺失模式个数。
假如 第三行的数据410111,表示有4个样本满足这行的缺失模式,1011表示这行的缺失模式是缺失了Solar.R变量,最右边的1表示这种缺失模式缺失了1个变量数据。
可以用VIM包获得缺失值的可视化表示
library(VIM)aggr_plot <- aggr(data, col=c('navyblue','red'), numbers=TRUE, labels=names(data), cex.axis=.7, gap=3, ylab=c("Histogram of missing data","Pattern"))marginplot(data[c(1,2)])
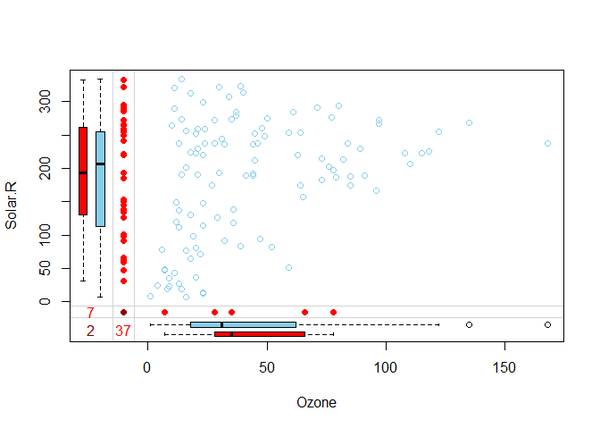 marginplot(data[c(1,2)])一次只表示2个变量的缺失情况,这里表示了第一和第二列变量。
marginplot(data[c(1,2)])一次只表示2个变量的缺失情况,这里表示了第一和第二列变量。
左边的红色箱线图表示有Ozone缺失的样本的Solar.R的分布,蓝色的箱线图表示的是剩下的数据点的分布。下方的2个箱线图的含义类似。
在MCAR的假设下,蓝色和红色的箱线图应该非常接近。
填充缺失值
这次我们详细介绍mice函数的用法
tempData <- mice(data,m=5,maxit=50,meth='pmm')summary(tempData)## Multiply imputed data set## Call:## mice(data = data, m = 5, method = "pmm", maxit = 50)## Number of multiple imputations: 5## Missing cells per column:## Ozone Solar.R Wind Temp ## 37 7 7 7 ## Imputation methods:## Ozone Solar.R Wind Temp ## "pmm" "pmm" "pmm" "pmm" ## VisitSequence:## Ozone Solar.R Wind Temp ## 1 2 3 4 ## PredictorMatrix:## Ozone Solar.R Wind Temp## Ozone 0 1 1 1## Solar.R 1 0 1 1## Wind 1 1 0 1## Temp 1 1 1 0## Random generator seed value: NAmice()函数用于生成填充矩阵tempData,其中的参数有:
data,需要填充的数据集
m,多重填补法的填补矩阵数。默认为5
method,填补用的方法,pmm代表预测均值匹配(predictive mean matching),用 methods(mice) 可以看到有哪些可用的方法
maxit,迭代次数,默认50次
如果想要查看填充的是那些值可以用以下代码:
tempData$imp$Temp## 1 2 3 4 5## 1 83 85 84 79 83## 9 72 77 72 57 80## 33 68 93 82 68 84## 41 87 78 81 82 78## 59 82 82 68 75 58## 129 82 79 71 76 76## 137 69 75 56 73 81最左边的一列表示的是被填充样本序号,之后的5列是多重填补法生成的5个填补矩阵对这个变量产生的填充数。
调用meth值可以查看每个变量用的是什么方法(前面我们指定了pmm方法)
tempData$meth## Ozone Solar.R Wind Temp ## "pmm" "pmm" "pmm" "pmm"当然也可以如前一节一样对每个变量指定各自的拟合方法。
最后生成完全数据集:
1表示用tempData$imp中的第一个矩阵来填充,如果想要用其他矩阵则可以改成2,3,4等等。
最担心的事情终于发生了,APP已经可以一键“脱掉”你的衣服了
我造的假我自己打,Adobe推出“反PS”
中国诚信全球垫底?讲讲《Science》现在的论文有多不靠谱
![]()