k均值聚类算法案例 r语言iris_R语言进阶之聚类分析
R语言拥有大量和聚类分析相关的函数,在这里我主要会和大家介绍K-means聚类、层次聚类和基于模型的聚类。
1. 数据预处理
在进行聚类分析之前,你需要进行数据预处理,这里主要包括缺失值的处理和数据的标准化。我们仍然以鸢尾花数据集(iris)为例进行详细讲解:
# 数据预处理mydata 1:mydata # 删除缺失值mydata # 数据标准化2. K-means聚类
在聚类分析中,K-means聚类算法是最常用的,它需要分析者先确定要将这组数据分成多少类,也即聚类的个数,这个通常可以用因子分析的方法来确定。比如我们可以用“nFactors”包的函数来确定最佳的因子个数,将因子数作为聚类数,不过关于聚类个数的确定还要考虑数据的实际情况与自身需求,这样分析才会更具有现实意义。 另外,我们也可以通过绘制碎石图来确定聚类个数,这和主成分的思想相似。
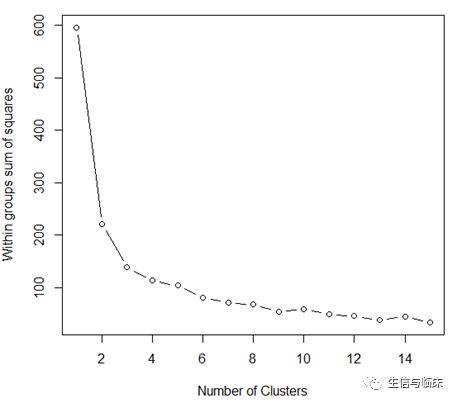
# 利用碎石图确定聚类个数wss # 计算离均差平方和for (i in 2:15) wss[i] centers=i)$withinss) #计算不同聚类个数的组内平方和plot(1:15, wss, type="b", xlab="Number of Clusters", ylab="Within groups sum of squares") # 绘图一般我们需要控制组内平方和的值要小,同时聚类的个数也不能太多,所以从图中可以看出聚类个数定在2~3比较好。
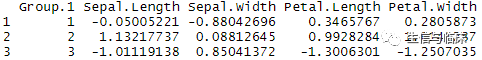
# K-Means聚类分析fit1 3) # 获取聚类均值aggregate(mydata,by=list(fit1$cluster),FUN=mean) # aggregate()是一个分类汇总函数从上面的结果中我们可以看出不同类别的各变量均值,从而对各类的特征有总体的了解,比如第2类是花瓣和花萼都普遍偏大的一类。
# 返回聚类的结果res $cluster)大家可以拿返回的聚类结果和真实分类对比一下,看看此次聚类效果如何。
3. 层次聚类
R语言提供了丰富的层次聚类函数,这里我给大家简单介绍一下用Ward方法进行的层次聚类分析。
# Ward层次聚类d "euclidean") fit2 "ward.D") plot(fit2) # 绘制树状图展示聚类结果groups 3) # 给聚成的3个类别加上红色边框rect.hclust(fit2, k=3, border="red")4. 基于模型的聚类
基于模型的聚类方法利用极大似然估计法和贝叶斯准则在大量假定的模型中去选择最佳的聚类模型并确定最佳聚类个数。我们可以使用R包“mclust”的Mclust()函数来实现这种模型聚类分析,同时你可以通过help(mclustModelNames)去查看各类模型的详细信息。
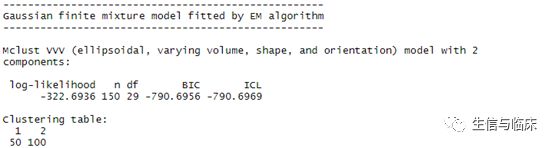
# 基于模型的聚类分析library(mclust)fit3 plot(fit3) # 绘图summary(fit3) # 输出结果从上面的结果来看,将总体聚成两类比较合适!
4. 聚类结果形象化展示
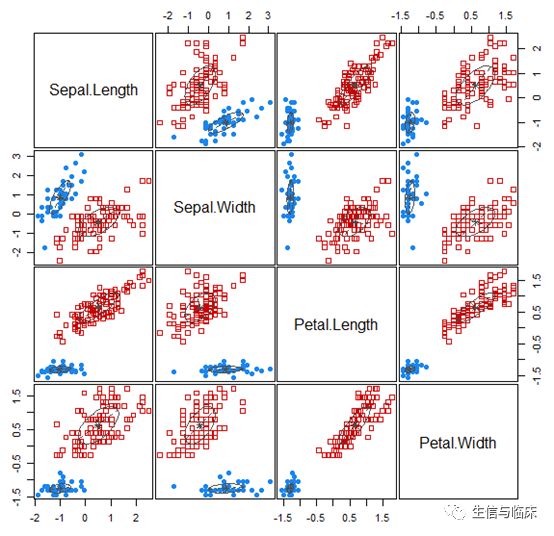
# 聚类结果展示# 将原数据聚成两类fit4 2)# 用前两个主成分绘制聚类图library(cluster)clusplot(mydata, fit4$cluster, color=TRUE, shade=TRUE, labels=2, lines=0)从图中看,样本被清晰分成两类,结果看起来挺不错的。
经过上述一系列的聚类分析,我们发现:如果仅仅使用花瓣和花萼的数据,鸢尾花数据集聚成两类最好,其中第一类是“setosa”,第二类则是“versicolor”和“virginica”。虽然该数据集自然分类是3类,但我们发现强行分成三类的效果并不好,这主要是因为仅仅利用花瓣和花萼的数据还无法将“versicolor“和”virginica“这两类进行很好的区分。
其实,在之前的判别分析中,我们已经发现”setosa”这一类的判别结果和其余两类均没有重叠,而“versicolor“和”virginica“这两个数据的线性判别的重叠部分较多,不好区分。
最后,如果真正想提高聚类结果和真实分类的接近度,我们通常需要增加有效的变量,这个才是关键!