今天继续给大家分享2020年11月4日在3区《DNA and Cell Biology》(IF=3.191)刊登的一篇生信分析在肝癌研究应用的文章,文章标题:Bioinformatic Analysis to Identify a Multi-mRNA Signature for the Prediction of Metastasis in Hepatocellular Carcinoma
研究背景
肝细胞性肝癌HCC是肝癌的主要组织学亚型,占原发性肝癌的90%,是全世界癌症相关死亡率的第三大常见原因。遗传、表观遗传改变、慢性乙型肝炎、丙型肝炎病毒感染、黄曲霉毒素暴露、吸烟、肥胖和糖尿病等是肝癌的主要危险因素。肝癌预后差的原因是复发率和转移率高。所以这篇论文的作者采用了生信分析方法构建一个分子模型以筛选出更准确预测HCC患者的肿瘤转移风险。
给大家看一幅早期HCC的真实照片
A&D:MRI的照片;B:超声照片;C:CT照片
Holzwanger DJ, Madoff DC. Role of interventional radiology in the management of hepatocellular carcinoma: current status. Chin Clin Oncol. 2018 Oct;7(5):49. doi: 10.21037/cco.2018.07.04. Epub 2018 Aug 7.
HCC的临床分期
数据来源
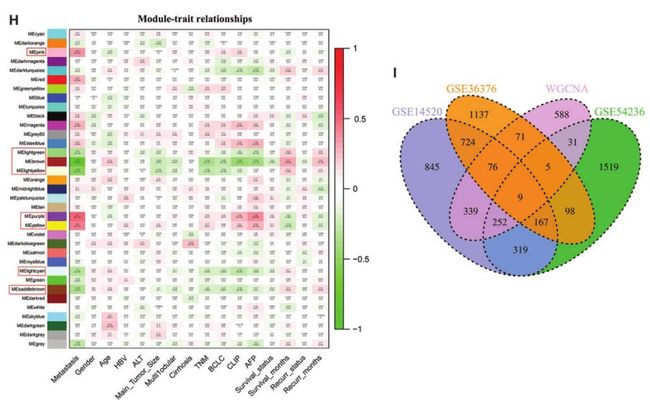
1.GEO数据库中获取包含1035个患者的GSE14520、GSE54236、GSE36376三个数据集。
2.选取GSE85680数据集分析血清外泌体的基因表达情况。
3.选取GSE36076数据集分析外周血单核细胞的基因表达情况。
研究思路
1.从公开数据库中获取HCC相关的数据集,准备做数据分析。
2.做差异表达分析和基因共表达网络分析
3.做LASSO Cox回归模型
4.风险评分模型的验证
5.hubgene的功能富集分析和蛋白互作网络的构建
6.用SPSS软件做统计学分析,P值小于0.05则具有显著性差异。
研究结果
1.鉴定转移性HCC相关的DEGs
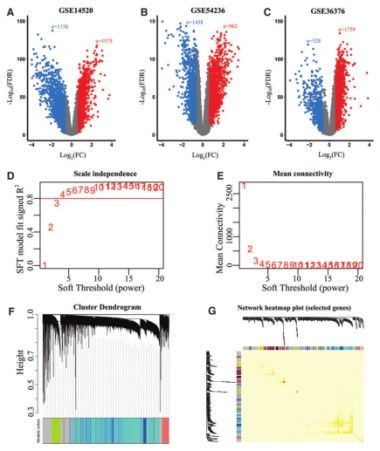
作者用了GER2R网络工具从GSE14520、GSE54236、GSE36376数据集鉴定DEGs。接着用WGCNA方法对GSE14520进行分析,结果发现共有8个基因模块明显地跟转移性HCC有相关性。最后把245个上调基因和97个下调基因鉴定出来并做进一步分析鉴定。
2.构建转移性HCC的mRNA分子识别标志
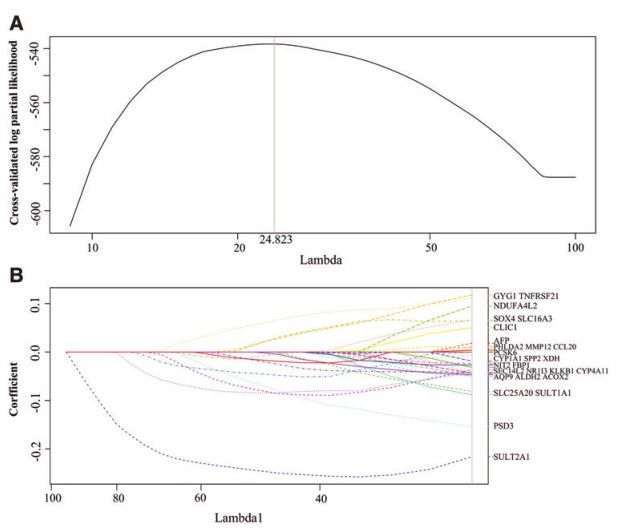
作者用那342个具有相似生物学作用的DEGs从GSE14520数据集中构建出能有效预测HCC metastasis的mRNA signatures。作者把221个患者的基因分类为高表达和低表达。P<0.25和AUC>0.55范围内选出候选mRNA并构建LASSO的Cox模型。分析的基因包括糖蛋白GYG1、TNF受体超家族成员TNFRSF21等等。
3. 27个mRNA signature的GSEA和PPI网络
为了鉴定27个mRNA signature的生物学功能,作者进行基因富集分析。大多数基因参与外泌体和有膜的细胞器的形成以及参与代谢、转运和氧化还原反应,也跟HCC metastasis
过程有着密切关系。接下来,作者用STRING数据库构建PPI网络,分析27个mRNA标志分子的潜在关系。结果发现每个标志分子都紧密相连着,它们都位于关键的节上。当中三个基因SOX4、SLC25A20、TNFRSF21在外周血单核细胞中差异表达。
文章的主要亮点
-作者自己构建了综合性多个mRNA识别标志作为预测早期转移性HCC的模型。
-作者他们分析的27个mRNA基因,发现全部都富集在外泌体、膜界细胞器和电子载体,还参与到转运和代谢生物学过程中。
-作者用WGCNA和LASSO方法筛选出的基因都参与了肿瘤形成过程,跟HCC转移有关。
文章的不足之处
--文章中的基因转录数据只是用RNA测序完成的,而非定量RT-PCR。因为RNA测序不需要用内参基因。
--文章的数据都是来自于网络上公开数据库,有些关键的临床病理特征是找不到的,这可能限制了mRNA识别标志的应用。
--这篇研究论文也没有用临床样本做过验证。所以在下一步研究需要用临床样本以能确认这些分子标志具有预测HCC转移的价值。
文章的研究结论
总体而言,文章作者通过网络数据库,构建出一个能预测HCC转移的模型。另,这27个基因面板可以用于寻找共调控miRNA网络以及预测转移性HCC的分子标志物。因此作者这项研究能在临床上进一步帮助改进高转移性HCC患者的个性化治疗策略。
今日分享到此结束,如果有朋友对生信工具不太熟悉,赶紧点赞,跟着我们一起学习吧~
我们时刻都在努力学习,这样可以带给你更多帮助!