(二)PCA数学原理推导及python实现(待修改)
主成分分析(principal component analysis)是一种常用的无监督学习方法。利用降维的思想,把多指标转化为少数几个综合指标。PCA属于有损压缩。
原数据集的变量很多,变量之间可能存在相关性,从而增加了问题分析的复杂性。如果分别对每个指标进行分析,分析往往是孤立的,不能完全利用数据中的信息,因此盲目减少指标会损失很多有用的信息,从而产生错误的结论。考虑将关系紧密的变量变成尽可能少的新变量,使这些新变量是两两不相关的,那么就可以用较少的综合指标分别代表存在于各个变量中的各类信息。主成分分析与因子分析就属于这类降维算法。
降维在一定的信息损失范围内,可以为我们节省大量的时间和成本。使数据更容易使用。能去除噪声。使得结果更容易理解。
一:算法流程
对于数据矩阵 X n × p X_{n\times p} Xn×p,每一行代表一个样本,每一列代表不同的属性。
- X标准化,将矩阵X每一列减去该列的平均值,得到 X ~ \tilde {X} X~使得不同样本之间同一属性的均值为0。
- 计算 X ~ \tilde {X} X~的协方差矩阵 S = 1 n X ~ T X ~ S=\frac{1}{n} \tilde {X}^T \tilde {X} S=n1X~TX~。
- 求协方差矩阵S的特征值和特征向量,将特征值按从大到小的顺序排列,相对应特征向量纵向排列。或者对S进行SVD分解,
S = U Σ V T S=U\Sigma V^{T} S=UΣVT
U即特征向量集合,即新坐标系的集合 Σ \Sigma Σ 为特征值集合(计算时特征值都会大于0,且结果会从小到大排列) - 将特征值从大到小排列,确定降低为K维,取前几个特征值所对应的特征向量,即为新坐标系的坐标轴。
- 降维之后数据表示为 X n e w = X ~ ( u 1 , u 2 , . . . u k ) X_{new}=\tilde {X}(u_1, u_2,...u_k) Xnew=X~(u1,u2,...uk)
二:原理解释
1. 坐标变换
u ⃗ , v ⃗ \vec{u} ,\vec{v} u,v为单位正交列向量, A o l d , A n e w A_{old},A_{new} Aold,Anew是坐标点,列向量表示。
A n e w = ( u ⃗ , v ⃗ ) T A o l d A_{new}=(\vec{u} ,\vec{v})^TA_{old} Anew=(u,v)TAold
例 ( 5 2 − 1 2 ) \begin{pmatrix} \frac {5}{\sqrt{2}} \\ -\frac {1}{\sqrt{2}} \end{pmatrix} (25−21) = ( 1 2 − 1 2 1 2 1 2 ) T \begin{pmatrix} \frac {1}{\sqrt{2}} & -\frac {1}{\sqrt{2}} \\ \frac {1}{\sqrt{2}} & \frac {1}{\sqrt{2}} \end{pmatrix}^T (2121−2121)T ( 3 2 ) \begin{pmatrix} 3 \\ 2 \end{pmatrix} (32)
2. 拉格朗日算子
在 g ( x , y ) = c g(x,y)=c g(x,y)=c的条件下,求 f ( x , y ) f(x,y) f(x,y)的极值。
构造 F ( x , y , z ) = f ( x , y ) + λ [ c − g ( x , y ) ] F(x,y,z)=f(x,y)+\lambda[c-g(x,y)] F(x,y,z)=f(x,y)+λ[c−g(x,y)]求解
{ ∂ F ∂ x = 0 ∂ F ∂ y = 0 ∂ F ∂ λ = 0 \begin{cases} \frac {\partial F}{\partial{x}}=0 \\ \frac {\partial F}{\partial{y}}=0 \\ \frac {\partial F}{\partial{\lambda}}=0 \end{cases} ⎩⎪⎨⎪⎧∂x∂F=0∂y∂F=0∂λ∂F=0
3. 协方差矩阵
将矩阵 X X X标准化得到 X ~ \tilde {X} X~,得到协方差矩阵为 S = 1 n X ~ T X ~ S = \frac {1}{n} \tilde {X}^T \tilde {X} S=n1X~TX~
4. 特征值、特征向量
对于方阵 A n × n A_{n \times n} An×n, A u = λ u Au=\lambda u Au=λu, ( A − λ I ) u = 0 (A-\lambda I) u=0 (A−λI)u=0,满足
A = U Λ U − 1 A=U \Lambda U^{-1} A=UΛU−1
其中 U U U为特征向量, Λ \Lambda Λ为相应的特征值。
特征值分解可以得到特征值与特征向量,特征值表示的是这个特征到底有多重要,而特征向量表示这个特征是什么,可以将每一个特征向量理解为一个线性的子空间,我们可以利用这些线性的子空间干很多的事情。不过,特征值分解也有很多的局限,比如说变换的矩阵必须是方阵。
5. SVD(singular value decomposition)奇异值分解
对于矩阵 A m × n A_{m \times n} Am×n, A = U Σ V − 1 A=U \Sigma V^{-1} A=UΣV−1
U m × m , V n × n U_{m \times m},V_{n \times n} Um×m,Vn×n是标准正交矩阵,列向量为单位长度,且相互正交, U U T = I m , V V T = I n UU^T=I_m,VV^T=I_n UUT=Im,VVT=In。 Σ m × n \Sigma_{m \times n} Σm×n对角矩阵,对角元素非负,且从大到小排列。 A T A A^TA ATA的特征向量为V的列。 A A T AA^T AAT的特征向量为U的列。 A T A 或 A A T A^TA或AA^T ATA或AAT的特征值求平方根,构成 Σ \Sigma Σ.
A T A V = V Σ T U T U Σ V T V = V Σ T Σ A A T U = U Σ V T V Σ T U T V = U Σ Σ T A^TA V =V\Sigma^TU^TU\Sigma V^T V =V\Sigma ^T \Sigma \\ AA^T U = U\Sigma V^T V\Sigma^TU^T V =U\Sigma \Sigma ^T ATAV=VΣTUTUΣVTV=VΣTΣAATU=UΣVTVΣTUTV=UΣΣT
有一些SVD的实现算法可以先不求出协方差矩阵也能求出我们的右奇异矩阵V。也就是说,我们的PCA算法可以不用做特征分解而是通过SVD来完成,这个方法在样本量很大的时候很有效。实际上,scikit-learn的PCA算法的背后真正的实现就是用的SVD,而不是特征值分解。
6. PCA推导
思路:最大化数据投影后的方差
新的单位长度正交基 u j u_j uj,数据点 x i x_i xi(已标准化)在该基底上的投影为 x i T u j x_i^T u_j xiTuj,所有点在该基底的方差 J j J_j Jj为
J j = 1 n ∑ i = 1 n ( x i T u j ) 2 = u j T 1 n ∑ i = 1 n ( x i x i T ) u j = 1 n u j T X T X u j J_j = \frac{1}{n} \sum_{i=1}^n (x_i^T u_j )^2 = u_j^T \frac{1}{n} \sum_{i=1}^n (x_i x_i^T) u_j = \frac{1}{n} u_j^T X^T X u_j Jj=n1i=1∑n(xiTuj)2=ujTn1i=1∑n(xixiT)uj=n1ujTXTXuj
在 u j T u j = 1 u_j^T u_j = 1 ujTuj=1条件下求 J j J_j Jj极大值。
构造 F ( u j ) = 1 n u j T X T X u j + λ j ( 1 − u j T u j ) ∂ F ∂ u j = 0 ⟹ 1 n X T X u j = λ i u j F(u_j) = \frac{1}{n} u_j^T X^T X u_j + \lambda_j (1-u_j^T u_j) \\ \frac {\partial F}{\partial{u_j}}=0 \Longrightarrow \frac{1}{n} X^T X u_j = \lambda_i u_j F(uj)=n1ujTXTXuj+λj(1−ujTuj)∂uj∂F=0⟹n1XTXuj=λiuj
即当 u j , λ j u_j,\lambda_j uj,λj为协方差矩阵 S S S的特征向量和特征值时有极值。 J j = λ j J_j = \lambda_j Jj=λj
数据从p维投影到新的p维坐标,方差不变。即X的协方差阵的特征值相加对应X的方差。(此时的方差指数据在每个坐标轴上投影的方差相加。)
V a r X = ∑ j = 1 p J j = ∑ j = 1 p λ j VarX=\sum_{j=1}^p J_j=\sum_{j=1}^p \lambda_j VarX=j=1∑pJj=j=1∑pλj
第一个新坐标轴选择是原始数据中方差最大的方向,第二个新坐标轴选取是与第一个坐标轴正交的平面中使得方差最大的,第三个轴是与第1,2个轴正交的平面中方差最大的。大部分方差都包含在前面k个坐标轴中,后面的坐标轴所含的方差几乎为0。我们可以忽略余下的坐标轴,只保留前面k个含有绝大部分方差的坐标轴。
7. 选择降维后的维度k
以图像处理为例,一个惯例是保留99%的方差。对于其他领域的应用,可以保留90%~98%的方差。
满足下列式子的最小k值即为所求
1 n ∑ i = 1 n ∣ ∣ x ( i ) − x a p p r o x ( i ) ∣ ∣ 2 1 n ∑ i = 1 n ∣ ∣ x ( i ) ∣ ∣ 2 ≤ 0.01 \frac {\frac{1}{n} \sum_{i=1}^n||x^{(i)}-x_{approx}^{(i)}||^2} {\frac{1}{n} \sum_{i=1}^n||x^{(i)}||^2} \leq 0.01 n1∑i=1n∣∣x(i)∣∣2n1∑i=1n∣∣x(i)−xapprox(i)∣∣2≤0.01分子表示原始点与投影点之间的距离之和,而误差越小,说明降维后的数据越能完整表示降维前的数据。如果这个误差小于0.01,说明降维后的数据能保留99%的信息。等价于 1 − ∑ 1 k λ i ∑ 1 p λ i ≤ 0.01 1-\frac{\sum_1^k \lambda_i}{\sum_1^p \lambda_i} \leq 0.01 1−∑1pλi∑1kλi≤0.01
三:scikit-learn PCA
- 简介
在scikit-learn中,与PCA相关的类都在sklearn.decomposition包中。最常用的PCA类就是sklearn.decomposition.PCA。
补充:除了PCA类以外,最常用的PCA相关类还有KernelPCA类,它主要用于非线性数据的降维,需要用到核技巧。在使用的时候需要选择合适的核函数并对核函数的参数进行调参。另外一个常用的PCA相关类是IncrementalPCA类,它主要是为了解决单机内存限制的。有时候我们的样本量可能是上百万+,维度可能也是上千,直接去拟合数据可能会让内存爆掉, 此时我们可以用IncrementalPCA类来解决这个问题。IncrementalPCA先将数据分成多个batch,然后对每个batch依次递增调用partial_fit函数,这样一步步的得到最终的样本最优降维。此外还有SparsePCA和MiniBatchSparsePCA。他们和上面讲到的PCA类的区别主要是使用了L1的正则化,这样可以将很多非主要成分的影响度降为0,这样在PCA降维的时候我们仅仅需要对那些相对比较主要的成分进行PCA降维,避免了一些噪声之类的因素对我们PCA降维的影响。SparsePCA和MiniBatchSparsePCA之间的区别则是MiniBatchSparsePCA通过使用一部分样本特征和给定的迭代次数来进行PCA降维,以解决在大样本时特征分解过慢的问题,当然,代价就是PCA降维的精确度可能会降低。使用SparsePCA和MiniBatchSparsePCA需要对L1正则化参数进行调参。 - sklearn.decomposition.PCA参数介绍
PCA类基本不需要调参,一般来说,我们只需要指定我们需要降维到的维度,或者我们希望降维后的主成分的方差和占原始维度所有特征方差和的比例阈值就可以了。
- n_components:这个参数可以帮我们指定希望PCA降维后的特征维度数目。最常用的做法是直接指定降维到的维度数目,此时n_components是一个大于等于1的整数。当然,我们也可以指定主成分的方差和所占的最小比例阈值,让PCA类自己去根据样本特征方差来决定降维到的维度数,此时n_components是一个(0,1]之间的数。当然,我们还可以将参数设置为"mle", 此时PCA类会用MLE算法根据特征的方差分布情况自己去选择一定数量的主成分特征来降维。我们也可以用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- whiten :判断是否进行白化。所谓白化,就是对降维后的数据的每个特征进行归一化,让方差都为1.对于PCA降维本身来说,一般不需要白化。如果你PCA降维后有后续的数据处理动作,可以考虑白化。默认值是False,即不进行白化。
- svd_solver:即指定奇异值分解SVD的方法,由于特征分解是奇异值分解SVD的一个特例,一般的PCA库都是基于SVD实现的。有4个可以选择的值:{‘auto’, ‘full’, ‘arpack’, ‘randomized’}。randomized一般适用于数据量大,数据维度多同时主成分数目比例又较低的PCA降维,它使用了一些加快SVD的随机算法。 full则是传统意义上的SVD,使用了scipy库对应的实现。arpack和randomized的适用场景类似,区别是randomized使用的是scikit-learn自己的SVD实现,而arpack直接使用了scipy库的sparse SVD实现。默认是auto,即PCA类会自己去在前面讲到的三种算法里面去权衡,选择一个合适的SVD算法来降维。一般来说,使用默认值就够了。
- 除了这些输入参数外,有两个PCA类的成员值得关注。第一个是explained_variance_,它代表降维后的各主成分的方差值。方差值越大,则说明越是重要的主成分。第二个是explained_variance_ratio_,它代表降维后的各主成分的方差值占总方差值的比例,这个比例越大,则越是重要的主成分。
四:范例
(1)
import numpy as np
def pca(X,k):#k is the components you want
#mean of each feature
n_samples, n_features = X.shape #矩阵X的行数和列数
mean=np.array([np.mean(X[:,i]) for i in range(n_features)]) #把均值写成一个数组
#normalization
norm_X=X-mean #计算的时候自动补齐
#scatter matrix
scatter_matrix=1/n_samples * np.dot(np.transpose(norm_X),norm_X) #样本协方差阵 dot矩阵乘,*矩阵对应相乘 transpose默认为矩阵的转置
#Calculate the eigenvectors and eigenvalues
eig_val, eig_vec = np.linalg.eig(scatter_matrix) #计算矩阵特征值和特征向量
eig_pairs = [(np.abs(eig_val[i]), eig_vec[:,i]) for i in range(n_features)] #abs绝对值 把特征值对应的特征向量放一块
# sort eig_vec based on eig_val from highest to lowest
eig_pairs.sort(reverse=True) #降序排列
# select the top k eig_vec
feature=np.array([ele[1] for ele in eig_pairs[:k]]) #选出k个特征值对应的向量
#get new data
data=np.dot(norm_X,np.transpose(feature)) #向量显示为列向量
return data
X = np.array([[-1, 1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]])
print(X)
print(pca(X,1))
输出结果
[[-1 1]
[-2 -1]
[-3 -2]
[ 1 1]
[ 2 1]
[ 3 2]]
[[-0.50917706]
[-2.40151069]
[-3.7751606 ]
[ 1.20075534]
[ 2.05572155]
(2)
from sklearn.decomposition import PCA
import numpy as np
X = np.array([[-1, 1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]])
pca=PCA(n_components=1)
pca.fit(X)
print(pca.transform(X))
输出如果
[[ 0.50917706]
[ 2.40151069]
[ 3.7751606 ]
[-1.20075534]
[-2.05572155]
[-3.42937146]]
(3)在实际使用PCA时,我们不需要选择k,而是直接设置n_components为float数据。有的时候我们降维只是为了观测数据(visualization),这种情况下一般将k选择为2或3。
from sklearn.decomposition import PCA
import numpy as np
from sklearn.preprocessing import StandardScaler
x=np.array([[10001,2,55], [16020,4,11], [12008,6,33], [13131,8,22]])
# feature normalization (feature scaling)
X_scaler = StandardScaler()
x = X_scaler.fit_transform(x)
# PCA
pca = PCA(n_components=0.9)# 保证降维后的数据保持90%的信息
pca.fit(x)
print(pca.transform(x))
结果
[[ 2.36863319 0.38298087]
[-1.50952734 1.23481789]
[ 0.14360068 -0.58040917]
[-1.00270653 -1.03738959]]
(4) sklearn.decomposition.PCA参数介绍范例
#%%[1]生成随机数据并可视化
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
#%matplotlib inline
from sklearn.datasets import make_blobs
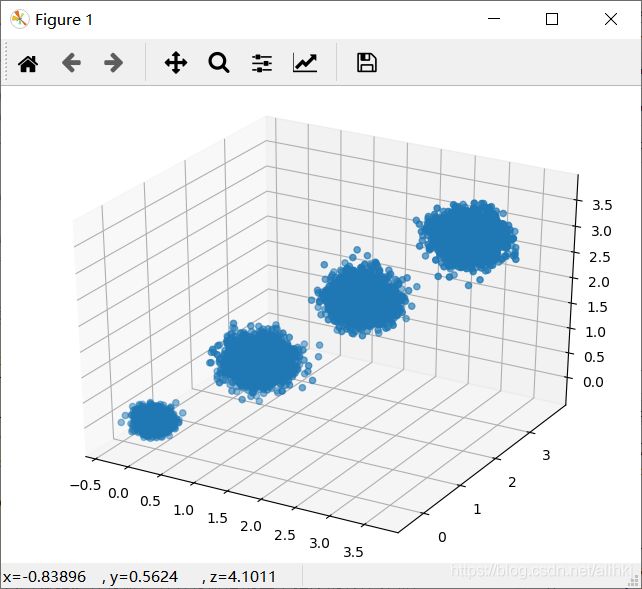
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本3个特征,共4个簇
X,y = make_blobs(n_samples=10000, n_features=3, centers=[[3,3, 3], [0,0,0], [1,1,1], [2,2,2]], cluster_std=[0.2, 0.1, 0.2, 0.2],
random_state =9)
fig = plt.figure()
ax = Axes3D(fig)
ax.scatter(X[:, 0], X[:, 1], X[:, 2],marker='o') #在ax3维坐标系的基础上绘制散点图
#%%[2]先不降维,只对数据进行投影,看看投影后的三个维度的方差分布,
from sklearn.decomposition import PCA
pca = PCA(n_components=3)
pca.fit(X)
print(pca.explained_variance_ratio_)
print(pca.explained_variance_)
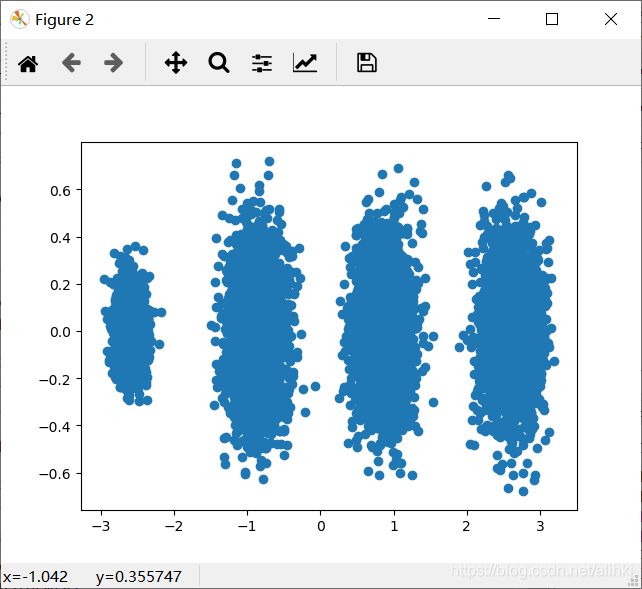
#%%[3]进行降维,从三维降到2维
pca = PCA(n_components=2)
pca.fit(X)
print(pca.explained_variance_ratio_)
print(pca.explained_variance_)
#%%[4]看看此时转化后的数据分布,
fig = plt.figure()
X_new = pca.transform(X)
plt.scatter(X_new[:, 0], X_new[:, 1],marker='o')
plt.show()
#%%[5]看看不直接指定降维的维度,而指定降维后的主成分方差和比例。95% 99%
pca = PCA(n_components=0.95)
pca.fit(X)
print(pca.explained_variance_ratio_)
print(pca.explained_variance_)
print(pca.n_components_)
pca = PCA(n_components=0.99)
pca.fit(X)
print(pca.explained_variance_ratio_)
print(pca.explained_variance_)
print(pca.n_components_)
#%%[6]MLE算法自己选择降维维度的效果
pca = PCA(n_components='mle')
pca.fit(X)
print(pca.explained_variance_ratio_)
print(pca.explained_variance_)
print(pca.n_components_)
[0.98318212 0.00850037 0.00831751]
[3.78521638 0.03272613 0.03202212]
[0.98318212 0.00850037]
[3.78521638 0.03272613]
不直接指定降维的维度,而指定降维后的主成分方差和比例。
[0.98318212]
[3.78521638]
1
[0.98318212 0.00850037]
[3.78521638 0.03272613]
2
让MLE算法自己选择降维维度的效果
[0.98318212]
[3.78521638]
1