不仅仅编译R语言本身会非常的麻烦,实际上还有些R包为了提高运行速度将一些功能封装到C/C++中,随后在安装的时候会进行编译。
编译通过则万事大吉,如果不通关就是一番折腾。比如说我最近在服务器上安装DESeq2就遇到了这种事情,下面是解决的过程。
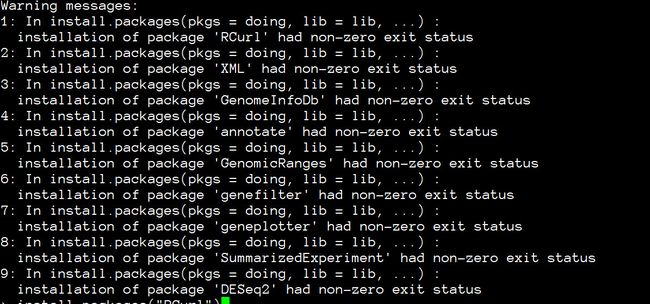
并不是所有的warning都可以忽视,比如说如下这种就不行。因为他说DESeq2的编译结果是“非零返回”,也就是失败了。而失败的原因则是前面这个包都失败了。
source("http://bioconductor.org/biocLite.R")
biocLite("DESeq2")
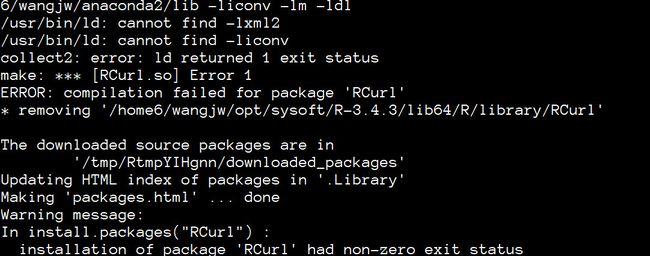
那我们逐个解决,使用install.packages("RCurl")安装第一个失败的包。
上面的报错信息cannot find -lxxx告诉我们,由于缺少两个动态库,xml和iconv, 导致编译不通过。那我们借助百度去安装这两个包,以xml为例
# xml2
wget -4 ftp://xmlsoft.org/libxml2/libxml2-git-snapshot.tar.gz
tar xf libxml2-git-snapshot.tar.gz
cd libxml2-2.9.7/
./configure --prefix=$HOME/opt/sysoft/libxml2-2.9.7 CFLAGS=-I$HOME/opt/anaconda2/include/python2.7/
make && make install
# iconv
wget -4 https://ftp.gnu.org/pub/gnu/libiconv/libiconv-1.15.tar.gz
tar xf libiconv-1.15.tar.gz
cd libiconv-1.15
./configure --prefix=$HOME/opt/sysoft/libiconv-1.15
make && make install
编译libxml需要Python的头文件,选择自己的Python安装路径。
安装完之后,还是无法直接使用R安装,因为R不知道我们把这些软件装在了哪里。解决方案就是在家目录下新建一个文件夹.R,然后在其中创建一个文件Makevars.
Makevars的语法和Makefile一致,在编译的时候里面的变量会成为编译工具的参数。
The most common use of a Makevars file is to set additional preprocessor options (for example include paths) for C/C++ files via
PKG_CPPFLAGS, and additional compiler flags by settingPKG_CFLAGS,PKG_CXXFLAGS,PKG_FFLAGSorPKG_FCFLAGS, for C, C++, FORTRAN or Fortran 9x respectively (see Creating shared objects).
由于我的服务器管理员安装的工具不多,所以我自己安装了很多工具,也就有很多的路径要添加。
CXXSTD = -std=gnu11
# dynamic link library
LIBGCC = -L$(HOME)/opt/sysoft/gcc-4.9.0/lib64 -Wl,-R$(HOME)/opt/sysoft/gcc-4.9.0/lib64
LIBEXPAT = -L$(HOME)/opt/sysoft/expat-2.2.5/lib -Wl,-R$(HOME)/opt/sysoft/expat-2.2.5/lib
OPENMPI = -L$(HOME)/opt/sysoft/openmpi-3.0.0/lib -Wl,-R$(HOME)/opt/sysoft/openmpi-3.0.0/lib
BZIP2 = -L$(HOME)/opt/sysoft/bzip2-1.06/lib -Wl,-R$(HOME)/opt/sysoft/bzip2-1.06/lib
XZ = -L$(HOME)/opt/sysoft/xz-5.2.3/lib -Wl,-R$(HOME)/opt/sysoft/xz-5.2.3/lib
ZLIB = -L$(HOME)/opt/sysoft/zlib-1.2.11/lib -Wl,-R$(HOME)/opt/sysoft/zlib-1.2.11/lib
CURL = -L$(HOME)/opt/sysoft/curl-7.56.1/lib -Wl,-R$(HOME)/opt/sysoft/curl-7.56.1/lib/
ICONV = -L$(HOME)/opt/sysoft/libiconv-1.15/lib -Wl,-R$(HOME)/opt/sysoft/libiconv-1.15/lib
XML2 = -L$(HOME)/opt/sysoft/libxml2-2.9.7/lib -Wl,-R$(HOME)/opt/sysoft/libxml2-2.9.7/lib
LDFLAGS := $(LIBGCC) $(LIBEXPAT) $(BZIP2) $(XZ) $(ZLIB) $(CURL) $(ICONV) $(XML2)
之后在编译XML时候,出了一个小问题,后来发现是因为编译XML需要xml2-config,这个工具位于我们刚刚编译的libxml2-2.9.7/bin下,所以将该目录添加到环境PATH中即可。
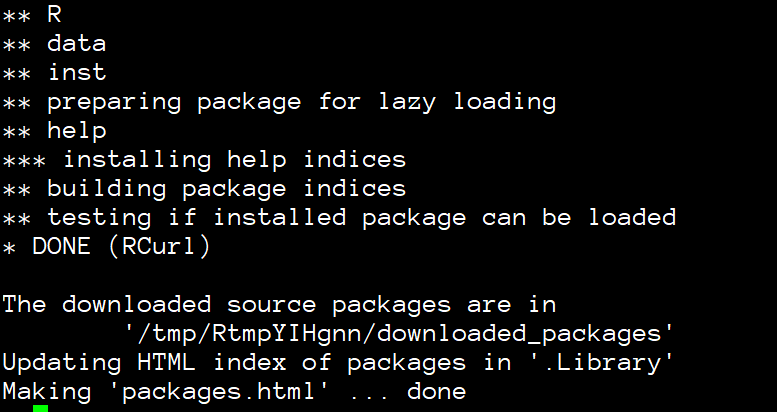
解决依赖包之后,DESeq2的也终于安装上去了。
参考资料:
- https://cran.r-project.org/doc/manuals/r-release/R-exts.html#Using-Makevars
- https://stackoverflow.com/questions/43597632/understanding-the-contents-of-the-makevars-file-in-r-macros-variables-r-ma
- http://www.ruanyifeng.com/blog/2015/02/make.html