这篇文献学习笔记分享的是一篇有关于增强子和启动子的综述。文章在今年2月发表在了Nature Reviews Genetics。这是一篇长笔记,没有时间看的同学可以收藏起来慢慢看,对于认识启动子和增强子的作用还是很有帮助的。当然更推荐大家下载英文原文阅读。
题目:Determinants of enhancer and promoter activities of regulatory elements
国内下载不了的童鞋请在这里下载文章:here
摘要
增强子的活性和基因启动子的活性,对于细胞内的协调转录过程是非常重要的。虽然有一些方法学可以用来鉴定增强子和启动子,目前大家一般默认的是这两种元件是不同的。然而,一些研究表明,基因的调控元件可能同时具备增强子和启动子的功能。在这篇综述里,作者主要总结一些研究结果,集中讨论决定调控元件活性的启动子和增强子。本文还讨论了目前一些通过DNA可接近性和非互斥能力起始(或增强)转录来鉴定调控元件的方法。
正文
转录调控元件在复杂的基因组里担任了一个重要的角色,在分化、细胞组织稳态、对外界刺激的应答和疾病里有着动态的作用。在病理相关的细胞类型中有相当一部分性状相关的遗传变异,并且它们的改变与一些先天性疾病和癌症有关。因此,了解调控元件及其功能的决定因素是一个重要挑战。
起激活作用的调控元件主要有两种:启动子和增强子。前者定义了转录的起始,后者决定了转录的加强。但是,现在在回顾这两个定义,其实是很模糊的。因为我们是根据区域的大小,而武断的把它们分成两种调控元件。随着基因组技术的驱动,人们已经对生物过程中鉴定启动子和增强子有了很大的进展。虽然这些技术使得在全基因组范围内识别调控元件成为可能,但它们也使得增强子和启动子具有一些共同的特性和功能。举个例子,它们的染色质结构和序列结构是非常相似的,有一些研究表明启动子也有着增强子的活性,另一些研究也表明,活化的增强子可以启动在其边界的转录的起始,从而扮演了启动子的角色。因此,增强子和启动子的区别越来越不清楚,需要开发新的模型来区别启动子和增强子。为了构建这种模型,有必要将感兴趣的调控元件的影响从其他调控要素和庞大的染色质环境中分离出来。一些研究提出了一些方法,可以大规模测定潜在的调控元件的启动子或增强子。从这些数据中,可以解释为什么调控元件同时具有启动子和(或)增强子的特征。
本文中我们主要回顾一些最近的研究结果,有关于启动子和增强子的特征,以及它们功能的决定性因素。我们还对比新的和以前的定义,根据DNA可接近性和潜在的增强子/启动子提出了一个更新的模型,列出了待解决的问题和挑战,以便我们今后会转录调控元件更深的理解。
(一)转录调控元件
RNA pol II活性已经被研究了好几十年了。这一过程的核心是RNA pol II转录起始位点(TSS)的选择,定义为第一个基因组核苷酸的转录本。TSS的选择过程受DNA结合的转录因子的影响(参考文献23、24),RNAPolII和其他因子形成RNAPolII前起始复合物。这种转录因子与特定DNA序列元件的结合通常位于在TSS周围的一定范围内,称为“核心启动子”,定义其为TSS周围的±50碱基对(bp)区域。核心启动子的作用被认为是决定TSS的精确位置以及转录的方向(通过转录因子和RNA polII与特定核心启动子序列元件结合)。
最有名的核心启动子元件是TATA box和initiator元件(INR)。TATA box最开始被认为是每一个TSS上游24-30bp的普遍元件,它被TATA结合蛋白(TBP)识别,TBP是前起始复合物的核心部分。 位于TSS的嘧啶-嘌呤INR元件也是如此,但使用全基因组方法对许多TSSs的研究表明,核心启动子结构是非常复杂和多样的。例如,在哺乳动物中只有一小部分的核心启动子有明确的TATA box。这可能是由于一般转录因子(GTF)作为一种复杂多样的、可交换的蛋白,具有序列偏好性。例如,转录因子TF IID包含TBP和TBP相关因子TAF,允许灵活进行核心启动子的识别。
然而核心启动子包含足够的信息,来选择TSS进行转录,RNA PolII起始速率可以被其他信号整合。DNA结合转录因子TF可以直接或间接的,通过募集共激活因子(CBP-p300、Mediator或者SAGA复合体)影响核心启动子RNA polII的募集、起始和延伸。这些转录因子可以是附近的,也可以是远端的,就核心启动子基因组的接近性而言,这些转录因子通常来自于顺式调控模块之间多个TFs的合作。因为一些TFs与核心启动子区域近端结合,而不是在核心启动子区域内,在TSS周围一个较大的区域,包括核心启动子和这个“近端启动子”区域,通常被称为“启动子”,尽管这个区域里可能只有一部分对调控转录起始是重要的。因此,如果“启动子”指的是一个基因组区域,它几乎总是随TSSs的位置来定义。
转录调控可能还被远端的转录因子结合事件所影响(就是通常说的增强子结合),有些增强子位于距离核心启动子1Mb远的位置。远端位置的调控是通过基因组的有利折叠,使得调控元件在三维空间上接近。增强子最初是在猴病毒里被发现的,然后在哺乳动物基因组里被发现,许多研究表明它们可以增加基因表达,无论它们的方向以及和核心启动子的距离。大部分早起的增强子研究工作依赖报告基因分析实验,把一个候选的增强子序列放在报告基因的minimal核心启动子的上游或者下游(box 1)。这项技术成为了增强子生物学研究的主力,直到最近才出现了高通量测序的研究手段。
增强子被发现的40多年来,虽然增强子功能的很多模式被报道,包括转录激活、RNA polII转移, RNA polII 启动子暂停/释放等等,几乎所有对增强子的定义都假定了增强子和启动子在基因组上的位置相距很远,有着不同的分子功能。
BOX1
在一个启动子报告分析实验里(下图a)基因组上的一个可能起始转录的DNA序列与一个报告基因融合,编码一个蛋白(比如说是绿色荧光蛋白),这样当基因表达的时候翻译成蛋白,就会产生信号。然后再把这一段序列进行截短,然后观察信号的强弱。这种方法可以精确到某一段携带全部或者大部分介导转录起始能力的序列。序列的截短过程通常被称为"promoter bashing"。而增强子报告实验(下图b)则是把基因组上某一段可能会增强转录的DNA序列融合到报告基因的minimal启动子的上游或者下游。这就可以测定不同的增强子增强minimal启动子的能力了。
(二)基因组内的调节元件
基于高通量DNA测序的一系列技术使得增强子和启动子在全基因组范围内大规模的研究,包括它们在不同生物过程中的作用,如发育、细胞类型分化,以及疾病。在这里,我们简要回顾检测和定量增强子和启动子的主要方法,这也将作为一个背景来说明我们对转录调控元件的整体理解是如何逐渐从最初明确的将的启动子/增强子区分开,而逐渐改变想法的。
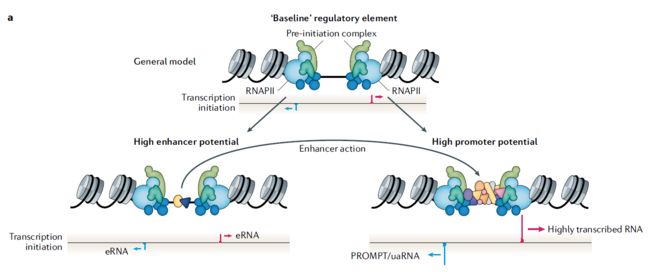
许多方法直接从测序稳定或新生RNA的5’端 (Box 2)从而直接识别基因的TSSs,使得该领域能够为绝大多数哺乳动物细胞绘制核心启动子区域。再加上通过对新生RNA的3 '端测序,从停滞的RNAPII间接推断启动子。这些技术揭示了一些在早期基于单基因报告分析的研究中没有发现的意想不到的特征(图1a)。这些特性包括以下几点:(1)首先,RNAPII起始通常分散在一个局部区域,在同一个核小体耗尽区域(NDR),从而导致多个接近的TSSs的位置有着不同的起始频率。(2)其次,大多数基因有许多不同的TSS簇(称为“alternative启动子”),它们来自于独立的NDRs中的核心启动子,通常相距数百或数千个bp,其中alternative启动子的选择可能改变最终的蛋白质产物。(3)第三,RNAPII在进入主动延伸状态之前停留在TSS下游。(4)第四,可能也是最出乎意料的,绝大多数基因TSSs都伴随着在相反链上的一个额外的近端、上游的TSS。后一种特征被称为“发散式转录”,它产生于每个TSS位于接近侧核小体边缘NDR的独立核心启动子上。一般来说,上游发散的TSS产生短的(<500 bp)未拼接的转录本,称为“启动子-上游转录本”(PROMPTs)或“上游反义RNA”(uaRNAs),可以被核外泌体降解(一种从3 '端降解RNA的外切酶复合物)。PROMPTs和mRNA之间对外泌体敏感性的差异,部分是由于缺失了早期poly(a)位点,以及mRNA TSSs下游的5 ' splice位点(5 ' SSs)的富集,以及PROMPT TSSs下游的相反模式。
BOX2 基因组范围内的基因TSS和核心启动子的鉴定
由于TSS是鉴定核心启动子的中心,基于RNA测序的技术对这项任务时非常有用的。这些技术基于全长cDNA测序(flcDNA-seq),通常基于大规模的Sanger测序,或者对全长cDNA最开始的20-50碱基高通量测序(例如CAGE和TSS-seq)(下图a)。后一种方法更有优势,可以同时鉴定TSS的位置,估测产生的RNA的丰度。
后来出现了一套互补的技术,可以评估转录速率而不是稳定状态RNA的数量。这些都是基于将标记的核苷酸合并到细胞中,并对3 '端进行测序((global run- on测序(GRO- seq))和精确核运行测序(PRO - seq)),或新生RNA的5′末端测序(GRO-cap, PRO-cap, 5′GRO - seq),或分离与RNA polII结合或染色质结合的RNA(例如NET-seq,哺乳动物NET-seq (mNET-seq), 新生转录本3′末端测序(3′NT-seq)或起始位点相关RNA测序(Start- seq))(下图b部分)。从技术上讲,对新生RNA3 '端进行测序的方法并不是为了检测TSSs而设计的,因为它们也可能从延长或暂停的RNAPII上捕获到RNA。然而,由于RNAPII在转录起始后短暂暂停,这些方法的reads峰可以作为转录起始的代表,从而检测启动子。
新生RNA测序技术的优势是RNA周转的影响有限,因此可以测量转录,而不是稳定状态的RNA水平。相反,稳态技术通常只需要少量的RNA,因此可以用于细胞数量有限的实验。
同时,ChIP-seq和ChIP-chip的相结合使得测量结合TFs和基因TSSs周围染色质状态成为可能,特别是对于特定的组蛋白翻译后修饰。研究发现转录活化的TSSs的核小体被耗尽,并在TSSs下游的第一个核小体中富集H3K4me3和H3K27ac。同样的技术还显示,绝大多数结合TFs的基因TSS附近都位于分叉的TSS对之间的NDR内,大多数活跃的基因启动子都有积累的停滞或暂停的RNAPII(图1a)。基于Chip和DNA可接近性的技术在早期的增强子研究中很有帮助(BOX3)。各种组蛋白修饰的特种证,特别是甲基化和乙酰化,推断结合增强子相关的共激活因子或在相关增强子研究中,建立了用于识别增强子和基因启动子的规则(图1 b)。使用这些特定的模式,比如高H3K27ac信号和高H3K4me1与H3K4me3的比例被默认为增强子,迅速成为几家大型研究联盟(例如ENCODE和Roadmap Epigenomics)在基因组区域划分为不同的类型的转录调控元件的方法。
当结合RNA测序和ChIP-seq技术时,一个意想不到的发现是许多候选增强子以不同的方式启动了NDRs边缘的所谓增强子RNA(eRNAs)的转录。eRNAs很短,通常是未剪接和非聚腺苷化的转录本,它们可以被外泌体降解,因此它们与基因启动子NDRs的PROMPTs有很多相似之处(图1b)。与基因启动子相反的是,在基因启动子中,PROMPTs的转录比mRNA的转录更少(图1a中蓝色reads和红色reads的峰高差很远),而候选增强子相关的转录通常更趋于双向平衡(图1b中蓝色reads和红色reads峰高差不多),而且这两种转录本都是外泌体底物。我们和其他人已经证明,基于这些特性,仅从5 '端RNA测序数据就可以预测增强子。此外,被转录的候选增强子比仅通过组蛋白修饰预测的非转录的增强子更有可能在报告基因分析中被验证。
总的来说,这些发现表明,根据上述各自方法所定义的启动子和增强子具有一些共同的特征和能力,但在其他方面可能有所不同。这促进了对这些相似性的调控元件的批判性研究。
BOX3 通过DNA结合蛋白和DNA可接近性鉴定增强子
ChIP-chip和ChIP-seq技术已经成为鉴定候选增强子的核心技术,通过针对细胞或组织内已知的重要转录因子(TFs),或通过评估基因组内组蛋白修饰蛋白的富集程度(下图a),有时结合目标蛋白质参与增强子活性,例如P300-CBP组蛋白乙酰转移酶。其他评估DNA可接近性的方法,首先是通过DNaseI超敏性实验(DNase- seq),最近的研究中一般通过测定转座酶可接近性(ATAC- seq)作为预测增强子的手段(见下图b部分)。所有方法都是将得到的DNA测序reads比对到参考基因组上,mapping的密度可以用来评估结合的强度以及DNA可接近性(见下图c)。通过分析组蛋白修饰P300结合位点和一些之前被鉴定出的增强子发现:增强子出现在更高水平的H3K4me1,而不是H3K4me3 (图1)。基于这些模式可以建立计算模型来预测增强子的位置(和TSS)。随后提出的H3K27ac可以区分活性增强子和非活性增强子,其他组蛋白乙酰化与H3K27ac具有相似或特异性的预测效果。
(三)增强子和启动子的相似性
我们在此总结增强子和启动子的相似性,并考虑最近的基因组数据,得出增强子和启动子在整体染色质、序列和核心启动子结构方面出人意料地相似,这与之前对于两种调控元件的定义并不一致。当它们处于活跃状态时,两者都被NDR包围,被RNAPII结合,并在NDR边缘发散地启动转录。而编码蛋白质的基因启动子则产生正义链上的mRNA和反义链上的PROMPTs,增强子在两个方向上被转录为eRNAs。这意味着基因启动子和增强子共享相同的启动子架构,都有一双发散的TSS,每个都有自己的核心启动子(图1)。这种启动子架构似乎也共享对序列的偏好性:TSS往往有INR元件,对上游TATA盒有比较弱的偏好性,即使是在eRNA的TSS上。
哺乳动物基因启动子和增强子之间的一个显著差异是总体CG含量。大约50%的基因启动子与CpG岛重叠,而几乎没有增强子与CpG岛重叠(图1)。另一个明显的差异是相关的转录活性和RNA产生的数量。虽然许多候选增强子可以作为自发的启动子(体外),在HeLa细胞里,被激活的基因TSS比增强子产生的RNA多17倍。GM12878细胞中,TSS转录活性也比增强子多2-3倍(例如:利用精准核run-on测序实验PRO - seq)。因此,增强子启动较少的转录,而且产生的RNA经常在细胞核中被降解,从而进一步消耗最终RNA拷贝数。
然而,重要的是要考虑到这种平均差异是由高表达基因的TSSs驱动的,调控元件的整体染色质修饰和序列特征与其转录量密切相关。例如:高转录活性和/或广泛表达TSSs更有可能驻留在CG富集区域,侧翼更容易被H3K4me3修饰的组蛋白富集,这是两个用来区分增强子和启动子的关键特性(图1)。事实上,许多候选增强子也与H3K4me3相关联,考虑到转录活性的差异,各TSSs的染色质修饰水平高度相似(图2)。因此,大多数被用于区分增强子和启动子的特征,反映在转录活性和RNA输出产量上的差异。因此,经典的增强子/启动子组蛋白修饰并不是进行分类的最佳选择,因为它们可能只反映转录起始的能力(启动子活性),而不是增强远端转录的能力(增强子活性)。
早期尝试定义增强子和启动子,是假设它们具有不同的功能。如前所述,许多调控元件既具有增强子活性,也具有启动子活性,因为它们可以启动局部转录。最近的研究表明,基因的启动子可能也具有增强子活性,这使得情况变得更加复杂。例如:与体内增强子活性人类胚胎干细胞的POU5F1基因位点被注释的45个已经被鉴定的调控元件,其中有17个是启动子。在人类K562细胞中,20719个测试基因启动子在体外实验中,3%的启动子具有增强子活性。在体内实验中,小鼠胚胎干细胞中和长非编码RNA的12个启动子里,有5个具有增强子活性。此外,染色质构象数据表明,相当一部分与基因启动子相互作用的远端区域,实际上是其他基因的启动子,这就表明了增强活性实际上来源于基因的启动子。例如,INS基因启动子与SYT8基因启动子长距离相互作用,以调节其在人类胰岛中的表达,数百种启动子与启动子的相互作用可能介导了表达定量性状位点(expression quantitative trait loci, eQTLs)在人类原代血细胞中的作用。
因此,综上所述,先前指定为增强子的调控元件通常具有启动子活性,并且通常是根据与启动子活性相关的特征而不是与增强子活性相关的特征进行预测的,许多已知的基因启动子具有增强子活性。
(四)调控元件的新模型
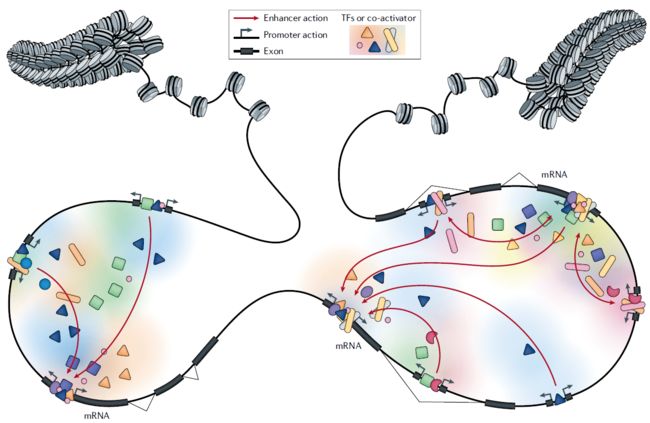
由于许多调控元件具有启动子和增强子的双重作用,我们和其他研究人员提出了一个模型,该模型假设启动子和增强子活性不是相互排斥的,而是假设调控元件可能具有不同程度的两种能力。在这个模型中,任何NDR能够在局部或远端影响转录起始的都被定义为“调控元件”。具体地说,根据上述基因组学技术的发现,调控元件具有以下特性:(1)首先,它集中在一个NDR中,通常被TFs结合。(2)第二,它有一定程度的启动子活性:招募RNAPII,在NDR边缘启动转录。(3)第三,它也可能具有一定程度的增强子活性:在其他调控元件上影响转录起始的能力(图3a)。
与之前的模型在概念上的不同是没有强制的增强子/启动子二分法:一个调控元件可以有任何结合或任何程度的启动子和/或增强子能力。该模型还解决了我们在概念和定义上存在的一些“历史问题”。首先,在文献中启动子基于从TSS任意长度(例如:-300到+ 100,或者-2000到+200),一个共同的假设是真正的启动子必须启动mRNA转录或其他有功能的/稳定的RNA。在我们提出的模型中,调控元件的长度基于DNA的可接近性,所产生的RNA类型及其稳定性与启动子活性的概念脱钩,启动子活性仅指局部启动转录的能力。后者的特性是有意义的,因为RNAPII转录起始在调控元件似乎遵循相同的规则,无论产生的RNA类型以及调控元件是否也有增强子功能。因此,一个调控元件具有强增强子活性,起始eRNA的转录也被认为具有一定程度的启动子活性。第二,在经典定义中,增强子必须能够增强远端转录起始,无论距离和方向。为什么一个调控元件能够在另一个位点增强转录起始,但只在合适的距离内并有特定方向的元件就不能被认为具有增强子活性?在上面的框架中,我们并没有施加这样的约束。
(五)测定调控潜能
如果我们能够测量调控元件的内在增强子和启动子的强度,然后推断是什么驱动了各自的潜能,那么上面提出的模型框架只是概念性上的。从概念上说,有两种主要的、互补的实验来回答这些问题:基于CRISPR的体内方法(BOX4)和大规模报告基因分析(MPRAs)(BOX5)。
基于CRISPR的体内基因组编辑方法(Box 4)允许直接编辑、诱变和删除具有增强子和/或启动子活性的调控元件(CRISPR - cas9),或间接干扰(CRISPRi)或激活(CRISPRa)这些元件,然后测量一个或多个感兴趣区域的RNA输出。这类方法的主要优势在于,它们考虑了基因组的复杂性,包括局部染色质背景和多个调控元件之间的相互作用,因为是在其内源性基因组背景下靶向位点的。有两个主要的缺点:首先,在一个更大的区域内,很难评估独立的调控元件的因果关系以及作用。例如,两个具有增强子活性的调控元件可能是冗余的,其阿红一个可以补偿另一个的缺失。其次,很难对一个基因或基因簇周围的一个或几个区域进行详细的筛查。这意味着,尽管这些方法可以为选定的基因组区域提供更多的信息,但要从这些数据中建立通用的、定量的和预测模型仍是一项挑战。
BOX4 体内基于CRISPR的方法调控活性
基于CRISPR系统的酶靶技术的最新发展(例如:Cas9核酸酶)通过single guide RNA (sgRNAs)来切割特定的DNA链,从而更精确的干扰使调控元件。特别是CRISPR-Cas9已被用于系统地删除基因座内的区域(见下图a部分)。在这些研究中,sgRNAs库被设计用于针对基因组区域进行删除。在给定的细胞中,库中的一对sgRNA的表达被用来删除一段区域(图中红色阴影区域)。再评估敲除该区域对近端基因活性的影响。在此系统的基础上,发展了变异分析方法:CRISPR干扰(CRISPRi)利用sgRNAs靶向核酸酶缺陷的Cas9 (dCas9),与KRAB和SID一起干扰调控活性(下图b部分)。dCas9系统还可以被用来激活调控元件,称为CRISPR激活(CRISPRa),通过dCas9与转录激活因子(例如VP64)融合(下图c部分)。CRISPR工具的通用性为研究转录调控提供了很大的希望。
MPRAs (Box 5)试图将单个序列的启动子和/或增强子潜能与它们自身基因组背景提供的调控潜能脱钩。从本质上说,这些方法建立在经典的增强子和启动子报告基因分析的基础上,但通过barcode集成和高通量测序的结合,可以一次性测定数千个候选元件。除了大批量之外,基于质粒的MPRA方法的主要优势也是相对于体内基因组编辑方法的劣势:如前所述,它们可以评估调控元件与自身背景(包括局部染色质状态)解耦的增强子/启动子潜力。因此,有了这些数据,就可以建立模型,推测驱动它们的启动子或增强子潜力。因此,在本综述中,我们主要关注MPRA特征,尽管,正如我们下面指出的,体内基因组编辑方法和MPRAs是互补的,这些方法/数据应该结合起来。
BOX5 高通量平行报告基因分析测定增强子和启动子的潜能
大规模并行报告基因分析(MPRAs)采用了经典的reporter分析的载体设计,候选调控序列被放在报告基因载体上(BOX1)。在测试增强子的潜力的时候,候选序列被放置在指定最小启动子的上游或下游(下图a部分),启动子测试是通过将候选区域直接放置在报告基因前面(下图b部分),这可以在没有增强子或者有增强子的情况下进行测定。大规模的经典的reporter分析,是通过高通量测序和量化的barcodes(下图c和d)。这些条形码通常被整合到报告基因UTR 5′和3′端非翻译区,进行批量测序。因此,reporter的RNA水平作为输出,而不是产生报告蛋白的数量。虽然不同MPRA策略已经被开发出来,许多方法依赖于游离质粒,并且仅限于只能测试短序列(<200 bp)。此外,MPRA读出可以被测试之外的其他调控元件影响,如minimal启动子结合候选增强子序列结合,这表明一些增强子和和核心启动子兼容性的限制。尽管如此,大多数增强子MPRA研究用相同的minimal启动子测试所有候选增强子序列。最后,基于质粒的MPRAs是有限的,因为他们不能决定染色质的背景,染色质意识的调控元件的潜力。为此,人们开发了慢病毒MPRAs,以测试其在天然染色质背景下的大量候选调控元件。
(六)调控潜力的决定因素
基于MPRA的方法产生了一些新的见解。特别是,它已经有可能获得第一个序列特征驱动的强启动子和/或增强子视图。MPRA方法也使研究这些活性之间的关系相关性。例如,调控元件是否可能同时具有强增强子活性和强启动子活性?是否所有有增强子活性的区域都有一些启动子活性?在这里,我们回顾了旨在回答这些问题的研究(图3b)。
Nguyen等人利用启动子和增强子MPRAs测试候选增强子H3K27ac富集的基因相关启动子,发现CREBBP协同激活因子的结合对小鼠皮层神经元的激活作用。有趣的是,MPRA的readout显示了增强子和启动子的正相关性活性。然而,大多数候选增强子只有有限的启动子活性,被测基因启动子倾向于具有强启动子活性,但令人惊讶的是,启动子里增强子的活性与被测试的候选增强子相似。这些结果表明,调控元件在本质上具有增强子和启动子两种活性,但其中一些具有特异编码显著增强启动子活性的特性。
这些发现在使用MPRAs的研究中得到了印证,这些研究旨在鉴定启动子活性的特征(图3b右)。首先,CpG整体含量与启动子呈正相关,而与增强子活性无关。然而,并不是CpG二核苷酸本身导致了更高的启动子活性,而是有利于转录因子结合位点的CpG序列。事实上,已经有研究表明,一个TF结合位点或其他序列模式(例如ChIP-seq)与启动子活性有关。由于TFs在细胞之间是不同的,这些结果至少在某种程度上是依赖于特定于环境的,考虑这一点很重要。Weingarten- Gabbay等人使用MPRAs发现,强的启动子活性与核心启动子元件高度相关,特别是具有典型间距和方向的TATA盒和INR位点。特定TFs的位点预测——例如,SP1、ETS、CREB和RFX——也被发现与高启动子活性相关。有趣的是,只有一些TFs:例如ETS和SP1具有附加效应,因此一个区域内预测的结合位点数量与启动子活性相关。最重要的是,高启动子活性与几个TFs结合位点的重合有关,特别是广泛表达的TFs。这反过来又与DNA可接近性相关:高可接近性区域内的调控元件通常具有高启动子活动,可接近性的程度与结合的TFs的表达水平相关。事实上,核小体排斥序列的引入导致总体上更高的启动子活性。
突变相关的增强子的活性经常与转录旖旎的结合位点有关,并且具有细胞特异性。说明了TFs的不仅在启动子活性,也在增强子活性上的重要性。与启动子活性一样,某些TFs结合位点序列的存在、它们的组合以及这些位点的数量与增强子强度有关。然而,和与高启动子活性相关的TFs不同,同样具有高增强子活性的mRNA基因启动子富集了倾向于低CG含量的TFs的结合位点(图3b左)。此外,MPRA研究评估了核心启动子对不同的共激活子的反应,表明某些共激活子和核心启动子之间的相容性受限是由于它们的CG含量。因此,除了以上讨论的核心启动子强度和结合位点复杂性,增强子和启动子潜能的差异可能受到TFs或TF招募的共激活子与调控元件结合方式的影响。事实上,AP1和NFY的结合偏向于具有增强子活性的调控元件,而其他TFs的结合——例如CREB——偏向于具有启动子活性的调控元件。由于高CpG含量区域通常是核小体排斥的,因此可能需要将某些倾向于低CpG序列的TFs(如AP1)与CpG含量低的调控元件结合,以调节染色质的可接近性。这些可能包括所谓的pioneer转录因子,能够结合核小体DNA,招募染色质重组因子。事实上,候选增强子的活性与SWI/SNF (BAF)染色质重塑复合体的适当募集有关,并且破坏TF结合位点和影响染色质可接近性的遗传变异在候选增强子中高度富集。
因此,如果考虑CpG含量的差异,MPRA推断的增强子活性的调控元件在很大程度上与低启动子活性的调控元件相似。这表明编码增强子活性的序列特征也泛化为(低)启动子活性,但反过来却是不一定的。在果蝇中有一个有趣的平行实验,结果显示调控元件的增强子的强度依赖于次优的TF结合位点序列,而优化TF结合亲和度会导致启动子活性的增加和增强子活性的丧失。但也有某些例外。例如,具有高增强子活性的注释的mRNA基因启动子被高表达,但究竟是什么决定了如此强的二象性还有待确定。除了少数有偏好的TFs外,驱动增强子活性的区别特征普遍缺乏,这就解释了为什么与基因启动子相比,具有高增强子活性和弱启动子活性的已知区域具有更高的进化周转率。高启动子活性可能需要对某些特性进行选择,而增强子活性的限制可能较低。另外,这种模式可能意味着有更多不同的方式来编码增强子活性,而这些方式可能更依赖于特定的细胞类型或实验背景。
(七)模型揭示增强子/启动子的二重性
考虑到调控元件的增强子和启动子的潜力的普遍性,值得推测这种二重性的来源和背后的原因。一些模型已经提出目前的eRNAs的功能作用或增强子转录行为,从而提供了一个具有启动子活性的调控元件同时具有高增强子活性的原因。由于许多eRNAs转录水平低,易被降解,并且转录自具有高进化周转率的DNA,关于eRNA功能依赖于高拷贝数和/或序列特异性相互作用的假设在大多数情况下不太可能是对的。更有可能是基于行为而不是转录产物的功能,因为它们不受上述约束的影响。例如:发散性转录可能引起负超螺旋,从而影响染色质结构和调控元件的可接近性,或者它可能允许通过RNAPII羧基终端区域相关的乙酰转移酶在核小体侧面进行组蛋白修饰,从而阻止抑制性mark的修饰。或者,发散性转录可能只是一种从NDR取代RNAPII的方法。另一种观点认为,具有增强子活性的调控元件的转录可能仅仅是高RNAPII浓度集中的可接近染色质处的RNAPII的“虚假”转录所引起的噪音。然而,具有增强子潜能的候选区域通常是可接近的,但不转录。
或者,RNAPII(启动子活性)的招募和启动可能会导致增强子活性。在这个模型中,有一个相似之处通过转录调节的hub和TF-RNAPII液体冷凝模型,在三维空间中关联的调节元素可能通过同型吸引协同工作,以增加或维持TFs、GTFs、共激活因子和RNAPII的局部浓度,这是暂时性的在物理上接近的元件之间绑定、接合和重新定位(这段不太清楚怎么翻译,建议看原文体会)。根据这一观点,多个调控元件在三维空间上相互接近,增加了TF和RNAPII招募到靶基因TSSs的可能性(图4)。这种以TF-和RNAPII-为中心的调控元件间协作模型的一个特征是:TF结合的潜力或应答能力的差异,解释了特定调控元件之间的增强子或协同激活因子的不相容性。此外,调控元件在招募转录结构时的竞争可以解释为什么某些元件具有更强的增强子潜力和更弱的启动子潜力,或者相反,但为什么某些TFs允许同时具有强增强子和启动子活性还有待研究。
正如我们上面讨论的,鉴于观察到的大多数具有增强子活性的调控元件的高进化更替,增强子的潜力不大可能作为一种在大多数情况下被选择的通用能力,即使这些元件是高度保守的。相反,在这篇综述中讨论的结果指向了一个模型,在这个模型中增强子潜力(本质上是低启动子潜力)是通过转录因子和其他转录结构的结合位点直接编码的。高启动子潜能需要额外的限制,并且是在基础的增强子潜能之上获得的,这可能会或者不会导致增强子潜能的降低。我们注意到在该模型中,增强子活性和低启动子活性是被激活的调控元件的基本状态(图3a)。转座子元件最近被认为是调控的重要来源,其内在增强子潜力和弱启动子活性可能是进一步改变的基础。这样,通过改变NDR相关和下游DNA序列,具有增强子活性的调控元件可能被重新利用,使其具有更强的启动子活性,并产生更长的、更高丰度的RNA。
(八)结论
虽然转录调控是生物学中研究最多的课题之一,但只有现在我们才开始系统地解释调控元件的内在机制。这一机会是由于高通量方法的发展和应用,在体外和体内评估调控活性。我们在此列出在这项工作中面临的一些挑战和机遇。
近年来,许多关于转录调控的研究都是描述性的。例如,专注于与增强子活性相关的基因组模式,并使用这些模式在全基因组范围内预测细胞或组织里候选活性增强子的“图谱”。虽然这项研究是值得称赞也非常有用,但这些方法有明显的风险。重要的是,在脊椎动物中具有强增强子的特征良好的调控元件的数量很少,这些可能不是最具代表性的具有增强子的调控元件。因此,特征的模式(染色质修饰,eRNA产生等)用于预测具有增强子能力的调控元件是基于非常小的训练集的基础。这就导致了一种情况,候选增强子被一种类型或一组数据预测(例如,ChIP-seq),然后通过其他数据被预测。这就带来了确定偏差、过拟合、过度简化的风险。
基于MPRA和CRISPR的基因组区域启动子和增强子潜力大量筛选方法的发展,包括质粒或在基因组内,有很大的希望增加我们对潜在的调控元件与明确的增强子或启动子活性的理解,以减少认知偏差。结合基因组学或转录组方法,和基于MPRA和/或CRISPR的筛选可以为建立增强子和启动子活性的预测和定量模型开辟新的途径。
找到连接MPRA方法和基于CRISPR的方法的桥梁也很重要,这样MPRAs所测量的调控元件中内在的增强子和启动子潜力可以与它们周围的调控元件和染色质背景互补,反之亦然。从而充分利用每种方法的优点。单个研究小组不太可能解决这些挑战中的任何一个,但需要侧重于筛查研究方法之间的实质性结合(MPRA和/或体内基因组编辑技术)基因组学或转录组方法和统计模型。换句话说,我们认为这是机器学习、生物物理学、基因组学和生物信息学交叉的关键领域。
以下是一些有待解决的关键问题。首先,为什么某些TFs可以驱动启动子而不能驱动增强子活性,为什么只有一些TFs具有附加效应?第二,在给定的细胞类型中,是哪些TFs(或辅助激活因子)和结合位点的组合可以驱动启动子和/或增强子的活性?第三,增强子的活性似乎被序列编码得更加多样化,这表明调控元件获得增强子活性的方式多种多样。但是在一个给定的系统中这些不同的方式是什么,信号之间的相互作用是什么?第四,强基因启动子同时具有强增强子活性的潜在机制是什么?最后,调控元件的相容性背后的机制是什么(也就是说,一个调控元件可能增强某些具有启动子活性的调控元件的转录,但不是全部)?
虽然我们主要集中讨论了TF -和序列相关特征决定调控元件的激活(增强子和启动子)功,基因的转录活性还取决于其他调控元件和决定因素:包括染色质重塑,近端沉默子和绝缘子的活性,阻遏蛋白的结合,周围染色质三维拓扑结构的折叠,以及调控元件对外部的反应。这些因素中很多都是相关的:例如,CpG富集的调控元件较少依赖染色质重构事件。尽管如此,分析并整合大规模定量测定增强子和启动子潜力是非常重要的。在我们上面提出的模型中,只考虑了增强子和启动子的能力,但同样的模型可以扩展,如局部或远端抑制活性。例如,一个对基因启动子具有增强子活性的调控元件可能在某些情况下作为其他启动子的沉默子或绝缘子。
尽管面临巨大的挑战,我们现在处于一个非常有前途和令人兴奋的研究转录调控的时期。我们目前的知识,加上上述概述的方法,有希望让我们最终理解更多的转录调控背后的大部分机制。这将在生物研究、医学和生物技术方面具有深远的意义。换句话说,我们可能正处于从描述和分类调控现象的转录调控研究领域向建立基因调控的定量生物学模型转变的过渡期。