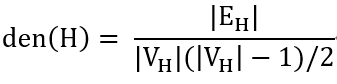前情回顾:
Gephi网络图极简教程
Network在单细胞转录组数据分析中的应用
网络数据统计分析笔记|| 为什么研究网络
网络数据统计分析笔记|| 操作网络数据
网络数据统计分析笔记|| 网络数据可视化
前面的几节的内容基本是在讲如何构建一个图,接下来我们看看拿到图之后可以做哪些分析。当我们提到图时,脑海中反应的是一张网。要仔细描述清楚网的特点就需要一些术语,就像我们描述一组数据会用到平均值和标准差一样。网络数据的描述分析的目的是把图讲清楚,这就需要一套统计量。
网络数据的描述分析过程是网络的特征化过程。
- 节点和边的特征
- 网络的凝聚性度量
- 社团发现
- 同配性与混合
节点和边的特征
网络图中最基本的元素是节点和边。首先我们需要的就是对他们的度量。我们之前介绍过节点度(degree)的概念:节点链接边的数量。节点的强度(strength)是指于该节点所连边的权重之和。
library(sand)
data(karate)
degree(karate)
Mr Hi Actor 2 Actor 3 Actor 4 Actor 5 Actor 6 Actor 7 Actor 8 Actor 9 Actor 10 Actor 11
16 9 10 6 3 4 4 4 5 2 3
Actor 12 Actor 13 Actor 14 Actor 15 Actor 16 Actor 17 Actor 18 Actor 19 Actor 20 Actor 21 Actor 22
1 2 5 2 2 2 2 2 3 2 2
Actor 23 Actor 24 Actor 25 Actor 26 Actor 27 Actor 28 Actor 29 Actor 30 Actor 31 Actor 32 Actor 33
2 5 3 3 2 4 3 4 4 6 12
John A
17
strength(karate)
Mr Hi Actor 2 Actor 3 Actor 4 Actor 5 Actor 6 Actor 7 Actor 8 Actor 9 Actor 10 Actor 11
42 29 33 18 8 14 13 13 17 3 8
Actor 12 Actor 13 Actor 14 Actor 15 Actor 16 Actor 17 Actor 18 Actor 19 Actor 20 Actor 21 Actor 22
3 4 17 5 7 6 3 3 5 4 4
Actor 23 Actor 24 Actor 25 Actor 26 Actor 27 Actor 28 Actor 29 Actor 30 Actor 31 Actor 32 Actor 33
5 21 7 14 6 13 6 13 11 21 38
John A
48
hist(degree(karate), col="lightblue", xlim=c(0,50),
xlab="Vertex Degree", ylab="Frequency", main="",breaks = 50)
# CHUNK 2
hist(strength(karate), col="pink",
xlab="Vertex Strength", ylab="Frequency", main="")
不同的数据集有不同的度分布情况,如酵母蛋白的互作网络:
library(igraphdata)
data(yeast)
yeast
IGRAPH 65c41bb UN-- 2617 11855 -- Yeast protein interactions, von Mering et al.
+ attr: name (g/c), Citation (g/c), Author (g/c), URL (g/c), Classes (g/x), name (v/c), Class
| (v/c), Description (v/c), Confidence (e/c)
+ edges from 65c41bb (vertex names):
[1] YLR197W--YDL014W YOR039W--YOR061W YDR473C--YPR178W YOR332W--YLR447C YER090W--YKL211C YDR394W--YGR232W
[7] YER021W--YPR108W YPR029C--YKL135C YIL106W--YGR092W YKL166C--YIL033C YGL026C--YKL211C YOR061W--YGL019W
[13] YGL115W--YER027C YGL049C--YGR162W YDR394W--YOR117W YDL140C--YML010W YLR291C--YKR026C YGR158C--YDL111C
[19] YDR328C--YDL132W YOL094C--YNL290W YDR460W--YPR025C YBR154C--YOR341W YBR154C--YOR116C YIL062C--YKL013C
[25] YBR154C--YOR207C YBR154C--YPR010C YER027C--YDR477W YLR291C--YGR083C YPL093W--YDR496C YER006W--YMR049C
[31] YER006W--YMR290C YFR052W--YHR200W YOR261C--YFR004W YHR052W--YDR496C YDL140C--YBR154C YDR394W--YOR259C
[37] YDR280W--YGR195W YOR260W--YDR211W YMR193W--YML009C YGR162W--YOL139C YPR187W--YPR110C YPL093W--YDR101C
+ ... omitted several edges
ecount(yeast) #边数量
[1] 11855
vcount(yeast) # 节点数
[1] 2617
d.yeast <- degree(yeast)
head(d.yeast)
YLR197W YOR039W YDR473C YOR332W YER090W YDR394W
40 19 9 13 21 37
hist(d.yeast,col="blue",
xlab="Degree", ylab="Frequency",
main="Degree Distribution")
考虑到分布递减的趋势,双对数坐标在表达度的信息时更为有效。
dd.yeast <- degree_distribution(yeast)
head(dd.yeast)
[1] 0.00000000 0.26518915 0.12877340 0.09247230 0.06763470 0.05502484
d <- 1:max(d.yeast)-1
ind <- (dd.yeast != 0)
plot(d[ind], dd.yeast[ind], log="xy", col="blue",
xlab=c("Log-Degree"), ylab=c("Log-Intensity"),
main="Log-Log Degree Distribution")
可以计算下降的速率,来反映度指数(degree exponent)。
衡量节点以何种方式彼此连接的方式,用节点邻居的平均度(Average nearest neighbor degree)。
a.nn.deg.yeast <- knn(yeast,V(yeast))$knn
plot(d.yeast, a.nn.deg.yeast, log="xy",
col="goldenrod", xlab=c("Log Vertex Degree"),
ylab=c("Log Average Neighbor Degree"))
节点中心性
关于图节点的问题很多,本质上都是在试图理解他在网络中的重要性。度量中心性(centrality)正是为了量化重要性。节点中心性度量有:节点度中心性、接近中心性、介数中心性以及特征向量中心性
节点度中心性(Degree centrality)
在一个网络图G=(V,E)中,节点v的度指的是与v相连的E中边的数量。接近中心性(closeness centrality)
接近中心性主要用于计算每个顶点到其他所有顶点的最短距离之和。然后将得到的和反过来确定该节点的接近中心性得分。原生的接近中心性计算方式如下:
- 介数中心性(betweenness centrality)
度量试图概括的是某个节点在多大程度上“介于”(between)其他节点对之间。节点的“重要性”与其再网络路径中的位置有关。
- 特征向量中心性(eigenvector centrality)
特征向量中心性的基本思想是,一个节点的中心性是相邻节点中心性的函数。也就是说,与你连接的人越重要,你也就越重要。
特征向量中心性和点度中心性不同,一个点度中心性高即拥有很多连接的节点特征向量中心性不一定高,因为所有的连接者有可能特征向量中心性很低。同理,特征向量中心性高并不意味着它的点度中心性高,它拥有很少但很重要的连接者也可以拥有高特征向量中心性。
简而言之:
点度中心性:一个人的社会关系越多,他/她就越重要
中介中心性:如果一个成员处于其他成员的多条最短路径上,那么该成员就是核心成员
接近中心性:一个人跟所有其他成员的距离越近,他/她就越重要
特征向量中心性:与你连接的人社会关系越多,你就越重要
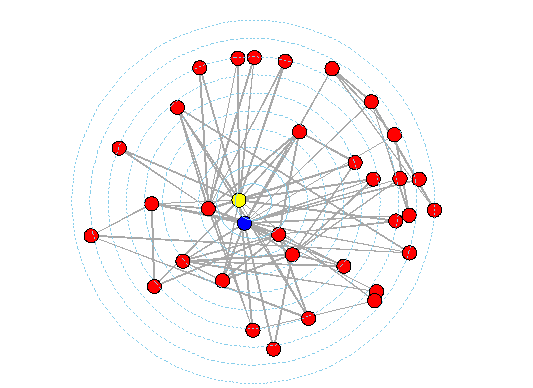
展示节点中心性的一种直观方式是使用辐射布局。空手道俱乐部网络中心性描述,可以看出主管和教练处于中心位置。
A <- as_adjacency_matrix(karate, sparse=FALSE)
library(network)
g <- network::as.network.matrix(A)
library(sna)
sna::gplot.target(g, degree(g,gmode="graph"),
main="Degree", circ.lab = FALSE,
circ.col="skyblue", usearrows = FALSE,
vertex.col=c("blue", rep("red", 32), "yellow"),
edge.col="darkgray")
library(CINNA)
visualize_graph( karate, centrality.type="Markov Centrality")
CINNA是一个提交到CRAN资源库的R包,是为网络科学中的中心性分析而编写的。该方法可用于多种中心性测度的组合、比较、评价和可视化。
proper_centralities(karate)
[1] "subgraph centrality scores" "Topological Coefficient"
[3] "Average Distance" "Barycenter Centrality"
[5] "BottleNeck Centrality" "Centroid value"
[7] "Closeness Centrality (Freeman)" "ClusterRank"
[9] "Decay Centrality" "Degree Centrality"
[11] "Diffusion Degree" "DMNC - Density of Maximum Neighborhood Component"
[13] "Eccentricity Centrality" "Harary Centrality"
[15] "eigenvector centralities" "K-core Decomposition"
[17] "Geodesic K-Path Centrality" "Katz Centrality (Katz Status Index)"
[19] "Kleinberg's authority centrality scores" "Kleinberg's hub centrality scores"
[21] "clustering coefficient" "Lin Centrality"
[23] "Lobby Index (Centrality)" "Markov Centrality"
[25] "Radiality Centrality" "Shortest-Paths Betweenness Centrality"
[27] "Current-Flow Closeness Centrality" "Closeness centrality (Latora)"
[29] "Communicability Betweenness Centrality" "Community Centrality"
[31] "Cross-Clique Connectivity" "Entropy Centrality"
[33] "EPC - Edge Percolated Component" "Laplacian Centrality"
[35] "Leverage Centrality" "MNC - Maximum Neighborhood Component"
[37] "Hubbell Index" "Semi Local Centrality"
[39] "Closeness Vitality" "Residual Closeness Centrality"
[41] "Stress Centrality" "Load Centrality"
[43] "Flow Betweenness Centrality" "Information Centrality"
[45] "Dangalchev Closeness Centrality" "Group Centrality"
[47] "Harmonic Centrality" "Local Bridging Centrality"
[49] "Wiener Index Centrality" "Weighted Vertex Degree"
sna::gplot.target(g, closeness(g,gmode="graph"),
main="Degree", circ.lab = FALSE,
circ.col="skyblue", usearrows = FALSE,
vertex.col=c("blue", rep("red", 32), "yellow"), # 颜色与节点对应
edge.col="darkgray")
sna::gplot.target(g, betweenness(g,gmode="graph"),
main="Degree", circ.lab = FALSE,
circ.col="skyblue", usearrows = FALSE,
vertex.col=c("blue", rep("red", 32), "yellow"),
edge.col="darkgray")
sna::gplot.target(g, evcent(g),
main="Degree", circ.lab = FALSE,
circ.col="skyblue", usearrows = FALSE,
vertex.col=c("blue", rep("red", 32), "yellow"),
edge.col="darkgray")
在有向图中,节点的中心性用枢纽hub与权威authority来描述。有名的算法是HIST。1999年,Jon Kleinberg 提出了HITS算法。作为几乎是与PageRank同一时期被提出的算法,HITS同样以更精确的搜索为目的,并到今天仍然是一个优秀的算法。
HITS算法的全称是Hyperlink-Induced Topic Search。在HITS算法中,每个节点被赋予两个属性:hub属性和authority属性。同时,节点被分为两种:hub节点和authority节点。hub,中心的意思,所以hub节点指那些包含了很多指向authority节点的链接的节点。
# CHUNK 10
par(mfrow = c(1,2))
l <- layout_with_kk(aidsblog)
plot(aidsblog, layout=l, main="Hubs", vertex.label="",
vertex.size=10 * sqrt(hub_score(aidsblog)$vector))
plot(aidsblog, layout=l, main="Authorities",
vertex.label="", vertex.size=10 *
sqrt(authority_score(aidsblog)$vector))
边的特征
同样地,我们会问在网络中哪条边是比较重要的呢?边也可以用中心性的指标进行度量。
> eb <- edge_betweenness(karate)
> E(karate)[order(eb, decreasing=T)[1:3]]
+ 3/78 edges from 4b458a1 (vertex names):
[1] Actor 20--John A
Mr Hi --Actor 20
Mr Hi --Actor 32
网络图的凝聚性特征
网络图的凝聚性,是为了描述这样一种性质,即网络图中节点的子集与相应的边以何种程度凝聚在一起。在网络工作中,许多问题归结为涉及网络工作内聚性的问题,即顶点的子集在多大程度上内聚(形象来说,就是粘在一起)。在社交网络中,你的朋友的朋友是否彼此之间也会成为朋友呢?细胞内的蛋白质集合是如何紧密结合在一起的?万维网的页面结构是否倾向于区分不同类型的内容?一个互联网拓扑结构的那一部分似乎构成了’主干’。
“凝聚性”节点子集一般指这样的节点集合:
(1)内部联系紧密,
(2)与其它节点相对分离。
更正式地说,图分割算法通常试图寻找图G=(V,E)中节点集V的一个分割ℒ={C1,…,CK},使得连接Ck与Ck’之中节点的边集合E(Ck,Ck’)规模相对较小,而连接Ck内部节点的边集合E(Ck)=E(Ck,Ck)规模较大。
分割后的产生的各部分子图也常被称为“模块”(Module),各模块也常视为一种“子图”类型用于描述问题。
描述网络凝聚性的一种方法是规定某种感兴趣的子图类型,一个典型的例子是团。团是一类完全子图,集合内所有节点都由边相互连接,因而是完全凝聚的节点子集。所有尺寸的团的普查(census)可以提供一个“快照”,帮助了解图的结构。不过经常存在冗余问题,即大尺寸的团包含了小尺寸的团。“极大团”(maximal clique)是不被任何更大的团包含的一类图团。实际上,大尺寸的团很稀少,团的存在要求图G本身相当稠密,但现实世界的网络多是稀疏的。
团的概念存在各种弱化了条件的版本。例如,图G的k核(k-core)是一个图G的子图,其中所有节点的度至少为k,且不被包含于满足条件的其它子图中(即它是满足条件的最大的子图)。核的概念在可视化中非常流行,因为他提供了一种将网络分解到类似洋葱的不同“层”(layer)的方法。这种分解可以与辐射布局有效地结合起来。
在团及其变体之外,有一些其他类型子图可以用于定义网络凝聚性。二元组(dyad)和三元组(triad)是两个基本的量(Davis and Leinhardt, 1967; Holland and Leinhardt, 1970)。二元组关注两个节点,它们在有向图中有三种可能的状态:空(null,不存在边)、非对称(asymmetric,存在一条有向边)、双向(mutual,两条有向边)。类似地,三元组是三个节点,共有16种可能的状态(图下所示)。对图G中每个状态观察到的次数进行统计,得到的是这两类子图可能状态的一个普查,可以帮助深入理解图中连接的本质。
par(mfrow = c(1,2))
table(sapply(cliques(karate), length)) # 团的普查
1 2 3 4 5
34 78 45 11 2
##子图与普查
#所有尺寸的团的普查可以提供一个快照,将显示各尺寸的团的数量
census <- table(sapply(cliques(karate), length))
census
plot(census)
cliques(karate)[sapply(cliques(karate), length) == 5]
[[1]]
+ 5/34 vertices, named, from 4b458a1:
[1] Mr Hi Actor 2 Actor 3 Actor 4 Actor 14
[[2]]
+ 5/34 vertices, named, from 4b458a1:
[1] Mr Hi Actor 2 Actor 3 Actor 4 Actor 8
clique_num(yeast) # 团的数量
[1] 23
cores <- coreness(karate) # 核
sna::gplot.target(g, cores, circ.lab = FALSE,
circ.col="skyblue", usearrows = FALSE,
vertex.col=cores, edge.col="darkgray")
# CHUNK 17
aidsblog <- simplify(aidsblog)
dyad_census(aidsblog)
$mut
[1] 3
$asym
[1] 177
$null
[1] 10405
密度与相对密度
一个图的“密度”(density)是指实际出现的边与可能的边的频数之比,反映了网络的凝聚性特征。例如,对于不存在自环和多重边的(无向)图G,子图H=(VH,EH)的密度为:
den(H)的值处于0到1之间,提供了一种H与团的接近程度的度量。由于子图H可以自由选择,这使简单的密度概念变得很有趣。例如,令H=G,得到的是整个图G的密度;而令H=Hv为节点v∈V的邻居集合以及节点间的边,度量的是v直接相邻邻居的密度。
detach("package:sna")
detach("package:network")
ego.instr <- induced_subgraph(karate,
neighborhood(karate, 1, 1)[[1]])
ego.admin <- induced_subgraph(karate,
neighborhood(karate, 1, 34)[[1]])
edge_density(karate)
0.1390374
edge_density(ego.instr)
[1] 0.25
edge_density(ego.admin)
[1] 0.2091503
相对频率也可以用于定义图中的“聚集性”(clustering)概念,反映了网络的凝聚性特征。例如,术语“聚类系数”(clustering coefficient)的标准定义如下:
其中,τ∆ (G)指的是图G中三角形个数(三角形指尺寸为3的团),而τ3 (G)是连通的三元组(即由两条边连接的三个节点,有时也称为2-星,2-star)个数。clT (G)的值也被称为图的“传递性”(transitivity)。表示“传递性三元组的比例”。
注意,clT (G)是对全局聚集性的度量,所概括的是联通三元组闭合形成三角形的相对频率。
transitivity(karate)
[1] 0.2556818
transitivity(karate, "local", vids=c(1,34))
[1] 0.1500000 0.1102941
有向图中独有的一个概念是“互惠性”(reciprocity),即有向网络中的边多大程度上是互惠的。计算相对频率的单位可以是二元组或者有向边,对应有两种表示这一概念的方法。当采用二元组作为基本单位,互惠性被定义为有互惠(双向)有向边的二元组数量,除以只有单一非互惠的二元组数量。另一种情况下,互惠性定义为互惠边的总数除以所有边的数量。
reciprocity(aidsblog, mode="default")
# ---
## [1] 0.03278689
# ---
reciprocity(aidsblog, mode="ratio")
# ---
## [1] 0.01666667
# ---
连通性与图分割与图流
若图G中存在从节点u到另一个节点v的一条通路,则可以称节点v从u是“可达的”(reachable)。若所有节点从任一其他节点均可达,则称图G为“连通的”(connected)。图的“组件”(component)是一个最大化的连通子图,即它是图G的一个连通子图,且任意增加V中的一个剩余节点时都会破坏连通性。
is_connected(yeast)
# ---
## [1] FALSE
# ---
普查寻找组件
comps <- decompose(yeast)
table(sapply(comps, vcount))
2 3 4 5 6 7 2375
63 13 5 6 1 3 1
其中组件1 大约占到90%的节点,一般现实组件都发现了小世界的特性。小世界(small world)网络模型将晶格模型的可传递性与随机网络模型的低路径长度结合在一起,其由一个网络结构的图出发,对一小部分边进行随机重连(rewiring)(Watts and Strogatz, 1998)。具体而言,从排成一圈的Nv个节点的集合开始,将每个节点与其一侧的r个邻居进行连接;之后以概率p移除每条边的一个端点,与另一个等概率选取的新节点相连,并避免生成自环与多重边。
小世界一般有以下两个特性:
- 节点间最短路径的长度同长较小
- 网络的聚集性很高
yeast.gc <- decompose(yeast)[[1]]
# CHUNK 25
mean_distance(yeast.gc)
[1] 5.09597
diameter(yeast.gc)
# ---
## [1] 15
# ---
transitivity(yeast.gc) # 这意味着大约50% 的连通三元形成了三角形。
[1] 0.4686663
与组件概念相比,一个更好的连通性定义源于以下问题:如果从图中任意移除包括k个节点(或边)的子集,剩余的子图是否还是连通的?这种思路可以使用节点和边连接通度的概念,以及节点和边的割等相关概念进行精确定义。若图G满足(1)节点数Nv>k,(2)移除基数|X| 如果移除图中的某个节点(边)的集合破坏了图的连通,这个集合称为“点割集(边割集)”(vertex-cut,edge-cut)。能破坏图连通性的单个节点称为“割点”(cut vertex),有时也称为“关节点”(articulation point)。 在有向图中的扩展 图分割(graph partitioning)问题在复杂网络方面的文献中也常被称为社团发现(community detection)问题。模块度(modularity)是社团发现中用来衡量社团划分质量的一种方法,一个相对好的结果在社团(community)内部的节点连接度较高,而在社团外部节点的连接度较低。 分割(partitioning)泛指将元素的集合划分到“自然的”子集之中的过程。更正式地,一个有限集S的分割ℒ={C1,…,CK}是将S分割为K个不相交的非空子集Ck,满足UKk=1Ck=S。在网络图的分析中,分割是一种无监督的方法,用于发现具有“凝聚性”的节点子集,揭示潜在的关系模式。开发社团发现的算法一直是研究的热点,算法有很多,如层次聚类、谱分割等,更多的领域综述可参考Fortunato等的文章(Fortunato and Lancichinetti, 2009)。 层次聚类有两个方向,由小到大的凝聚,由大到小的分割,具体可以参考文章数量生态学笔记||层 贪婪算法的凝聚聚类,Community structure via greedy optimization of modularity。 分群后的标签颜色与分组 谱聚类算法由于其算法流程简单、计算简洁与 Kmeans 算法相比不容易陷入局部最优解,能够对高维度、非常规分布的数据进行聚类。谱聚类算法是利用图谱理论来进行算法分析,思想是把数据分析问题看成是图的最优分割问题,将数据样本看成是各个数据点,然后将数据点描绘成一个图表,根据图表关系计算出相应的相似矩阵,找到一种最优分割方法计算出相似矩阵的最小特征向量,最后利用相应算法得出最后的聚类结果。 谱聚类算法是将样本点看成为一个个顶点,将顶点之间用带权的边连接起来,带权的边可以看成是顶点之间的相似度。聚类从而可以看成如何分割这些带权的边,继而将聚类问题转化为怎么进行图分割的问题,但是如果这样的话新的问题又产生了,那就是怎样找到一种最优方法来划分这个图,才能使同组之间的样本权重尽可能高,不同组的权重尽可能的低。权重太低的边是多余的就要舍去,常用保留边的方法是要建立最邻近图谱,在最邻近图谱中每个顶点只与K 个相似度最高的点连接,其余的边是要舍弃的边。 建立最邻近图谱的方法就是把聚类问题转化为图分割的问题,转化之后就存在两个问题: (1)数据点与数据点之间的相似度的定义; 图分割说白了就是在图结构空间的聚类,同一般的聚类一样,对结果的验证很重要,尽管常常不容易。 我们用蛋白网络图做以说明。 分类和聚类的结果做一比较 节点间依据某些特征进行选择性连接,被称为同配混合(assortative mixing),用于量化给定网络中同配程度的指标称为同配系数(assortativity coefficient),本质上是相关系数概念的一种变体。 需要注意的是,这里涉及的节点特征可以是分类、有序或者连续的变量。 考虑分类特征的情况:假设图G中每个节点可以使用M个类别中的一个进行标记,此时的同配系数定义为: 其中,fij是G中连接第i类节点与第j类节点的边所占的比例,而fi+和f+i分别是结果矩阵5f的边际行和与列和。 同配系数ra的值介于-1和1之间。当图的混合模式与保留边际度分布时随机分配边的结果一致,该值为0。类似地,当图的混合模式为完全同配(即边只连接同一类节点)时该值为1。但是,当混合模式为完全异配,即图中每条边连接的都是不同类型的节点时,上式中的系数不一定为-1。更多分析见Newman的综述(Newman, 2002)。 当感兴趣的节点特征为连续变量而非离散,使用(xe,ye)表示由一条边e∈E连接的两个节点的特征。量化这一特征同配性的一个自然选择是(xe,ye)的Pearson相关系数: 该值是对所有观察变量对(x,y)的概括,fxy、fx+和f+y的定义与分类变量类似,σx与 σy分别是{fx+}与{f+y}频率分布的标准差。 同配系数r经常在网络结构研究中用于概括相邻节点的度-度相关性。 汇总网络描述性质 https://zhuanlan.zhihu.com/p/40227203vertex_connectivity(yeast.gc)
# ---
## [1] 1
# ---
edge_connectivity(yeast.gc)
# ---
## [1] 1
yeast.cut.vertices <- articulation_points(yeast.gc)
length(yeast.cut.vertices)
350
# CHUNK 30
is_connected(aidsblog, mode=c("weak"))
# ---
## [1] TRUE
# ---
# CHUNK 31
is_connected(aidsblog, mode=c("strong"))
# ---
## [1] FALSE
# ---
# CHUNK 32
aidsblog.scc <- components(aidsblog, mode=c("strong"))
table(aidsblog.scc$csize)
# ---
##
## 1 4
## 142 1
# ---
图分割
层次聚类
次聚类kc <- cluster_fast_greedy(karate)
# CHUNK 34
length(kc)
# ---
## [1] 3
# ---
sizes(kc)
# ---
## Community sizes
## 1 2 3
## 18 11 5
# ---
membership(kc) # 社团归宿
Mr Hi Actor 2 Actor 3 Actor 4 Actor 5 Actor 6 Actor 7 Actor 8 Actor 9 Actor 10 Actor 11
2 2 2 2 3 3 3 2 1 1 3
Actor 12 Actor 13 Actor 14 Actor 15 Actor 16 Actor 17 Actor 18 Actor 19 Actor 20 Actor 21 Actor 22
2 2 2 1 1 3 2 1 2 1 2
Actor 23 Actor 24 Actor 25 Actor 26 Actor 27 Actor 28 Actor 29 Actor 30 Actor 31 Actor 32 Actor 33
1 1 1 1 1 1 1 1 1 1 1
John A
1
plot(kc,karate)
library(ape)
dendPlot(kc, mode="phylo")
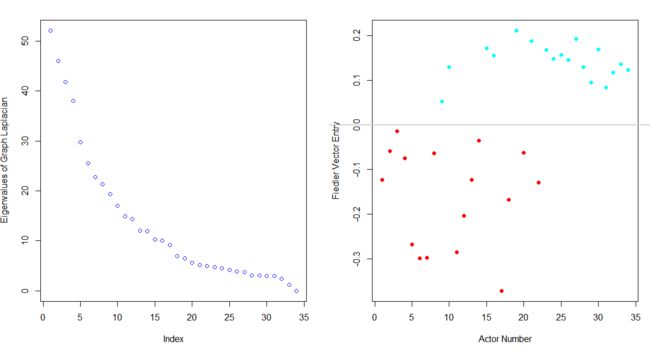
(2)建立最邻近图谱之后要从哪条边或者从哪些边分割最优。# CHUNK 38
k.lap <- laplacian_matrix(karate)
eig.anal <- eigen(k.lap)
# CHUNK 39
plot(eig.anal$values, col="blue",
ylab="Eigenvalues of Graph Laplacian")
# CHUNK 40
f.vec <- eig.anal$vectors[, 33]
# CHUNK 41
faction <- get.vertex.attribute(karate, "Faction")
f.colors <- as.character(length(faction))
f.colors[faction == 1] <- "red"
f.colors[faction == 2] <- "cyan"
plot(f.vec, pch=16, xlab="Actor Number",
ylab="Fiedler Vector Entry", col=f.colors)
abline(0, 0, lwd=2, col="lightgray")
# CHUNK 42
func.class <- vertex_attr(yeast.gc, "Class")
table(func.class)
# ---
## func.class
## A B C D E F G M O P R T U
## 51 98 122 238 95 171 96 278 171 248 45 240 483
# ---
yc <- cluster_fast_greedy(yeast.gc)
c.m <- membership(yc)
c.m
YLR197W YOR039W YDR473C YOR332W YER090W YDR394W YER021W YPR029C YIL106W YKL166C
5 5 3 7 4 4 4 12 7 27
YGL026C YOR061W YGL115W YGL049C YDL140C YLR291C YGR158C YDR328C YOL094C YDR460W
4 5 12 3 1 3 10 9 7 17
YBR154C YOR116C YIL062C YPR010C YER027C YPL093W YER006W YFR052W YOR261C YHR052W
1 1 8 4 12 5 5 4 4 5
YDR280W YOR260W YMR193W YGR162W YPR187W YDR101C YOL041C YHR197W YBL045C YOR207C
10 3 1 3 1 1 5 5 4 1
YPL259C YLL008W YPL043W YGL220W YOR117W YOR310C YNL002C YBR126C YKL014C YCR077C
12 5 5 4 4 5 (略)
# CHUNK 44
table(c.m, func.class, useNA=c("no"))
# ---
## func.class
## c.m A B C D E F G M O P R T U
## 1 0 0 0 1 3 7 0 6 3 110 2 35 14
## 2 0 2 2 7 1 1 1 4 39 5 0 4 27
## 3 1 9 7 18 4 8 4 20 10 23 8 74 64
## 4 25 11 10 22 72 84 81 168 14 75 16 27 121
## 5 1 7 5 14 0 4 0 2 3 6 1 34 68
## 6 1 24 1 4 1 4 0 7 0 1 0 19 16
## 7 6 18 6 76 7 9 3 7 8 5 1 7 33
## 8 8 12 67 59 1 34 0 19 60 10 7 6 73
## 9 4 1 7 7 2 10 5 3 2 0 3 0 11
## 10 0 0 0 6 0 0 0 2 0 5 0 11 1
## 11 0 9 0 10 1 3 0 0 0 0 0 2 4
## 12 0 1 3 0 0 0 0 6 10 0 0 0 2
## 13 0 1 1 2 0 1 0 0 2 0 0 16 10
## 14 1 0 4 1 0 1 0 0 4 0 1 0 11
## 15 0 1 0 0 0 2 0 2 0 0 1 0 8
## 16 0 1 2 0 0 1 0 0 10 0 0 0 0
## 17 0 0 1 3 0 0 0 2 0 0 0 2 3
## 18 0 0 0 0 3 1 0 9 0 0 1 0 1
## 19 0 1 1 1 0 0 0 0 0 0 0 0 3
## 20 0 0 0 6 0 0 0 1 0 0 0 1 2
## 21 1 0 0 0 0 0 0 0 6 0 0 1 0
## 22 0 0 0 0 0 0 0 1 0 0 0 0 8
## 23 0 0 0 0 0 0 0 4 0 0 0 0 0
## 24 0 0 0 0 0 0 2 2 0 0 0 1 0
## 25 0 0 0 0 0 0 0 5 0 0 0 0 0
## 26 0 0 1 0 0 0 0 4 0 0 1 0 1
## 27 3 0 4 0 0 1 0 0 0 0 0 0 0
## 28 0 0 0 0 0 0 0 0 0 6 0 0 0
## 29 0 0 0 1 0 0 0 1 0 0 3 0 0
## 30 0 0 0 0 0 0 0 0 0 2 0 0 2
## 31 0 0 0 0 0 0 0 3 0 0 0 0 0
# ---
同配混合(assortative mixing)
v.types <- (V(yeast)$Class=="P")+1
v.types[is.na(v.types)] <- 1
assortativity_nominal(yeast, v.types, directed=FALSE)
# ---
## [1] 0.5232879
# ---
# CHUNK 46
assortativity_degree(yeast)
# ---
## [1] 0.4610798
# ---
as_adjacency_matrix(yeast) -> igraph
igraph <- network::as.network.matrix(igraph)
nodes_num <- length(V(yeast))
nodes_num
vcount(yeast)
edges_num <- length(E(yeast))
edges_num
#平均度(average degree)
average_degree <- mean(degree(yeast))
#或者,2x边数量/节点数量
average_degree <- 2*edges_num/nodes_num
average_degree
#平均加权度(average weighted degree),仅适用于含权网络
#average_weight_degree <- mean(strength(igraph))
#节点和边连通度(connectivity)
nodes_connectivity <- vertex.connectivity(yeast)
nodes_connectivity
edges_connectivity <- edge.connectivity(yeast)
edges_connectivity
#平均路径长度(average path length)
average_path_length <- average.path.length(yeast, directed = FALSE)
average_path_length
#网络直径(diameter)
graph_diameter <- diameter(yeast, directed = FALSE)
graph_diameter
#图密度(density)
graph_density <- graph.density(yeast)
graph_density
#聚类系数(clustering coefficient)
clustering_coefficient <- transitivity(yeast)
clustering_coefficient
#介数中心性(betweenness centralization)
betweenness_centralization <- centralization.betweenness(yeast)$centralization
betweenness_centralization
#度中心性(degree centralization)
degree_centralization <- centralization.degree(yeast)$centralization
degree_centralization
#模块性(modularity),详见 ?cluster_fast_greedy,?modularity,有多种模型
fc <- cluster_fast_greedy(yeast)
modularity <- modularity(yeast, membership(fc))
#互惠性(reciprocity),仅适用于有向网络
#reciprocity(igraph, mode = 'default')
#reciprocity(igraph, mode = 'ratio')
#选择部分做个汇总输出
igraph_character <- data.frame(
nodes_num, #节点数量(number of nodes)
edges_num, #边数量(number of edges)
average_degree, #平均度(average degree)
nodes_connectivity, #节点连通度(vertex connectivity)
edges_connectivity, #边连通度(edges connectivity)
average_path_length, #平均路径长度(average path length)
graph_diameter, #网络直径(diameter)
graph_density, #图密度(density)
clustering_coefficient, #聚类系数(clustering coefficient)
betweenness_centralization, #介数中心性(betweenness centralization)
degree_centralization, #度中心性
modularity #模块性(modularity)
)
igraph_character %>%
+ knitr::kable(caption = "Power Centrality Descriptives for NEO Big 5 correlation network")
Table: Descriptives
| nodes_num| edges_num| average_degree| nodes_connectivity| edges_connectivity| average_path_length| graph_diameter| graph_density| clustering_coefficient| betweenness_centralization| degree_centralization| modularity|
|---------:|---------:|--------------:|------------------:|------------------:|-------------------:|--------------:|-------------:|----------------------:|--------------------------:|---------------------:|----------:|
| 2617| 11855| 9.059992| 0| 0| 5.095629| 15| 0.0034633| 0.4686178| 0.1299893| 0.0416437| 0.6957512|
https://kateto.net/networks-r-igraph
https://www.jianshu.com/p/9f9108983bcd
https://blog.csdn.net/yyl424525/article/details/103108506
https://www.cnblogs.com/rubinorth/p/5800624.html
https://tidygraph.data-imaginist.com/reference/index.html
https://cran.r-project.org/web/packages/CINNA/vignettes/CINNA.html
https://cloud.tencent.com/developer/article/1667095
https://www.jianshu.com/p/6c980d6aaeb4
https://blog.csdn.net/qq_32416677/article/details/80496756
https://zhuanlan.zhihu.com/p/27874050
https://cloud.tencent.com/developer/article/1667115
https://blog.csdn.net/makenothing/article/details/50110221