医学图像了解
医学图像了解
医学图像了解
医学图像
医学图像是反映解剖区域内部结构或内部功能的图像,它是由一组图像元素——像素(2D)或立体像素(3D)组成的。医学图像是由采样或重建产生的离散性图像表征,它能将数值映射到不同的空间位置上。像素的数量是用来描述某一成像设备下的医学成像的,同时也是描述解剖及其功能细节的一种表达方式。像素所表达的具体数值是由成像设备、成像协议、影像重建以及后期加工所决定的
医学图像有四个关键成分——像素深度、光度表示、元数据和像素数据。这些成分与图像大小和图像分辨率有关
图像深度(又称比特深度或颜色深度)是用来编码每个像素信息的比特数。比如说,一个8比特的光栅可以有256个从0到255数值不等的图像深度
光度表示解释了像素数据如何以正确的图像格式(单色或彩色图片)显示。为了说明像素数值中是否存在色彩信息,我们将引入“每像素采样数”的概念。单色图像只有一个“每像素采样”,而且图像中没有色彩信息。图像是依靠由黑到白的灰阶来显示的,灰阶的数目很明显取决于用来储存样本的比特数。在这里,灰阶数与像素深度是一致的。医疗放射图像,比如CT图像和磁共振(MR)图像,是一个灰阶的“光度表示”。而核医学图像,比如正电子发射断层图像(PET)和单光子发射断层图像(SPECT),通常都是以彩色映射或调色板来显示的
元数据是用于描述图像的信息。它可能看起来会比较奇怪,但是在任何一个文件格式中,除了像素数据之外,图像还有一些其他的相关信息。这样的图像信息被称为“元数据”,它通常以“数据头”的格式被储存在文件的开头,涵盖了图像矩阵维度、空间分辨率、像素深度和光度表示等信息
像素数据是储存像素数值的位置。根据数据类型的不同,像素数据使用数值显示所需的最小字节数,以整点或浮点数的格式储存
图像大小 = 数据头大小(包括元数据) + 行数 栏数像素深度(图像帧数)
医学图像格式
放射图像有6种主要的格式,分别为DICOM(医学数字成像和通讯)、NIFTI(神经影像信息技术)、PAR/REC(Philips磁共振扫描格式)、ANALYZE(Mayo医学成像)、NRRD(近原始栅格数据)和MNIC
现代神经影像学技术
脑电图(EEG),单光子发射体层成像(SPECT),正电子发射型计算机断层显像(PET),功能性磁共振成像(fMRI),侵入性光学成像(Invasive Optical Imaging),颅内电极记录(Intracranial Recording),脑皮层电图(ECoG)。其中应用最为广泛的是fMRI和PET
MNI空间是Montreal Neurological Institute根据一系列正常人脑的磁共振图像而建立的坐标系统
Native空间即原始空间,图像没有做任何处理,此时,不同被试图像之间不具有可比性,必须对所有被试的图像进行配准标准化到同一个模板上,这样所有被试的维度、原点、voxel size就一样了。使用MNI标准模板,就表示把图像转换至MNI空间了
脑成像数据主要有DTI、FMRI、3D三种模态。其中,DTI,3DT1是三维数据,FMRI是四维数据
- DTI:Diffusion Tensor Imaging,磁共振弥散张量成像
- FMRI:Functional Magnetic Resonance Imaging,功能性磁共振成像
- DICOM(Digital Imaging and Communications in Medicine)即医学数字成像和通信是医学图像和相关信息的国际标准(ISO 12052)。它定义了质量能满足临床需要的可用于数据交换的医学图像格式
PET是正电子发射断层显像(Positron Emission Tomography)的缩写,是一种先进的核医学影像技术;CT是计算机断层摄影术(Computed Tomography)的简称,是一种临床已广泛应用且仍在迅速发展的X线断层成像技术。将这两种技术有机地整合到同一台设备上,并把不同性质的图像进行同机融合显示,即形成了PET/CT
各类图像详解
MRI
MRI影像检查有一个突出特点,就是有着多种多样的成像序列。这些成像序列能够产生各具特点的MRI图像,不仅能够反映人体解剖形态,而且能够反映人体血流和细胞代谢等生理功能信息
MRI扫描方式可以简单的划分为常规扫描和功能扫描两大类。常规扫描主要反映解剖形态;功能扫描则以不同方式反映人体新陈代谢、血液流动等功能信息。常规扫描包括T1加权、T2加权成像,血管造影成像,以及动态增强成像等。功能成像包括了弥散加权成像(DWI),灌注加权成像(PWI),磁共振波普成像(MRS)和血氧饱和水平依赖成像(BOLD)等
T1加权突出显示解剖结构,T2加权则能够突出显示病灶
MRI是最常用也是最重要的颅脑病变检查手段。与CT相比,MRI没有骨性伪影,具有更好的软组织分辨能力。并且可以根据需要,灵活选择轴、冠、矢及斜位扫描,充分显示病变
MRI被应用于心脏检查的多个方面,包括:形态学检查,提供心脏解剖的高分辨率成像;心脏功能学检查,评价射血分数EF,每搏输出量SV,收缩末期和舒张末期容积,心输出量,以及瓣膜性能;心肌灌注和心肌活性;冠脉解剖和血流;心肌代谢;高分辨率血管壁斑块成像
脊椎检查中通常使用T1、T2扫描序列,并且会在矢状位、轴位、冠状位和任意角度生成多个扫描序列
MRI是前列腺增生和前列腺癌诊断的常用检查手段之一,通常使用T1和T2扫描,使用最多的扫描方向为横轴位。此外,也会选用动态增强扫描,弥散加权成像(DWI)和磁共振波谱成像(MRS)
对于肝脏,通常使用T1、T2扫描,以及动态增强扫描。动态增强扫描技术应视为肝脏特别是肝癌检查的常规方法
fMRI
fMRI原理
神经细胞活动时需要消耗氧气,这些氧气是依靠神经细胞周围的微血管中的血红蛋白输送过来的。因此,当神经活动时,其附近的血流会增加以及时补充消耗掉的氧气,最终使局部的血液增加,促使血液中的含氧血红蛋白和脱氧血红蛋白的浓度发生改变。而脱氧血红蛋白是一种顺磁性物质,其浓度的变化会引起磁共振信号强度的变化。fMRI扫描仪以一定的时间分辨率对这种连续变化的磁信号采样,最终得到能够反映神经细胞(元)活动的时间序列。在核磁医学上,把由于含氧血红蛋白和脱氧血红蛋白浓度的变化而引起的磁共振信号的变化称为血氧水平依赖(blood oxygen level dependent,BOLD)效应,并把相应的磁共振信号也称为BOLD信号
fMRI数据特点
- 信噪比低
- 数据维度高
- 数据分布差异较大
MRI与FMRI
MRI扫的是大脑的结构图像,也叫T1权重图像。它有着很高的空间分辨率,可以从中看到非常清晰的解剖结构,也可以从中区分出各种不同的组织
fMRI往往用于研究大脑的具体功能,扫出来的是功能图像,也叫做T2*权重图像。虽然它的空间分辨率比较低但是时间分辨率很高,可以在很短的时间内扫出一叠功能图像。这样就可以研究实验操作究竟是如何影响大脑的MRI信号的
FMRI数据预处理
数据预处理步骤包括:可视化(Visualization)、去伪影(Artifact removal)、时间配准(Slice time correction)、头动校正(Motion correction)、生理噪音校正(Correction for physiological effect)、结构功能配准(Co-registration)、标准化(Normalization)和时空间滤波(Spatial and temporal filtering)
fMRI分析的主要步骤
- 质量控制:确保数据不被伪迹破坏
- 扭曲校正:校正fMRI图像经常发生的空间扭曲失真
- 头动校正:校正头动,将扫描的时间序列图像重新对准
- 层间时间校正:校正图像不同层之间的时间差异
- 空间标准化:将不同个体的数据对准到一个通用空间结构上,使得所有数据可以合并进行组分析
- 空间平滑:有意模糊数据以降低噪声
- 时间过滤:在时间维度上过滤数据,以去除低频噪声
- 统计建模:将统计模型拟合到观测数据,以估计任务或刺激引起的响应
- 统计推断:估计结果的统计显著性,对在整个大脑中进行的大量统计检验进行校正
- 可视化:对结果进行可视化,并估计效应量
简化步骤:
- 对齐:在FMRI序列当中,由于人为的因素而引起的误差是很严重的,例如头部的微小移动。因此在处理数据之前应当将这些人为的因素减小到最小
- 标准化:由于个体之间存在差异,在提取个体之间的均值信号或者在标准空间的坐标系统下描述激活区时,需要将许多个体的图像形变至同一标准空间,即图像空间标准化。目前最常用的是Talairach和Tournoux标准脑图谱
- 配准:为了功能激活区的精确定位,通常将功能信息定位在分辨率较高的解剖图像(比如T1加权像)上,这就需要将功能图像和解剖图像进行配准
- 平滑:平滑主要有两个作用,即增加信噪比;可以对于图像部分效果进行增强
基于MRI标准坐标空间的三个主要坐标轴示意图
在用于神经成像数据的标准空间中,X代表左/右;Y代表前/后;Z代表上/下。在数据矩阵中,一个特定的体素可以被标记为[Xvox, Yvox, Zvox],通过这三个维度的坐标就可以确定体素的位置。如下所示: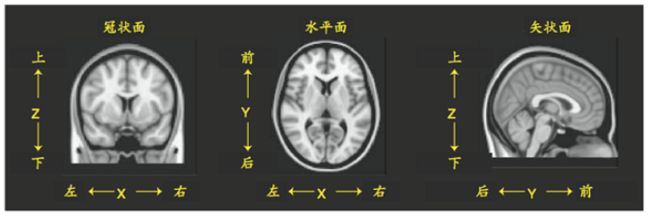
Dicom
它定义了质量能满足临床需要的可用于数据交换的医学图像格式,可用于处理、存储、打印和传输医学影像信息。DICOM可以便捷地交换于两个满足DICOM格式协议的工作站之间
一个DICOM文件由一个数据头和图像数据组成的。数据头的大小取决于数据信息的多少。数据头中的内容包括病人编号、病人姓名等等。同时,它还决定了图像帧数以及分辨率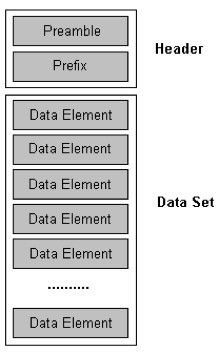
每个病人的一次扫描CT(scan)可能有几十到一百多个dcm数据文件(slices)。后缀为 .dcm,可以使用 python的dicom包读取,一般使用其pixl_array数据
Dicom格式数据处理过程
医学扫描图像(scan)其实是三维图像,使用代码读取之后开源查看不同的切面的切片(slices),可以从不同轴切割
其次,CT扫描图是包含了所有组织的,如果直接去看,看不到任何有用信息。需要做一些预处理,预处理中一个重要的概念是放射剂量,衡量单位为HU(Hounsfield Unit),下表是不同放射剂量对应的组织器官
| substance | HU |
|---|---|
| 空气 | -1000 |
| 肺 | -500 |
| 脂肪 | -100 ~ -50 |
| 水 | 0 |
| CSF | 15 |
| 肾 | 30 |
| 血液 | 30 ~ 45 |
| 肌肉 | 10 ~ 40 |
| 灰质 | 37 ~ 45 |
| 白质 | 20 ~ 30 |
| Liver | 40 ~ 60 |
| 软组织、contrast | 100 ~ 300 |
| 骨头 | 700(软骨) ~ 3000(皮质骨) |
计算方法:
Hounsfield Unit = pixel_value * rescale_slope + rescale_intercept一般情况rescale slope = 1, intercept = -1024
灰度值是pixel value经过重重LUT转换得到的用来进行显示的值,而这个转换过程是不可逆的,也就是说,灰度值无法转换为ct值。只能根据窗宽窗位得到一个大概的范围
一个简单的dicom图片处理
import dicom
import pylab
ds = dicom.read_file("1.2.840.113619.2.55.3.2831193967.596.1285460208.412.1.DCM")
print("图片属性: ", ds.dir("pat"))
print("病人: ", ds.PatientName)
# CT值得矩阵
pix = ds.pixel_array
# 读取图片
pylab.imshow(ds.pixel_array, cmap=pylab.cm.bone)
pylab.show()
# 将图片中小于300的值置0
for n, val in enumerate(ds.pixel_array.flat):
if val < 300:
ds.pixel_array.flat[n] = 0
ds.PixelData = ds.pixel_array.tostring()
ds.save_as("output.dcm")
NIfTI格式
NIFTI中一个主要的特点在于它包含了两个仿射坐标定义,这两个仿射坐标定义能够将每个立体元素指标(i,j,k)和空间位置(x,y,z)联系起来
标准NIfTI图像的扩展名是(.nii),包含了头文件及图像资料。同时NIfTI也可使用独立的图像文件(.img)和头文件(.hdr)
DICOM和NIFTI间的区别
DICOM和NIFTI之间最主要的区别在于NIFTI中的原始图像数据是以3D图像的格式储存的,而DICOM是以3D图像片段的格式储存的。这就是为什么在一些机器学习应用程序中NIFTI比DICOM更受欢迎,因为它是3D图像模型。处理一个单个的NIFTI文件,与处理上百个DICOM文件相比要轻松得多。NIFTI的每一张3D图像中只需储存两个文件,而在DICOM中则要储存更多文件。
PET
PET的独特作用是以代谢显像和定量分析为基础,应用组成人体主要元素的短命核素如11C、13N、15O、18F等正电子核素为示踪剂,不仅可快速获得多层面断层影象、三维定量结果以及三维全身扫描,而且还可以从分子水平动态观察到代谢物或药物在人体内的生理生化变化,用以研究人体生理、生化、化学递质、受体乃至基因改变。近年来,PET在诊断和指导治疗肿瘤、冠心病和脑部疾病等方面均已显示出独特的优越性
PET是英文 Positron Emission Tomography的缩写。其临床显像过程为:将发射正电子的放射性核素(如F-18等)标记到能够参与人体组织血流或代谢过程的化合物上,将标有带正电子化合物的放射性核素注射到受检者体内。让受检者在PET的有效视野范围内进行PET显像
CT的基本原理是图像重建,根据人体各种组织(包括正常和异常组织)对X射线吸收不等这一特性, 将人体某一选定层面分成许多立方体小块(也称体素)X射线穿过体素后,测得的密度或灰度值称为象素。X射线束穿过选定层面,探测器接收到沿X射线束方向排列的各体素吸收X射线后衰减值的总和,为已知值,形成该总量的各体素X射线衰减值为未知值,当X射线发生源和探测器围绕人体做圆弧或圆周相对运动时。用迭代方法求出每一体素的X射线衰减值并进行图像重建,得到该层面不同密度组织的黑白图像
PET-CT将PET与CT完美融为一体,由PET提供病灶详尽的功能与代谢等分子信息,而CT提供病灶的精确解剖定位,一次显像可获得全身各方位的断层图像,具有灵敏、准确、特异及定位精确等特点,可一目了然的了解全身整体状况,达到早期发现病灶和诊断疾病的目的
参考文献:
Python与医疗图像2
常见医疗扫描图像处理步骤
深度学习下的医学图像分析 2
MRI影像学习笔记
医学数据集及机器学习项目
面向fMRI数据的人脑功能划分