聚类算法之——DBSCAN密度聚类详解及sklearn包中的DBSCAN算法代码实现
sklearn官方文档参考资料:http://lijiancheng0614.github.io/scikit-learn/modules/generated/sklearn.cluster.DBSCAN.html#sklearn.cluster.DBSCAN
DBSCAN(Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法)是一种基于密度的空间聚类算法。 该算法将具有足够密度的区域划分为簇,并在具有噪声的空间数据库中发现任意形状的簇,它将簇定义为密度相连的点的最大集合。
西瓜书上对它的解释:
核心对象定义:
密度直达定义:
密度可达:
密度相连:
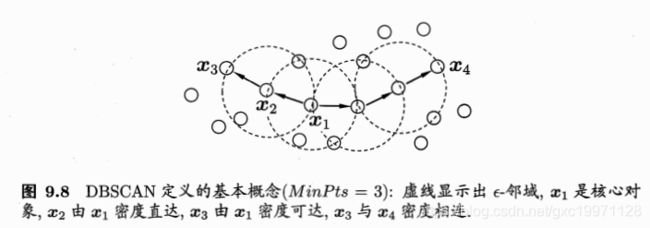
上面几种关系的直观图示如下:
如上图所示,X1的邻域内有多于3个的样本,所以X1是一个核心对象,X2位于X1的邻域内,所以X2由X1密度直达,X3与X2为密度直达,所以X3可由X1密度可达,而X4由X1密度相连。
簇C中的数据满足:
相连型:如果Xi与Xj属于C,则Xi与Xj密度相连;
最大型:如果Xi属于簇C,而Xj由Xi密度相连,则说明Xj也属于簇C
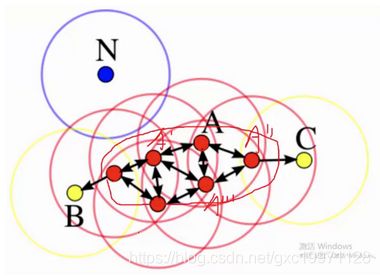
通俗理解如下图所示:(参考博客:https://blog.csdn.net/huacha__/article/details/81094891)
红色中A是核心店,黄色中B和C是边界点;蓝色中N是离群点
形象来说,我们可以认为这是系统在众多样本点中随机选中一个,围绕这个被选中的样本点画一个圆,规定这个圆的半径以及圆内最少包含的样本点,如果在指定半径内有足够多的样本点在内,那么这个圆圈的圆心就转移到这个内部样本点,继续去圈附近其它的样本点,类似传销一样,继续去发展下线。等到这个滚来滚去的圈发现所圈住的样本点数量少于预先指定的值,就停止了。那么我们称最开始那个点为核心点,如A,停下来的那个点为边界点,如B、C,没得滚的那个点为离群点,如N
从上面的描述中可以看出,该算法需要两个参数: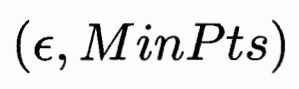 即半径和半径所画的圆圈住的点数。DBSCAN算法的过程为:
即半径和半径所画的圆圈住的点数。DBSCAN算法的过程为:
伪代码如下:
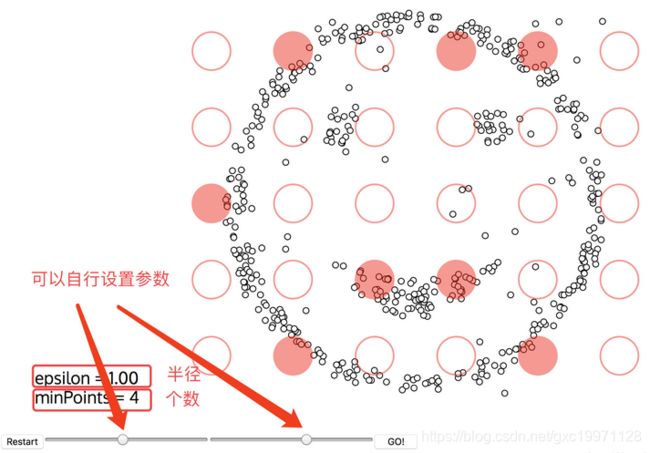
可以用图示来表示:(https://www.naftaliharris.com/blog/visualizing-dbscan-clustering/)
1.首先,设置好两个参数:
2.开始聚类:
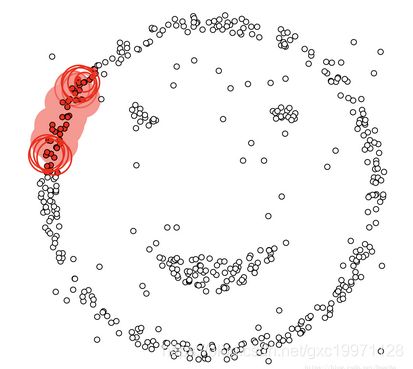
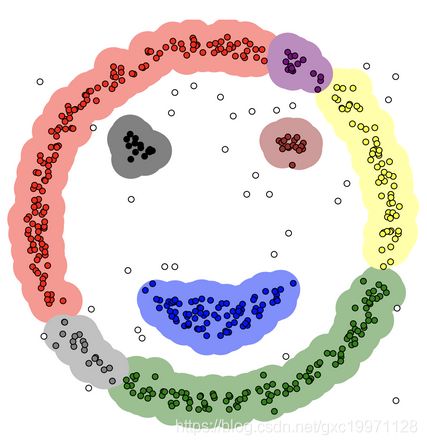
3.DBSCAN聚类结果:
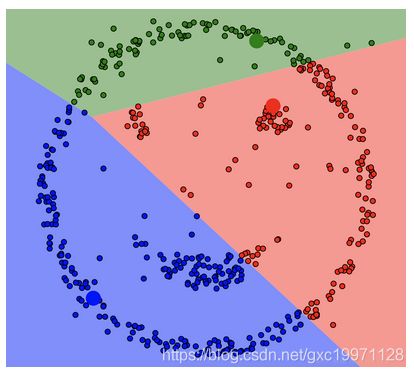
没有颜色标注的就是圈不到的样本点,也就是离群点,DBSCAN聚类算法在检测离群点的任务上也有较好的效果。如果是传统的Kmeans聚类:
代码实现:
import numpy as np # 数据结构
import sklearn.cluster as skc # 密度聚类
from sklearn import metrics # 评估模型
import matplotlib.pyplot as plt # 可视化绘图
data=[
[-2.68420713,1.469732895],[-2.71539062,-0.763005825],[-2.88981954,-0.618055245],[-2.7464372,-1.40005944],[-2.72859298,1.50266052],
[-2.27989736,3.365022195],[-2.82089068,-0.369470295],[-2.62648199,0.766824075],[-2.88795857,-2.568591135],[-2.67384469,-0.48011265],
[-2.50652679,2.933707545],[-2.61314272,0.096842835],[-2.78743398,-1.024830855],[-3.22520045,-2.264759595],[-2.64354322,5.33787705],
[-2.38386932,6.05139453],[-2.6225262,3.681403515],[-2.64832273,1.436115015],[-2.19907796,3.956598405],[-2.58734619,2.34213138],
[1.28479459,3.084476355],[0.93241075,1.436391405],[1.46406132,2.268854235],[0.18096721,-3.71521773],[1.08713449,0.339256755],
[0.64043675,-1.87795566],[1.09522371,1.277510445],[-0.75146714,-4.504983795],[1.04329778,1.030306095],[-0.01019007,-3.242586915],
[-0.5110862,-5.681213775],[0.51109806,-0.460278495],[0.26233576,-2.46551985],[0.98404455,-0.55962189],[-0.174864,-1.133170065],
[0.92757294,2.107062945],[0.65959279,-1.583893305],[0.23454059,-1.493648235],[0.94236171,-2.43820017],[0.0432464,-2.616702525],
[4.53172698,-0.05329008],[3.41407223,-2.58716277],[4.61648461,1.538708805],[3.97081495,-0.815065605],[4.34975798,-0.188471475],
[5.39687992,2.462256225],[2.51938325,-5.361082605],[4.9320051,1.585696545],[4.31967279,-1.104966765],[4.91813423,3.511712835],
[3.66193495,1.0891728],[3.80234045,-0.972695745],[4.16537886,0.96876126],[3.34459422,-3.493869435],[3.5852673,-2.426881725],
[3.90474358,0.534685455],[3.94924878,0.18328617],[5.48876538,5.27195043],[5.79468686,1.139695065],[3.29832982,-3.42456273]
]
#array函数用于返回一个数组
X = np.array(data)
#设置半径为1.5,最小样本量为3,建模。其中fit(X)用于从特征或距离矩阵执行DBSCAN聚类
db = skc.DBSCAN(eps=1.5, min_samples=3).fit(X) #DBSCAN聚类方法 还有参数,matric = ""距离计算方法
labels = db.labels_ #和X同一个维度,labels对应索引序号的值 为她所在簇的序号,也就是聚类之后的结果。若簇编号为-1,表示为噪声
print('每个样本的簇标号:')
print(labels)
raito = len(labels[labels[:] == -1]) / len(labels) #计算噪声点个数占总数的比例
print('噪声比:', format(raito, '.2%'))
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0) # 获取分簇的数目
print('分簇的数目: %d' % n_clusters_)
print("轮廓系数: %0.3f" % metrics.silhouette_score(X, labels)) #轮廓系数评价聚类的好坏
for i in range(n_clusters_):
print('簇 ', i, '的所有样本:')
one_cluster = X[labels == i]
print(one_cluster)
plt.plot(one_cluster[:,0],one_cluster[:,1],'o')
plt.show()