arxiv202210 | cTransformer:基于Transformer的De Novo Molecular Design生成模型
Preprint。
原文标题: A Transformer-based Generative Model for De Novo Molecular Design
地址:https://arxiv.org/abs/2210.08749
2022年10月17挂到arxiv上的。新鲜出炉的,有点启发,单目标控制生成。
一、问题
小分子药物设计旨在识别具有所需化学性质的新化合物。
从计算的角度来看,作者认为这是一个优化问题,在化学空间中搜索将最大化定量目标的化合物。然而,由于搜索空间非常大,这种优化任务在计算上是难以处理的。据估计,潜在的类药物分子的范围在10^23到10^60之间,但只有大约10^8个分子被合成。许多计算方法,如虚拟筛选,组合库,和进化算法等已经出现了很多。
二、模型方法
将分子设计问题表述为给定目标蛋白的条件顺序生成,并提出 conditional Transformer架构,自回归生成目标特异性化合物。
1、模型
为利用化学空间中大量未标记的训练数据,而不是seq2seq编码器-解码器模型,仅考虑解码器模型,预测给定token的下个token。
任务规范为:(1)自回归标记生成,(2)通过对训练集的观察,生成遵循SMILES语法(例如,原子类型、键类型和分子大小),(3)生成产生更多以前没有观察到的变体。
2、Incorporating Target Information
主要挑战是生成针对目标的SMILES序列。该模型需要记住有效的类药物结构和靶标特异性信息。挑战在于获取特定目标的信息并进一步生成特定目标的信息。作者提出利用Transformer decoder中的多头注意特性,对注意操作的Key和Value进行目标特定的embedding。将带有强制conditional embedding(例如,特定于目标的embedding,该Transformer模型表示为cTransformer
使用目标特定的embedding作为第二个multi-head attention的key和value,这允许解码器的每个位置都关注目标特定的embedding,并确保后续的token生成以目标特定的embedding为条件。
目标特定设计与解码器正交,可通过设置conditional embedding为zero embedding轻松删除。
对于基础模型预训练,Target-specific embedding初始化为zero embeddings;当涉及到目标时,Target-specific embedding使用特定于目标的embedding进行初始化。
训练过程:
1)首先通过将特定于目标的embedding设置为zero embeddings来预训练cTransformer(不提供特定于目标的信息)。
2)为了提供特定于目标的信息,使用
3)通过从经过训练的decoder中自回归采样token来生成类药物结构。还可以通过提供特定于目标的embedding来指定所需的目标。
三、实验
两个主要任务,第一个任务是分子生成建模(使用cTransformer的基础模型)。验证transformer对小分子生成任务是有利的。第二项任务是针对目标的生成,表明可以生成目标偏斜化合物
1、Molecular generative model
Dataset:MOSES分子数据集cTransformer执行无监督的预训练,目标特定的embedding初始化为零。包含从ZINC clean Lead Collection中提取的1760,739个药物分子,其中训练1,584,664个,测试176,075个。
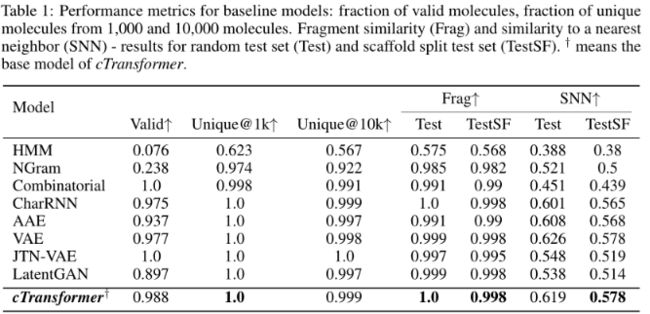
Evaluation Metrics:
- Fraction of valid (Valid) and unique molecules
- Unique@1K and Unique@10K,用于生成集中前1000和10000个有效分子。validity衡量的是模型是否捕捉到足够的化学约束(例如,价);Uniqueness度量模型是否与训练过的分子重叠。
- Fragment similarity (Frag):比较 BRICS fragments 在生成集和reference集中的分布。度量两个数据集中分子骨架的相似性。
- Similarity to a nearest neighbor (SNN):计算生成集中分子的指纹与reference集中最近的分子之间的平均Tanimoto相似度(也称为Jaccard指数)。
- Molecular weight (MW)
- LogP:辛醇-水分配系数。
- Synthetic Accessibility Score (SA):合成容易度(合成可及性)
- Quantitative Estimation of Drug-likeness (QED):根据可取性在0(所有属性不利)到1(所有属性有利)之间的值测量药物相似度。
效果对比:
比较生成数据集和测试数据集中四种分子特性的分布:MW、logP、QED和 SA。模型与真实的数据分布非常接近。cTransformer能够生成类药物分子:
2、Target-specific molecular generation
Dataset:EGFR 1381, S1PR1 795, and HTR1A 3485个分子
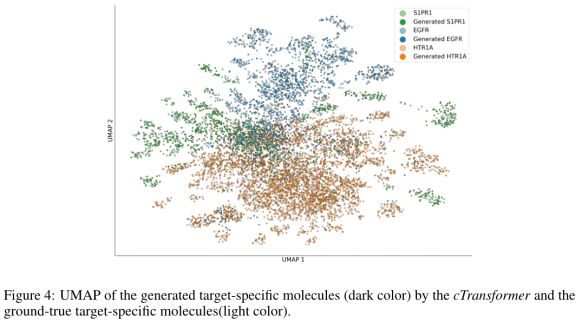
Results:通过可视化的化学空间生成目标特异性化合物。其假设是,可能与相同蛋白质目标相互作用的化合物将占据相同的亚化学空间。为了评估化学空间的重叠,首先为每个目标生成1000个化合物,然后计算生成的化合物和目标特定训练数据集的1024位FCFP6指纹向量。使用UMAP来构造二维投影。
每个点对应一个分子,并根据它的目标标签着色。深色和浅色分别代表生成的化合物和地面真实的靶特异性分子。显示了生成的目标特异性分子(深色)和真实的目标特异性分子(浅色)占据相同的亚化学空间。这些结果表明,cTransformer可以生成类似于训练集中的化合物,但仍然是新的结构。