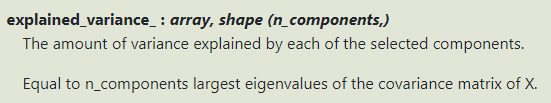python实现PCA降维
本文包括两部分,使用python实现PCA代码及使用sklearn库实现PCA降维,不涉及原理。
总的来说,对n维的数据进行PCA降维达到k维就是:
- 对原始数据减均值进行归一化处理;
- 求协方差矩阵;
- 求协方差矩阵的特征值和对应的特征向量;
- 选取特征值最大的k个值对应的特征向量;
- 经过预处理后的数据乘以选择的特征向量,获得降维结果。
实验数据
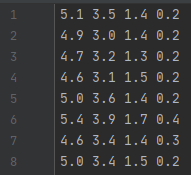
数据data.txt使用[2]中编写的数据,以下是部分数据截图:
shape为(31, 4),即31条特征数为4的数据。
使用python实现PCA
完整代码参考[2],这里逐步做详细介绍。
1. 导入数据
import numpy as np
import pandas as pd
datafile = "data.txt"
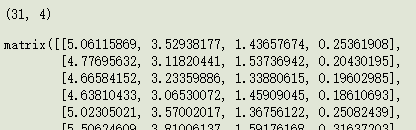
XMat = np.array(pd.read_csv(datafile,sep=" ",header=None)).astype(np.float)
XMat.shape(31, 4)2. 去除平均值
求得XMat每列的平均值:
average = np.mean(XMat, axis=0)
averagearray([5.01935484, 3.43870968, 1.47741935, 0.24516129])扩展均值shape由(1, 4)到(31, 4):
# avgs为average的前m=31行,因为average只有1行,因此这31行是一样的
# np.tile(average, (m, 1))表示是二维的,m行4列
m, n = np.shape(XMat)
avgs = np.tile(average, (m, 1))
print(avgs.shape)
# np.tile(average, m)是一维的,有m*4个值
print(np.tile(average, m))
print(np.tile(average, m).shape)(31, 4)avgsXMat减去其每列均值:
data_adjust = XMat - avgs
data_adjust3. 求XMat协方差矩阵的特征值和特征向量
因为是4列的数据即4维特征,因此协方差是4x4的:
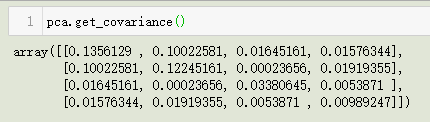
covX = np.cov(data_adjust.T)
covXarray([[0.1356129 , 0.10022581, 0.01645161, 0.01576344],
[0.10022581, 0.12245161, 0.00023656, 0.01919355],
[0.01645161, 0.00023656, 0.03380645, 0.0053871 ],
[0.01576344, 0.01919355, 0.0053871 , 0.00989247]])特征值 0.23301081 对应特征向量为第一条列向量 [-0.72506043,-0.67669491,-0.06368499,-0.11097564],这里的特征向量都归一化为单位向量:
featValue, featVec= np.linalg.eig(covX)
featValue
featVecarray([0.23301081, 0.04211748, 0.02128637, 0.00534878])array([[-0.72506043, -0.39050501, 0.5585571 , 0.09903116], [-0.67669491, 0.49557031, -0.48975083, -0.23798779], [-0.06368499, -0.77541379, -0.58351247, -0.23278934], [-0.11097564, -0.02548266, -0.32813302, 0.93774397]])
4. 选择最大的k个特征值对应的k个特征向量
按照特征值从大到小排序,index显示位置:
index = np.argsort(-featValue)
index
# 下面是从小到大
np.argsort(featValue)array([0, 1, 2, 3], dtype=int64)array([3, 2, 1, 0], dtype=int64)
因为特征向量是列向量,这里转化成横向量:
k = 2
selectVec = np.matrix(featVec.T[index[:k]])
selectVecmatrix([[-0.72506043, -0.67669491, -0.06368499, -0.11097564],
[-0.39050501, 0.49557031, -0.77541379, -0.02548266]])5. 将样本点投影到选取的特征向量上
即数据集*特征向量的转置:
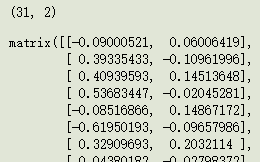
finalData = data_adjust * selectVec.T # (31, 4) * (4, 2) = (31, 2)
finalData.shape
finalData6. 计算重构误差[3]
还原对应投影后的数据:
reconData = (finalData * selectVec) + average
print(reconData.shape)
reconData根据[4]中的:
![]()
其中,m是样本个数,即数据的行数31。x是经过去均值处理的原始数据,这里是data_adjust。x(approx)是经过重构后还原的数据,这里是reconData。
求误差平方和,计算err1:
errMat = XMat - reconData
err1 = np.sum(np.array(errMat)**2) / n
err10.19976362846119633计算err2:
err2 = np.sum(data_adjust**2) / n
err22.2632258064516138计算η:
eta = err1/err2
eta0.0882650011729915根据[4],1-η=91%左右,说明该数据取k=2进行PCA降维时,能保留91%以上的信息。
使用sklearn库实现PCA降维
PCA的api详见[5],下面说明一些常用的属性和方法。
from sklearn.decomposition import PCA
pca = PCA(n_components=k)
pca.fit(XMat)1. n_components参数:
- 默认值为保留所有特征值,即不进行降维:
pca = PCA()
pca.fit(XMat)
pca.explained_variance_array([0.23301081, 0.04211748, 0.02128637, 0.00534878])explained_variance_ 即协方差矩阵的k个最大特征值,这里n_components是默认值,因此k=4。
- n_components == 'mle' 时, 会自动确认降维维度,但是好像结果就是n-1(n是原始数据的列数,即特征个数):
pca.explained_variance_array([0.23301081, 0.04211748, 0.02128637])k=3的结果。
- 0 < n_components < 1 时,即指定降维后的 方差和 占比,比例越大,降维后保留的信息越多,会自动确认降维的维度。
当n_components=0.9时是k=2的效果,n_components=0.95时是k=3的效果。
可以通过[6]中的画图来获取想要保留的信息和维度的关系:
import matplotlib.pyplot as plt
def pcaImg(XMat):
pca=PCA( )
pca.fit(XMat)
ratio=pca.explained_variance_ratio_
k = pca.n_components_
print("pca.n_components_", k)
# 绘制图形
x = [i+1 for i in range(k)]
y = [np.sum(ratio[:i+1]) for i in range(k)]
print(y)
plt.plot(x, y)
plt.xticks(1+np.arange(k,step=1))
plt.yticks(np.arange(0,1.01,0.05))
plt.grid()
plt.show()
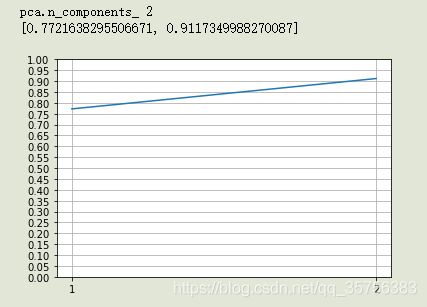
pcaImg(XMat)
横坐标表示k,纵坐标表示降维后可保留的信息。
可以看到,当k=1时能保留大概77%的信息;k=2时为91%,k=3时为98%,k=4时即没有降维,没有损失信息。
取 n_components = 0.9 时,打印结果:
确实是k=2。
2. components_ 属性
输出k个特征向量,每一行代表一个特征向量:
3. explained_variance_ 属性
输出所选择的k个最大特征值:
4. explained_variance_ratio_ 属性
保留的维度的方差百分比,就是每个特征值占其所有特征值总值的占比:
即k=1时,降维后可保留的信息为77%,k=2时为77%+24%等。
一般来说,占比过小的可以舍弃,即这里取k=2或3较好,具体情况具体分析。
5. n_components_ 属性
即设置的降维维度k:
6. fit_transform(X[, y]) 方法
获得降维后的结果,和 transform(X) 方法差不多:
7. get_covariance() 方法
获得协方差矩阵:
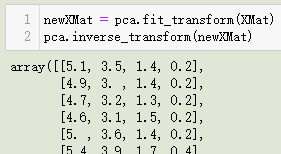
8. inverse_transform(X) 方法
由降维结果返回原始结果:
这里因为k=n,所有还原后就是原始值,实际上当k 当数据维度过高时可使用降维手段如PCA,来使得维度变低,避免维度灾难,同时加快运算速度。 PCA降维本质上就是一种信息压缩,如把shape为(31, 4)的数据压缩为(31, 2)。 关于降维维度k的确定,可以使用sklearn中的PCA模块来获取数据的方差百分比,根据以上绘图的方法来确定最佳k值。 有些研究工作表明,所选的主轴总长度占所有主轴长度之和的大约85% 即可。即保留的维度的方差总百分比到达85%以上,但实际效果还是依靠训练之后产生的预测结果的评估来决定。 确定好了k值后,即可快速根据API获取降维后的数据,来进行后续的实验。 [1] 降维算法一 : PCA (Principal Component Analysis) [2] 机器学习降维之PCA(python代码+数据) [3] PCA降维以及维数的确定 [4] PCA主成分数量(降维维度)选择 [5] sklearn.decomposition.PCA [6] 通过PCA选择合适降维维度总结
参考文档