Briefings in bioinformatics2022 | Knowledge-based BERT+:像计算化学家一样提取分子性质的方法
原文标题:Knowledge-based BERT+: a method to extract molecular features like computational chemists
论文地址:Knowledge-based BERT: a method to extract molecular features like computational chemists | Briefings in Bioinformatics | Oxford Academic
代码:GitHub - wzxxxx/Knowledge-based-BERT: K-BERT for molecular property prediction.
一、问题提出
主流的基于ML的分子性质预测方法大致可分为三类:基于描述符的、基于图的和基于smiles的。
基于描述符的模型的质量对用于训练的特征工程中分子特征相当敏感,描述符/指纹的生成需要广泛的人类专家知识。
基于图的方法将分子描述为具有节点(原子)和边(键)的分子图,而不是固定长度的特征向量,并通过图神经网络(GNN)框架,从由原子和键的性质定义的简单初始特征和分子的拓扑结构中自动提取分子特征。在一定程度上避免了人工提取描述符/指纹过程中丢失任务相关信息。然而,由于数据驱动方法的普遍问题,基于图的方法严重依赖于数据量,当数据集较小时,其性能甚至不如基于描述符的方法。此外,GNN容易出现过平滑问题,因此GNN的层数一般只有2-4层,这限制了其特征提取能力。因此,基于图的方法在某些任务上取得了优异的性能,但在药物发现方面并没有带来突破性的进展。
基于SMILES的方法在分子性质预测中的应用不如基于描述符和图的方法广泛。基于SMILES的分子性质预测可以看作是NLP问题。基于SMILES方法的一个内在优势是,可以直接从SMILES中提取分子特征,而不依赖于任何人工特征工程。由于分子的结构和化学信息隐含在SMILES中,不像分子描述符/指纹和分子图那样明确,因此基于smile的方法对特征提取能力和数据量有更高的要求。基于smiles的方法在预测分子性质方面的性能可能不如基于图和描述符的方法。
与自然语言不同的是,SMILES中“词汇”的数量要比自然语言少得多,而且有大量的“同义词”。基于NLP模型容易受到小型且往往难以察觉的干扰(如同义词)。即使是被认为是最健壮的NLP模型BERT,也可能因为几个同义词的替换而做出错误的判断,而人类会做出正确的判断。因此,为了避免模型受到这些同义词的影响,提出了一种新的预训练策略来学习化学家如何从SMILES中提取化学信息。
二、Methods and materials
1、Collection of datasets
Pre-training dataset
使用CHEMBL中的小分子对K-BERT进行预训练。对所有分子进行规范化处理,去除混合物、无机物和RDKit无法处理的分子。最后,将近180万个分子被用于预训练。
Small pharmaceutical datasets for molecular property predictions
评估KBERT在分子性质预测方面的性能,从ADMETlab 2.0中收集15个分子数小于2000的小型药物数据集。
Pgp-substrate (Pgp-sub), human intestinal absorption (HIA), human oral bioavailability20% (F20%), human oral bioavailability 30% (F30%), CYP-substrate (CYP1A2-sub, CYP2C19-sub, CYP2C9-sub,CYP2D6-sub and CYP3A4-sub), half-life (T1/2), drug-induced liver injury (DILI), FDA maximum recommended daily dose (FDAMDD), skin sensitization (SkinSen),carcinogenicity (Carcinogenicity) and respiratory toxicity (Respiratory).
Malaria dataset
用于验证K-BERT是否可以捕获手性信息
CHIRAL1 dataset
共使用204 778个分子进行进一步的预训练。
2、Model
1)预训练任务
3个预训练任务:原子特征预测任务、分子特征预测任务和对比学习任务
原子特征预测任务允许模型学习基于graph手动提取的信息(初始原子信息),分子特征预测任务允许模型学习基于描述符的方法中手动提取的信息(分子描述符/指纹),对比学习任务允许模型使同一分子的不同SMILES字符串的embedding更加相似,从而使K-BERT可以归纳出不同格式的SMILES。
Atom features prediction task:
使用RDKit计算分子中每个重原子的原子特征,本任务的目标是预测原子特征。原子特征包括程度、芳香性、氢原子数、手性和手性类型。因此,原子特征预测任务可以看作是一个多任务分类任务。
Molecular features prediction task:
利用RDKit计算分子的分子特征,本任务的目标是预测分子特征(MACCS fingerprints)。因此,分子特征预测任务也可以看作是一个多任务分类任务。
Contrastive learning task:
对于一个规范的SMILES输入,通过SMILES排列生成4种不同的SMILES形式。目标是使同一分子的不同SMILES string之间的embedding的余弦相似度最大化,使不同分子之间的embedding相似度最小化。因此,损失函数如下:
其中N为batch_size,Dn表示分子n生成的4种不同的smiles形式,Bn表示batch中除分子n外的其他分子。
2)Pretraining
transformer的hidden_size为768,heads为12,6个层transformer。大约180万个分子。基于原子tokenizer,预训练8个epoch。
3)Fine-tuning
保持相同的框架,并在预训练模型中加载前5个transformer block。将第6个transformer block和显式下游任务head以全连接层的形式重新初始化。然后,针对特定的下游任务进行K-BERT优化。
3、Data augmentation
下游任务的训练集、验证集和测试集中分子的SMILES均通过rdkit增加5(1 + 4)倍。在训练集中,每个分子被视为独立的分子,但同一分子的不同SMILES串被视为同一分子。对验证集和测试集中相同分子的不同SMILES的预测进行整合,得到分子的输出。
4、Model evaluation
1)baseline
XGBoost、focused FP、HRGCN+
按8:1:1的比例随机分割为训练集、验证集和测试集
用coefficient of determination(R2)、平均绝对误差(MAE)和均方根误差(RMSE)对回归任务进行评价。用受试者工作特征曲线下面积(ROC-AUC)和Matthews correlation coefficient(MCC)对分类任务进行评价。
三、实验
1、The performance of knowledge-based BERT
证实K-BERT在分子性质预测方面具有很强的预测能力:
2、Pre-training can significantly enhance the model's ability to extract molecular features
K-BERT在15个任务上都优于K-BERT -WP,平均提高了10.6%,这充分证明了预训练可以提高模型提取分子特征的能力
3、Contrastive learning pre-training task enables K-BERT to 'understand' SMILES better
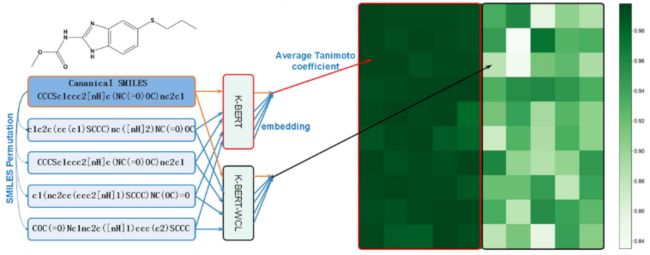
通过对180万个大规模未标记分子的预训练,K-BERT学会了计算化学家如何手动提取原子和分子特征。在K-BERT中transformer blocks的最后一层可以用作分子/原子的表示。计算由同一分子的不同smile生成的K-BERT WCL和K-BERT嵌入的相似性;
50个未出现在预训练数据集中的分子被用来评估同一分子的不同smile嵌入的相似性。对于每个分子,使用RDKit在规范SMILES的基础上生成另外4个不同的SMILES,然后使用K-BERT -WCL和K-BERT生成相应的embedding。采用Tanimoto系数的非二元形式来评价4种不同SMILES与规范SMILES之间的相似性。Tanimoto系数越接近1,不同SMILES的embedding越接近。
同一分子的SMILES非常高,因此重复训练这些分子给模型带来的信息很少。这些结果进一步证实了为什么SMILES排列的数据增强在K-BERT-WCL上无效。通过预训练,模型对SMILES有了很好的理解,知道如何从SMILES中提取特征。此外,K-BERT的平均embedding Tanimoto系数为0.994,说明对比学习预训练任务使KBERT能够更好地“理解”SMILES,区分同一分子的不同SMILES串。
如果模型能够“理解”SMILES,则同一原子的标记在同一分子的不同SMILES中对应的嵌入应该是相似的,以一个不在预训练数据集中的分子‘C=CCC(O)CC(C)(C)C’为例,生成了10个不同的SMILES。不同化学环境中的原子标记用不同的颜色标记,由K-BERT-WCL和K-BERT生成不同原子令牌的嵌入,并用t-SNE可视化:
4、K-BERT -FP can capture the size features of molecules that MACCS cannot capture
K-BERT -FP可以看作是一种新的指纹,K-BERT -FP在15个药物发现相关数据集上的性能与MACCS指纹相似,分子指纹的另一个重要功能是绘制化学空间。为了说明K-BERT -FP作为分子指纹图谱在化学空间中的映射能力,以化学空间树图(TMAP)的形式可视化了DrugBank中的分子,K-BERT -FP和MACCS在组织DrugBank数据集方面的表现都相当不错:
为了说明K-BERT -FP可以捕获MACCS不能捕获的分子大小信息,建立一个包含类似子结构但分子大小不同的分子数据集:
从甲烷、甲醇、甲胺、甲醛和甲酸中生成了7个初始片段,并选取了7个简单重复片段。然后,在初始片段的基础上,循环添加20个不同的重复组,生成一个具有相似子结构的数据集(Sim-Sub-Dataset),用XGBoost评估了K-BERT -FP和MACCS在Sim-Sub-Dataset上预测分子量的建模能力:
5、K-BERT -FP can capture chirality features through further pre-training
K-BERT还可以带来的是,可以根据特定的任务设计定制的分子指纹。通过改变预训练分子任务和在特定数据集中进行进一步的预训练,K-BERT可以生成包含需要的分子特征的定制K-BERT -FP。
为了说明macs不能处理手性问题,首先从CHIRAL1中选取2500个手性异构体(每个分子只有一个手性中心),并基于MACCS和K-BERT -FP以TMAP的形式可视化了它们的化学空间。
如图7A和B所示,MACCS和K-BERT -FP都不能处理手性问题。为了生成一个可以处理手性问题的自定义K-BERT -FP,进一步在CHIRAL1数据集上预训练K-BERT。使用了不同的训练前分子任务。一个是预测MACCS指纹图谱,另一个是预测分子的手性。分别生成两个模型,并用这两个模型生成的自定义KBERT -FPs (K-BERT -FP-CHIRAL1和K-BERT -FP-CHIRAL1R-S)对2500分子手性异构体的化学空间进行可视化。如图7C和D所示,K-BERT -FP-CHIRAL1R-S可以根据手性组织数据集,而K-BERT -FP-CHIRAK1不能,这说明需要使用特定的预训练任务来提取分子中的特定信息。经过进一步的预训练,该模型仍然可以根据一定的规则来区分分子,而不仅仅是基于R/S手性。如图7D所示,“R”分子聚集成一簇的立体异构体可以在另一簇中找到。