收藏|北大生信平台"单细胞分析、染色质分析"视频和PPT分享
生物信息学习的正确姿势
NGS系列文章包括NGS基础、在线绘图、转录组分析 (Nature重磅综述|关于RNA-seq你想知道的全在这)、ChIP-seq分析 (ChIP-seq基本分析流程)、单细胞测序分析 (重磅综述:三万字长文读懂单细胞RNA测序分析的最佳实践教程)、DNA甲基化分析、重测序分析、GEO数据挖掘(典型医学设计实验GEO数据分析 (step-by-step))、批次效应处理等内容。
北京大学生物信息平台在2018年的暑假和秋季组织了两期关于单细胞分析、染色质分析的研讨班,报告人来自于邓兴旺老师组、李程老师组、汤富酬老师组、谢晓亮老师组、张学工老师组、张泽民老师组。内容涉及单细胞分析的基本生物学问题、聚类、可视化、细胞图谱、发育图谱和多组学分析,染色质可及性、三维结构等。授权生信宝典发表出来,让更多朋友可以参与。
视频和PPT在https://space.bilibili.com/16813275/#/和http://202.205.131.32/forum/upload/forum.php?mod=viewthread&tid=324&extra=page%3D1,后台回复 北京大学生物信息平台获取可点击的链接。
秋季讨论班内容
描述为本人根据PPT和自己的理解所注,有不当处为本人而非演讲人责任,具体可听视频学习。
I. 单细胞分析模块
单细胞分析的生物学问题:
The biology of single-cell genomics,9月17日,主讲人:王龙腾(李程组)https://share.weiyun.com/5LgmWyO,(没有回放视频)龙腾一定是《工作细胞》的忠实粉丝,从这个萌化了的日本动漫入手引出人体大约有37兆2千亿细胞。之前流传一个说法,人体内的细菌比人的细胞多,在2006年的cell文章对此作了详细阐述,人体内细菌数目与人的细胞数目在同一数量级,但质量小的太多。一个70 kg的成年人,细胞重量在46 kg,细菌重量只有0.2 kg。
有点发散了,后面展开讲受精卵卵裂形成
ICM、胚胎干细胞、多能性干细胞、各个谱系进一步分化,以此作为铺垫, 既包含细胞之间的共性(可被常规bulk测序捕获),又展示细胞类型的丰富和异质性,这就引出了单细胞测序的优势。可以进行细胞分型、发育谱系构建、移植前遗传筛选等基础和临床的应用。单细胞多组学可以从染色质构想、结构层面解析调控的异质性。展望一下,超高分辨率成像也许可以所见既所得的解决这个“被高中生物骗了这么多年,原来人体内细胞的DNA是有不同的?”的问题。另外一个没提到的问题,单细胞测序细胞量大,尤其是来源于不同的个体或取样跨度时间长时,一定注意批次效应的影响,李程老师的
ComBat是GEO数据分析批次校正的常用工具。DESeq2差异基因分析和批次效应移除中也有介绍。scRNA-seq data visualization and clustering,10月8日,主讲人:陈文昌 邹子恒(清华大学张学工组)链接:https://share.weiyun.com/5TgOgDM,(视频地址:https://www.bilibili.com/video/av33441560)
文昌兄主要讲述了单细胞分析常用的降维算法
tSNE的原理和应用。PCA的线性降维我们比较熟悉了,把线性相关的变量转换为线性无关的向量。其操作方式是对原始矩阵的协方差矩阵进行对角化处理获得协方差矩阵为对角阵的一组新变量,即实现了线性降维。线性降维算法的一个主要问题是它们集中将不相似的数据点放置在较低维度区域时,数据点相距甚远。但是为了在低维、非线性流型上表示高维数据,我们也需要把相似的数据点靠近在一起展示,这并不是线性降维算法所能做的。
tSNE把点的高维空间的距离转换成点的相似度的概率,维持高维空间和低维空间中一对点之间的条件概率差值总和最小。同时使用t-分布的长尾性解决高维数据映射到低维时的重叠问题。t-SNE算法定义了数据的局部和全局结构之间的软边界,既可以使点在局部分散,又在全局聚集,同时照顾近距离和远距离的点。其性能优于任何非线性降维算法。具体实现见还在用PCA降维?快学学大牛最爱的t-SNE算法吧, 附Python/R代码。聚类算法有K-means (相似的SOM自组织特征图聚类)、
SC3 (一致聚类),Seurat (图聚类)、迭代聚类如层级聚类等。聚类的难点还在于其稳定性和聚类数目的确定,silhouette index是一个度量指标。单细胞分析与细胞图谱绘制,10月15日,主讲人:石强(李程组), 链接:https://share.weiyun.com/5kljVnj
以骨关节炎文章为例,展示了单细胞转录组的分析过程。从PCA结果看,细胞整体分型不明显,而且两个主成分的贡献率之和不足14%。但细胞在
PC2维度与骨关节炎的各个阶段有些吻合,随后选择与PC2正贡献和负贡献的基因进行可视化展示 (一维的可视化效果优于二维展示)。随后是
tSNE分型和Pseudospace发育轨迹分析、Marker鉴定等。可视化主要以热图、散点图、箱线图为主。功能分析主要是GSEA基因集富集分析。具体画图还是见高颜值免费在线绘图。单细胞分析与发育,10月29日,主讲人:李莉(汤富酬组),链接:链接:https://share.weiyun.com/5p7I1Nd
Cell Stem Cell文章 Single-Cell RNA-Seq Analysis Maps Development of Human Germline Cells and Gonadal Niche Interactions的一作讲坛。单细胞的分析都是套路分析,关键在于样品获取、实验的稳定性和结果的生物意义解读(乔院士和汤老师强强联合)。本文主要围绕胚胎生殖细胞发育过程讲述单细胞转录组、甲基化组和染色质可及性的分子调控。落脚点是调控网络和信号通路。生信老司机以中心法则为主线讲解组学技术的应用和生信分析心得 可以给生信的学习之路带来一些新的想法。单细胞多组学,11月5日,主讲人:张倩(李程组),链接:https://share.weiyun.com/5p7I1Nd
同一个细胞,不同的调控。单细胞多组学有
sci-CAR同时检测基因表达和染色质可及性,scNMT-seq同时检测染色质可及性、DNA甲基化和基因表达,connect-seq谱系追踪和转录组,在神经研究中应用较多。近来看到一篇追踪iPS重编程的工作,很有意思,回头解读下。这个视频重点在单细胞ATAC-seq技术的原理、应用和分析,有一部分Cicero的R代码实战。10X官网有不少视频,是了解单细胞技术的好的素材。基因组浏览器是展示多组学信号的合适工具。
II. 染色质分析模块
3D genome and its disorganization in diseases,11月19日,主讲人:李瑞风(李程组)链接:https://share.weiyun.com/5p7I1Nd
通常人类细胞核大小为
6 um,内有抻开后长度为3.0 m的46条染色体(3亿碱基对,每个碱基对高度是0.34 nm)。想象一下你如何能把3米的绳子放在6 um的小球中,更别说DNA上还有组蛋白的结合。染色体在细胞核内高度压缩缠绕,各自占有自己的一部分空间称为
chromosome territories。早期通过FISH技术鉴定,现在有Hi-C、ChIA-PET、OCEAN-C(李程老师组2018年发表,可同时检测TAD,compartment和开放染色质区域)等。3D基因组的3个层面:Compartment(Compartment A: 开放的活性区域,Compartment B: 致密的沉默区域, 10 Mb尺度),TAD(拓扑结构域,1 Mb尺度,不同物种和细胞系之间相对保守,与常见组蛋白修饰、CTCF结合关联较好),Loops(300 kb尺度,介导enhancer-promoter互作等, 细胞系之间变化较大)。后面是一作讲坛,结合李程老师组
Nature communication发表的关于多发性骨肉瘤的3D结构失调展示TAD与CNV的关系。3D基因组应用的一个例子还是趾的发育,这是增强子功能的经典案例,也是Cell和媒体的常客。ATAC-seq,分析流程与案例,11月26日,主讲人:孙林华(邓兴旺 钱伟强组)链接:https://share.weiyun.com/5p7I1Nd
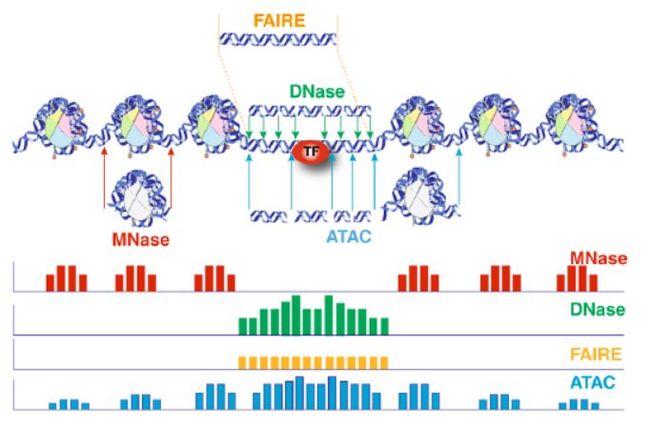
ATAC-seq全称
Assay for Transposase-Accessible chromatin用于检测开放的染色质区域,其基本操作是用Transposase Tn5切割DNA并在切点处连上接头序列,供下游分离扩增测序。实验难度低于ChIP系列的富集测序,结果具有广谱性,既可以展示核小体定位 (测序片段中间部分),也可以研究开放染色质区域(测序片段两端部分)。配合转录因子结合Motif分析可在一定程度起到替代ChIP-seq的作用,尤其是抗体缺失的情况下。这个报告讲述了ATAC-seq的原理、可用的生信分析工具、在动植物分子调控研究中的应用。ATAC-seq基本分析与ChIP类似,只是根据关注的重点确定参数的选择。
Introduction to Hi-C experiment and data analysis,12月3日,主讲人:李梦帆(李程组)链接:https://share.weiyun.com/5p7I1Nd
Hi-C实验和分析的系统介绍,比较详细,可以作为学习的起点。测序成本的下降,给Hi-C提供了很好的契机,测序一次浪费一半以上的
reads,背后都是钱啊。不过基于Hi-C的拓展应用如染色体组装、单体型鉴定都提供了好的应用场景。基于成像的染色质分析,12月10日,主讲人:侯英萍(李程组)链接:https://share.weiyun.com/5p7I1Nd
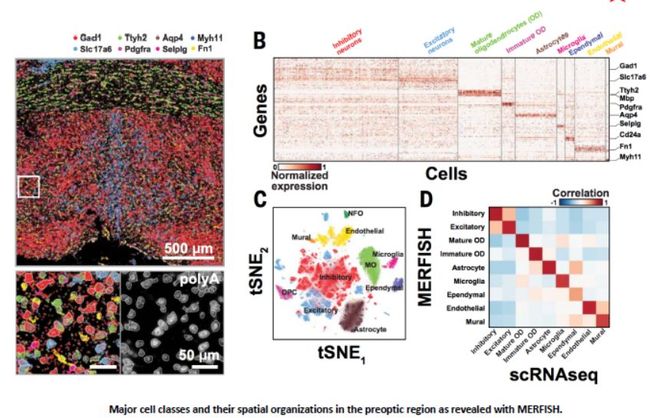
基于FISH (Fluoresence in situ hybridization)的原位
DNA结构鉴定和RNA表达的原位细胞类型鉴定。配合超高分辨率显微镜加精巧的实验设计,也可以一定程度的实现高通量的所见即所得。MERFISH是著名华人学者庄小威教授2015年开发的大规模RNA成像技术,根据不同细胞类型表达基因的不同在原位器官鉴定细胞类型的空间分布。染色质分析与多组学整合,12月17日,主讲人:刘玉婷(李程组) 链接:https://share.weiyun.com/5p7I1Nd
多组学整合分析本质是把不同组学鉴定出来的元件根据特征细分、分类后做图谱、做交集 (Nature综述|整合组学分析护航健康,推动精准医学时代的到来!)。其研究要么起始于文献,要么起始于对数据的观察,这里面还是基因组浏览器的作用不可忽视。观察到基本模式后,就是去写程序检测基本模式是否具有普遍性和显著性,这部分没有成熟的工具,工具组合可以提供一些帮助,最主要的是自己学些编程了。(回复生信宝典福利第一波获取我们自写的Python、Linux、R学习教程)
染色质分析中的新概念、新方法和新工具,12月24日,主讲人:贾璐萌(李程组)链接:https://share.weiyun.com/5p7I1Nd
一作解读,
OCCAN-C, 李程老师组2018年发表,可同时检测TAD,compartment和开放染色质区域。
III. 网上资源:
单细胞分析课程:http://hemberg-lab.github.io/scRNA.seq.course/index.html (你也可以用bookdown整理出这样漂亮的教程)
单细胞分析软件集成:https://github.com/seandavi/awesome-single-cell
染色质与三维基因组分析课程:https://github.com/hms-dbmi/3d-genome-processing-tutorial, https://github.com/hms-dbmi/hic-data-analysis-bootcamp
染色质分析相关软件:https://github.com/dekkerlab,https://github.com/theaidenlab/juicer,https://github.com/nservant/HiC-Pro,https://bioconductor.org/packages/release/bioc/html/HiTC.html
暑期讨论班内容
单细胞实验与分析(2018.6-8月,这部分没有配套视频)
An introduction to bulk and single-cell RNA-seq:2018年6月25日,主讲人:徐子晗(李程组) 链接:https://share.weiyun.com/5dLF79v
The advances in scRNA-seq techniques: 2018年7月2日,主讲人:李莉(汤富酬组) 链接:https://share.weiyun.com/59ryjRX
Pre-processing and variation of scRNA-seq data:2018年7月9日,主讲人:石强(李程组) 链接:https://share.weiyun.com/5bGebsN 介绍了基本单细胞分析流程和常用工具,主要讲述了噪音的来源和校正方法。单细胞前期的分析跟bulk测序还是一致的,工具也有通用性,39个转录组分析工具,120种组合评估(转录组分析工具哪家强-导读版)适合作为选择依据。
scRNA-seq data analysis: Imputation, Cluster, and differential expression: 2018年7月16日,主讲人:李子逸(张泽民组)链接:https://share.weiyun.com/5dN7wuZ 单细胞分析流程概述,cellranger的使用。即使在Windows下分析,也不要用Excel,以免Excel改变了你的基因名,30% 相关Nature文章受影响,NCBI也受波及。
scRNA-seq data analysis: single cell trajectory:2018年7月23日,主讲人:张倩(李程组)链接:https://share.weiyun.com/5uXwz15
From single-cell RNA-seq to transcription network:2018年7月30日,主讲人:李响(谢晓亮组)链接:https://share.weiyun.com/5ys73Yp。主要讲述了
WGCNA加权基因共表达网络的应用。定制单细胞扩增与泛组学测序:2018年8月13日,主讲人:宋立阳(谢晓亮组)链接:https://share.weiyun.com/5TMLNAk (单细胞扩增技术的比较)
系列教程
生物信息之程序学习
关于编程学习的一些思考
该如何自学入门生物信息学
如何优雅的提问
生信宝典视频教程
好色之旅-画图三字经
转录组分析的正确姿势
生信的系列教程
生信的系列书籍
文章用图的修改和排版 (1)
文章用图的修改和排版 (2)
简单强大的在线绘图
简单强大的在线绘图-升级版
简单强大的在线绘图-第3版
论文图表基本规范
学术图表的基本配色方法
数据可视化基本套路总结
英语写作常见错误总结和学习视频
教育部推出首批490门”国家精品在线开放课程”
你该知道的杂志分区和影响因子及最新表格下载
你和PPT高手之间,就只差一个iSlide
推荐 3 个超赞的 EXCEL 插件,让你 5 分钟从小白变大神
史上最全的图表色彩运用原理
生信宝典一周年福利第一波 - 电子书赠送
测序发展史:150年的风雨历程
生信老司机以中心法则为主线讲解组学技术的应用和生信分析心得
别再让投稿信耽误你发稿啦!(附cover letter模板)
生物信息学数据库分类概览 (第一版)
跟Cell editor学做scientific presentation (25个判断标准,18个不能做,8个建议)
往期精品(点击图片直达文字对应教程)
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
![]()
![]()
![]()