R语言自学笔记:数据处理、数据分布及数据检验
一. 基本的数据统计处理
1. 数据的展示
对于一组数据,可以有许多统计分析的方法。
(1) 基本信息
mean():求平均值
sum():求和
sd():求总体标准差——(样本-期望)/(样本数)
var():求样本标准差——(样本-期望)/(样本数-1)
range():求样本数据的范围
(2) 数据展示:
summary():展现一组数据的综合信息,返回一个list
> summary(mtcars$wt)
Min. 1st Qu. Median Mean 3rd Qu. Max.
1.513 2.581 3.325 3.217 3.610 5.424 quantile():根据遗嘱数据来分配概率,最小数概率对应0,最大数概率对应1
> quantile(mtcars$wt)
0% 25% 50% 75% 100%
1.51300 2.58125 3.32500 3.61000 5.42400 table():返回一组数据中,每个特异值出现的次数
> table(mtcars$cyl)
4 6 8
11 7 14 2. 数据的分组
等距和等大小分组:
mtcars1 <- mtcars %>% mutate(equal_size = ntile( qsec , 5),
equal_distance = cut( wt,
breaks = seq( from = min(wt), to = max(wt), ##设置断开的位置
by = (max(wt) - min(wt)) / 5 ), ##通过计算步长,层层递进得到最终的序列
include.lowest = T ))ntile函数通过将数据进行排序,更具分组需要,将数据分为n个组别,返回这组数据对应位置上的数据的组别;等距分组则完全依赖于将数字区间进行军方,利用cut函数将落到不同区间里的数据进行划分,二者的划分结果实质相同;
3. 分析数据的异常值
对于已知总体分布的数据常用 mean ± n*sd (n = 2,3...) 来限定样本中合理值范围,超过这些范围的可认作为异常值;对于未知参数的样本数据,有许多排除一场值的方法,一个经典的方法就是上、下四分位分别加减1.5倍的四分位范围从而限定数的上下限,在此之外的均为一异常值:
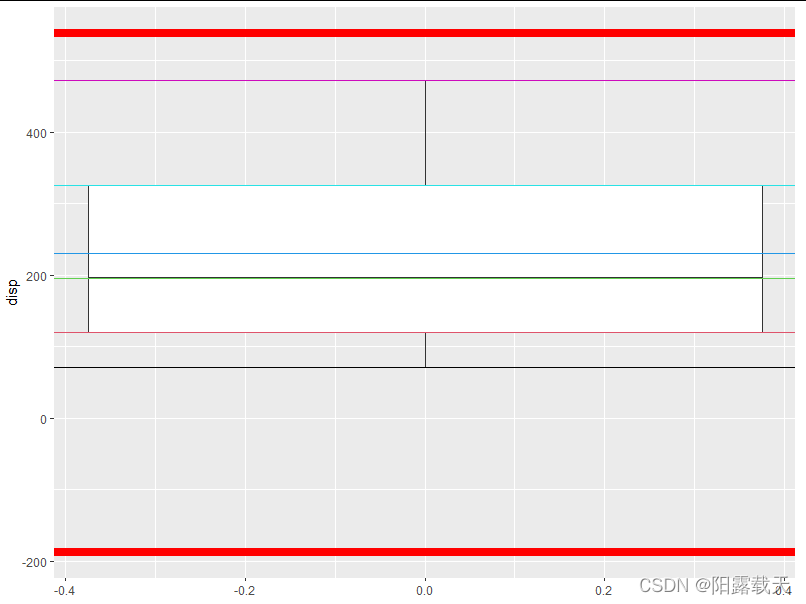
(当然在ggplot的geom_box当中,这个范围已经被默认,图中被单独列出的黑点即为异常值,此时不会有正常箱图里表示数据上下界的横线)
##先查看disp的四分位
> s <- summary( mtcars$disp)
> s
Min. 1st Qu. Median Mean 3rd Qu. Max.
71.1 120.8 196.3 230.7 326.0 472.0
##得到iqr(四分位的范围)的值
> iqr <- IQR(mtcars$disp)
> iqr
[1] 205.175
##绘图
ggplot(mtcars,aes(y = disp)) +
geom_boxplot() +
geom_hline(yintercept = s, color = 1:6) + ##根据summary里面的几个值,在箱图上标出对应的线
geom_hline(yintercept = c(s[2]-1.5*iqr , s[4]+1.5*iqr), color = 'red',size = 3) ##标出上下限就
可见数据中无异常值。
二. 数据分布
R语言内部可以依据不同分布生成随机数据,同时也可以对一列数据按照预设的分布推算其出现概率等。
1. 生成随机数
(1) rnorm()
依据随机分布生成一系列随机数:
rnorm(55, mean = 0, sd = 1)
##生成55个平均值为0,标准差为1的随机数(2) runif / sample
生成服从于均匀分布的随机数:
> runif(17,max = 1 ,min = 0.5)
[1] 0.7074430 0.9890402 0.9877037 0.7021419 0.5685381 0.6919902 0.7986621
[8] 0.9996719 0.9699007 0.5763348 0.6055292 0.9353894 0.8633643 0.5103040
[15] 0.8058729 0.8509983 0.6263499
##生成17个最大值为1,最小值为0.5,默认是0~1
> sample(0:10, 15, replace = TRUE)
[1] 4 7 8 2 0 2 6 9 1 2 7 9 0 4 6
##replace代表是否允许放回采样(3) rbinom
根据事件发生的概率,对一定次数次实验结果进行估计,生成事件发生的次数的序列:
rbi <- rbinom(23, size = 100, prob = 0.5)
[1] 45 60 49 44 51 45 56 39 42 50 54 54 43 58 34 52 50 51 48 39 58 48 52
##产生23个数据,其中进行100次实验,事件发生概率为0.5,生成的数字是这100次事件中事件发生的概率(4) rpois
根据指定的lamda来生成数据:
> pos <- rpois(33 , 4)
> pos
[1] 5 5 4 2 8 7 6 10 2 2 3 4 8 6 8 5 5 8 4 5 3 4 4
[24] 8 5 4 6 4 6 5 4 4 1(5) rexp
指定指数,产生数列:
> rexp(11,0.5)
[1] 0.2354817 4.2218206 1.2789491 0.5667985 6.8250768 11.7577884
[7] 3.4183153 0.2926459 0.6410425 3.0253369 0.7224791(6) rgamma
指定shape参数,产生数列:
> rgamma(9 , shape = 4)
[1] 4.818401 3.816967 3.380444 2.770275 4.406524 3.578493 2.511661
[8] 4.838542 3.382133
##scale默认为1当参数 shape = 1时,伽马分布就是参数为scale的指数分布
当shape = n/2,scale = 1/2 时,伽马分布就是自由度为n的卡方分布
2. 数据的分布的分析
以上函数均由 r_<...>组成,将“r”替换分别为p、q和d,构成依照某种分布来分析数据的概率特征的函数
以正态分布为例,生成函数rnorm()
# q: 一系列下分位点(可以理解是概率一个密度函数曲线对应的x轴上的点)。函数计算计算P[x < q]
pnorm(q, mean = 0, sd = 1)
# p:系列概率(所以要求p在 0~1 内)。函数通过概率反推计算“x轴上坐标点的位置”
qnorm(p, mean = 0, sd = 1)
# x:通过x轴上坐标点计算该处的概率密度
dnorm(x, mean = 0, sd = 1)三. 数据的统计检验
1. 数据的关联性分析
可以使用cor.test函数对两组数据的关联性进行分析:
> r <- with( mtcars, cor.test( mpg, wt ))
> r
Pearson's product-moment correlation
data: mpg and wt
t = -9.559, df = 30, p-value = 1.294e-10
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
-0.9338264 -0.7440872
sample estimates:
cor
-0.8676594
## true correlation is not equal to 0 表明二者有一定相关性
## cor是关联性系数,在例子中,表面wt和mpg二者有呈现负相关的趋势
## 使用 r$ 来查看r中不同数据,说明函数返回的还是一个列表2. 参数和非参数检验
参数检验和非参数检验的差别就在于是否关注于总体的分布情况
(1) student 检验:
使用t.test对数据进行检验,其中输入的数据的形式可分为3种,分别对应不同的检验目标
t.test(数据向量, mu = number) :分析number是否区别于这组数据,即number这个数值在数据中是否协调;
t.test(数据向量1,数据向量2):分析两个数据的分布差异是否显著。注意可以使用 paired = T(默认为F)参数,表明对两个数向量中的数据要一一对应着进行分析;
> t.test(mtcars$mpg,mtcars$wt,paried = T)
Welch Two Sample t-test
data: mtcars$mpg and mtcars$wt
t = 15.633, df = 32.633, p-value < 2.2e-16
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
14.67644 19.07031
sample estimates:
mean of x mean of y
20.09062 3.21725 t.test(类别1 ~ 类别2):类别可以是一个df中的某两列,其中一组数据levels只能由两种,分析关联性,例如分析性别(levels = 男或女)与寿命的关系
> with(mtcars,t.test(mpg,vs)) ##单纯两组数据之间的比较
Welch Two Sample t-test
data: mpg and vs
t = 18.382, df = 31.434, p-value < 2.2e-16
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
17.47381 21.83244
sample estimates:
mean of x mean of y
20.09062 0.43750
> with(mtcars,t.test(mpg~vs)) ##关联性的分析,vs的level只有“0” “1”
Welch Two Sample t-test
data: mpg by vs
t = -4.6671, df = 22.716, p-value = 0.0001098
alternative hypothesis: true difference in means between group 0 and group 1 is not equal to 0
95 percent confidence interval:
-11.462508 -4.418445
sample estimates:
mean in group 0 mean in group 1
16.61667 24.55714(2) wilcox.test()
释义:符号秩检验(Wilcoxon);基于符号检验其秩分分布的办法,本质上是一种检验平均秩的检验。即把样本的两次观测值相减,记录差值的符号和绝对值,并基于绝对值升序求秩分,比较两组数据的正值秩分或负值秩分,从而确定其差异性。
非参数检验的一种,用法和t.test()基本语法一样,两种检验所得p值可以不同。
四. 线性回归及预测
1. lm()
用于线性拟合:
lm(formula, data, subset, weights, na.action,
method = "qr", model = TRUE, x = FALSE, y = FALSE, qr = TRUE,
singular.ok = TRUE, contrasts = NULL, offset, ...)对于一般的使用而言,输入合适的data和formula即可使用该函数。data为一个df,限定了formula中各个量在df中的对应关系(变量之间的关系不是随意的,而是由df中实实在在的一行中的各列间的关系相互对应起来的),formula输入待拟合的变量之间关系:
> lm.model <- lm(wt ~ mpg + hp, data = mtcars)
> lm.model
Call:
lm(formula = wt ~ mpg + hp, data = mtcars)
Coefficients:
(Intercept) mpg hp
6.2182863 -0.1455218 -0.0005277
## 注意这里相当于构建了一个 wt = a*mpg + b*hp 的一个函数,a和b均为系数2. 线性模型的预测
利用lm生成的模型对数据进行预测,可以提取模型的参数后,生成线性模型随后对其进行预测,当让也可以使用predict() 函数:
para <- coef(lm.model) ##利用coef()函数提取模型里面的参数,返回一个向量方便使用;
lm.model <- lm(wt ~ mpg + hp, data = mtcars) ##加载模型
newdata <- mtcars %>% dplyr::select(hp, mpg ) ##提取原有的数据
wt.predicted <- predict( lm.modelplot, newdata ) ##利用原来的数据,结合模型,对wt进行预估
plot <-
ggplot( data.frame( reference = mtcars$wt, manual = wt.predicted ), ##将原wt和预测的wt分别作为两轴
aes( reference, manual ) ) +
geom_point() +
geom_smooth( method = "lm", se = F ); ##geom_smooth自适应地对x轴和y轴上的元素,以lm规则进行拟合
可见,预测数据与原有数据仍有差距,geom_smooth拟合的曲线并非 y = x 而有所偏差。