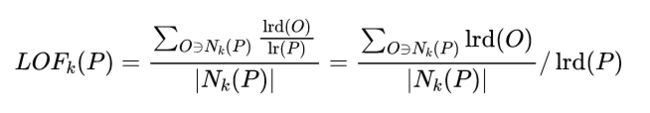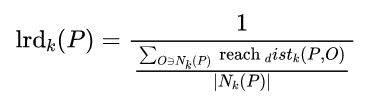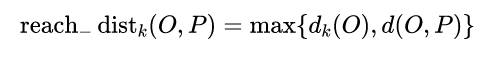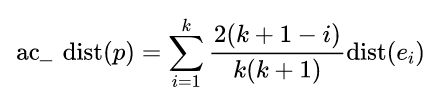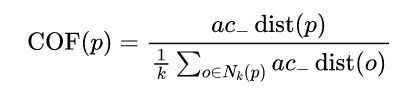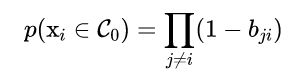异常检测方法总结
在数据挖掘中,异常检测(英语:anomaly detection)对不匹配预期模式或数据集中其他项目的项目、事件或观测值的识别。 通常异常项目会转变成银行欺诈、结构缺陷、医疗问题、文本错误等类型的问题。异常也被称为离群值、新奇、噪声、偏差和例外。
1. 异常检测的分类
目前比较公认的分类方式是分为三种:
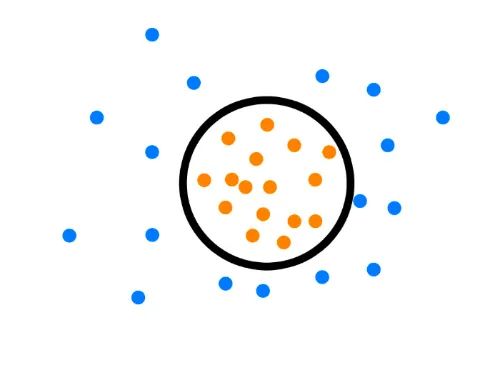
- 单点异常(Global Outliers):也可以称为全局异常,即某个点与全局大多数点都不一样,那么这个点构成了单点异常。例如,和三只小黄人相比,海绵宝宝的混入就可以算作是单点异常。
- 上下文异常(Contextual Outliers):这类异常多为时间序列数据中的异常,即某个时间点的表现与前后时间段内存在较大的差异,那么该异常为一个上下文异常点。例如,在某个温带城市夏天的气温时序数据中,其中有一天温度为10℃,而前后的气温都在25-35℃的范围,那么这一天的气温就可以说是一个上下文异常。
- 集体异常(Collective Outliers):这类异常是由多个对象组合构成的,即单独看某个个体可能并不存在异常,但这些个体同时出现,则构成了一种异常。集体异常可能存在多种组成方式,可能是由若干个单点组成的,也可能由几个序列组成。想象这样一个场景,某小区某天有一户人家搬家了,这是一件很正常的事,但如果同一天有10户同时搬家了,那就构成了集体异常,显然这不是一个正常小区会时常发生的事情。
2. 在异常检测中经常遇到哪些困难?
- 在大多数实际的场景中,数据本身是没有标签的,也存在一些数据集有标签,但标签的可信度非常低,导致放入模型后效果很差,这就导致我们无法直接使用一些成熟的有监督学习方法。
- 常常存在噪音和异常点混杂在一起的情况,难以区分。
- 在一些欺诈检测的场景中,多种诈骗数据都混在一起,很难区分不同类型的诈骗,因为我们也不了解每种诈骗的具体定义。
由于没有准确的标签,也没有对具体诈骗类型的理解,就导致我们陷入鸡生蛋 or 蛋生鸡的循环之中。要解决这种情况,目前比较常用的手段是,将无监督学习方法和专家经验相结合,基于无监督学习得到检测结果,并让领域专家基于检测结果给出反馈,以便于我们及时调整模型,反复进行迭代,最终得到一个越来越准确的模型。
到这里,我们都发现了,很多风控场景中最重要的一环就是:专家经验。
3. 异常检测算法的分类
异常检测涉及的场景非常丰富,那么异常检测算法可以从哪些角度进行分类呢?一般可以从以下四个角度作区分:
- 时序相关 VS 时序独立
- 全局检测 VS 局部检测
- 输出形式:标签 VS 异常分数
- 根据不同的模型特征
3.1. 时序相关 VS 时序独立
首先可以根据该场景的异常是否与时间维度相关。在时序相关问题中,我们假设异常的发生与时间的变化相关,比如一个人平时的信用卡消费约为每月5000元,但11月的消费达到了10000元,那这种异常的出现就明显与时间维度相关,可能是因为”万恶“的双十一。
而在时序独立问题中,我们假设时间的变化对异常是否发生是无影响的,在后续的分析建模中,也就不会代入时间维度。
3.2. 全局检测 VS 局部检测
在全局检测方法中,针对每个点进行检测时,是以其余全部点作为参考对象的,其基本假设是正常点的分布很集中,而异常点分布在离集中区域较远的地方。这类方法的缺点是,在针对每个点进行检测时,其他的异常点也在参考集内,这可能会导致结果可能存在一定偏差。
而局部检测方法仅以部分点组成的子集作为参考对象,基于的假设是,正常点中可能存在多种不同的模式,与这些模式均不同的少数点为异常点。该类方法在使用过程中的缺点是,参考子集的选取比较困难。
3.3. 输出形式:标签 VS 异常分数
这种分类方式是根据模型的输出形式,即直接输出标签还是异常分数。输出标签的方法比较简单直观,可以直接根据模型输出的结果判断每个点是否为异常点。
使用可以输出异常得分的模型,我们可以进一步看哪些点的异常程度更高,也可以根据需要设定阈值,比如设定百分比,找出异常程度排在前10%的异常点。
3.4. 根据不同的模型特征
最后,还可以根据模型本身的特点进行分类,大致可以分为以下几种:
- 统计检验方法
- 基于深度的方法
- 基于偏差的方法
- 基于距离的方法
- 基于密度的方法
- 深度学习方法
如今用于异常检测的算法已经非常多了,但万变不离其宗,无论优化到什么程度,它们都是由一些原始的模型和思想衍生出来的。下面来回顾一下这些最原始的模型思想。
4. 基于分布的方法
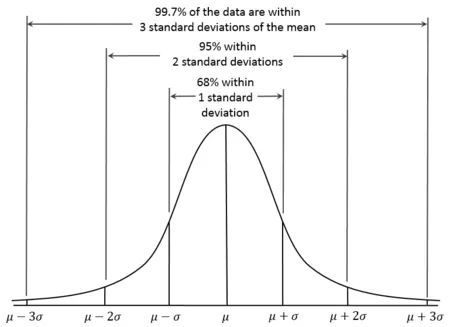
4.1. 3sigma
基于正态分布,3sigma准则认为超过3sigma的数据为异常点。
def three_sigma(s):
mu, std = np.mean(s), np.std(s)
lower, upper = mu-3*std, mu+3*std
return lower, upper4.2. Z-score
Z-score为标准分数,测量数据点和平均值的距离,若A与平均值相差2个标准差,Z-score为2。当把Z-score=3作为阈值去剔除异常点时,便相当于3sigma。
def z_score(s):
z_score = (s - np.mean(s)) / np.std(s)
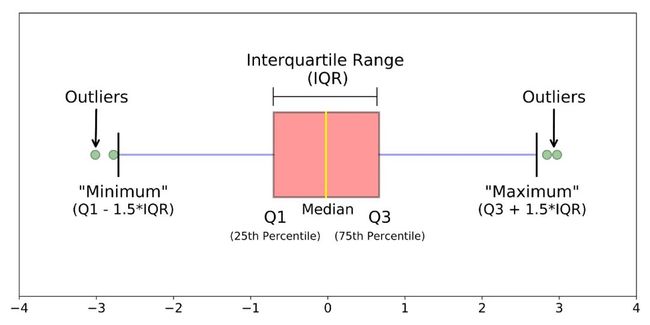
return z_score4.3. boxplot
箱线图时基于四分位距(IQR)找异常点的。
def boxplot(s):
q1, q3 = s.quantile(.25), s.quantile(.75)
iqr = q3 - q1
lower, upper = q1 - 1.5*iqr, q3 + 1.5*iqr
return lower, upper4.4. Grubbs假设检验
Grubbs’Test为一种假设检验的方法,常被用来检验服从正态分布的单变量数据集(univariate data set)Y中的单个异常值。若有异常值,则其必为数据集中的最大值或最小值。原假设与备择假设如下:
-
H0: 数据集中没有异常值
-
H1: 数据集中有一个异常值
使用Grubbs测试需要总体是正态分布的。算法流程:
- 样本从小到大排序
- 求样本的mean和dev
- 计算min/max与mean的差距,更大的那个为可疑值
- 求可疑值的z-score (standard score),如果大于Grubbs临界值,那么就是outlier
Grubbs临界值可以查表得到,它由两个值决定:检出水平α(越严格越小),样本数量n,排除outlier,对剩余序列循环做 1-4 步骤 [1]。详细计算样例可以参考。
from outliers import smirnov_grubbs as grubbs
print(grubbs.test([8, 9, 10, 1, 9], alpha=0.05))
print(grubbs.min_test_outliers([8, 9, 10, 1, 9], alpha=0.05))
print(grubbs.max_test_outliers([8, 9, 10, 1, 9], alpha=0.05))
print(grubbs.max_test_indices([8, 9, 10, 50, 9], alpha=0.05))局限:
- 只能检测单维度数据
- 无法精确的输出正常区间
- 它的判断机制是“逐一剔除”,所以每个异常值都要单独计算整个步骤,数据量大吃不消。
- 需假定数据服从正态分布或近正态分布
5. 基于距离的方法
KNN
依次计算每个样本点与它最近的K个样本的平均距离,再利用计算的距离与阈值进行比较,如果大于阈值,则认为是异常点。优点是不需要假设数据的分布,缺点是仅可以找出全局异常点,无法找到局部异常点。
from pyod.models.knn import KNN
# 初始化检测器clf
clf = KNN( method='mean', n_neighbors=3, )
clf.fit(X_train)
# 返回训练数据上的分类标签 (0: 正常值, 1: 异常值)
y_train_pred = clf.labels_
# 返回训练数据上的异常值 (分值越大越异常)
y_train_scores = clf.decision_scores_6. 基于密度的方法
6.1. Local Outlier Factor (LOF)
LOF是基于密度的经典算法(Breuning et. al. 2000),通过给每个数据点都分配一个依赖于邻域密度的离群因子 LOF,进而判断该数据点是否为离群点。它的好处在于可以量化每个数据点的异常程度(outlierness)。
数据点p的局部相对密度(局部异常因子)为点p邻域内点的平均局部可达密度跟数据点p的局部可达密度的比值, 即:
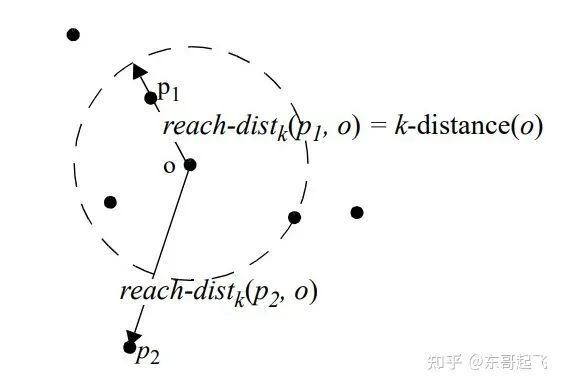
数据点P的局部可达密度=P最近邻的平均可达距离的倒数。距离越大,密度越小。
点P到点O的第k可达距离=max(点O的k近邻距离,点P到点O的距离)。
点O的k近邻距离=第k个最近的点跟点O之间的距离。
整体来说,LOF算法流程如下:
-
对于每个数据点,计算它与其他所有点的距离,并按从近到远排序;
-
对于每个数据点,找到它的K-Nearest-Neighbor,计算LOF得分。
from sklearn.neighbors import LocalOutlierFactor as LOF
X = [[-1.1], [0.2], [100.1], [0.3]]
clf = LOF(n_neighbors=2)
res = clf.fit_predict(X)
print(res)
print(clf.negative_outlier_factor_)6.2. Connectivity-Based Outlier Factor (COF)
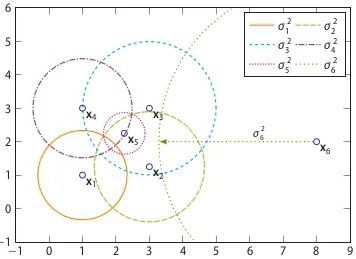
COF是LOF的变种,相比于LOF,COF可以处理低密度下的异常值,COF的局部密度是基于平均链式距离计算得到。在一开始的时候我们一样会先计算出每个点的k-nearest neighbor。而接下来我们会计算每个点的Set based nearest Path,如下图:
假使我们今天我们的k=5,所以F的neighbor为B、C、D、E、G。而对于F离他最近的点为E,所以SBN Path的第一个元素是F、第二个是E。离E最近的点为D所以第三个元素为D,接下来离D最近的点为C和G,所以第四和五个元素为C和G,最后离C最近的点为B,第六个元素为B。所以整个流程下来,F的SBN Path为{F, E, D, C, G, C, B}。而对于SBN Path所对应的距离e={e1, e2, e3,…,ek},依照上面的例子e={3,2,1,1,1}。
所以我们可以说假使我们想计算p点的SBN Path,我们只要直接计算p点和其neighbor所有点所构成的graph的minimum spanning tree,之后我们再以p点为起点执行shortest path算法,就可以得到我们的SBN Path。
而接下来我们有了SBN Path我们就会接着计算,p点的链式距离:
有了ac_distance后,我们就可以计算COF:
from pyod.models.cof import COF
cof = COF(contamination = 0.06, ## 异常值所占的比例
n_neighbors = 20, ## 近邻数量
)
cof_label = cof.fit_predict(iris.values) # 鸢尾花数据
print("检测出的异常值数量为:",np.sum(cof_label == 1))6.3. Stochastic Outlier Selection (SOS)
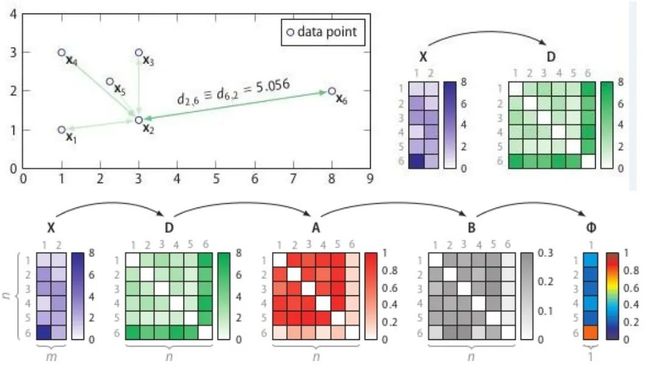
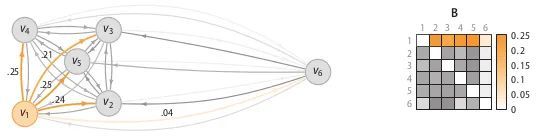
将特征矩阵(feature martrix)或者相异度矩阵(dissimilarity matrix)输入给SOS算法,会返回一个异常概率值向量(每个点对应一个)。SOS的思想是:当一个点和其它所有点的关联度(affinity)都很小的时候,它就是一个异常点。
SOS的流程:
- 计算相异度矩阵D;
- 计算关联度矩阵A;
- 计算关联概率矩阵B;
- 算出异常概率向量。
相异度矩阵D是各样本两两之间的度量距离, 比如欧式距离或汉明距离等。关联度矩阵反映的是 度量距离方差, 如图7, 点![]() 的密度最大, 方差最小;
的密度最大, 方差最小; ![]() 的密度最小, 方差最大。而关联概率矩阵B
的密度最小, 方差最大。而关联概率矩阵B
(binding probability matrix)就是把关联矩阵(affinity matrix)按行归一化得到的, 如图8所示。
得到了binding probability matrix,每个点的异常概率值就用如下的公式计算,当一个点和其它所有点的关联度(affinity)都很小的时候,它就是一个异常点。
import pandas as pd
from sksos import SOS
iris = pd.read_csv("http://bit.ly/iris-csv")
X = iris.drop("Name", axis=1).values
detector = SOS()
iris["score"] = detector.predict(X)
iris.sort_values("score", ascending=False).head(10)7. 基于聚类的方法
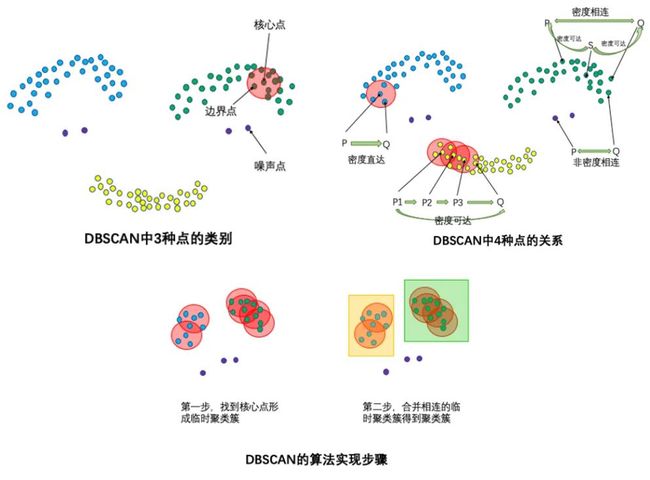
DBSCAN算法(Density-Based Spatial Clustering of Applications with Noise)的输入和输出如下,对于无法形成聚类簇的孤立点,即为异常点(噪声点)。
-
输入:数据集,邻域半径Eps,邻域中数据对象数目阈值MinPts;
-
输出:密度联通簇。
处理流程如下:
- 从数据集中任意选取一个数据对象点p;
- 如果对于参数Eps和MinPts,所选取的数据对象点p为核心点,则找出所有从p密度可达的数据对象点,形成一个簇;
- 如果选取的数据对象点 p 是边缘点,选取另一个数据对象点;
- 重复以上2、3步,直到所有点被处理。
from sklearn.cluster import DBSCAN
import numpy as np
X = np.array([[1, 2], [2, 2], [2, 3],
[8, 7], [8, 8], [25, 80]])
clustering = DBSCAN(eps=3, min_samples=2).fit(X)
clustering.labels_
array([ 0, 0, 0, 1, 1, -1])
# 0,,0,,0:表示前三个样本被分为了一个群
# 1, 1:中间两个被分为一个群
# -1:最后一个为异常点,不属于任何一个群8. 基于树的方法
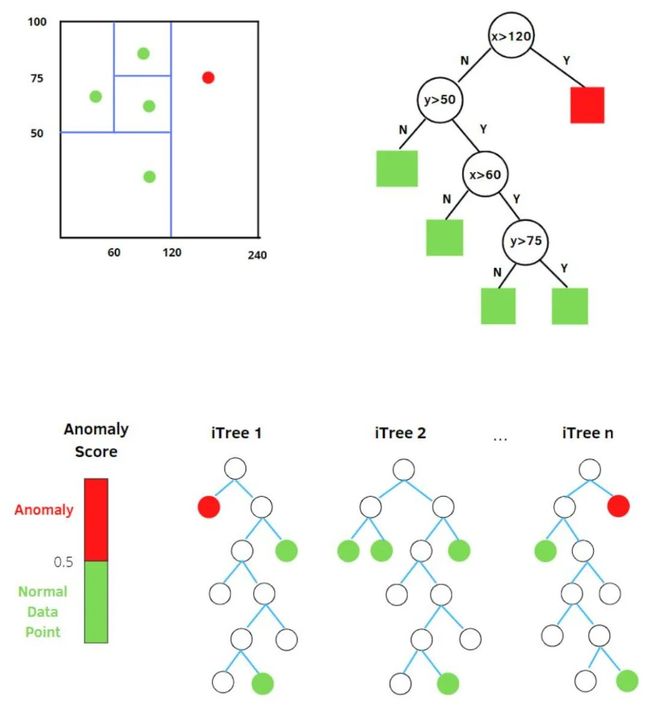
Isolation Forest (iForest)
孤立森林中的 “孤立” (isolation) 指的是 “把异常点从所有样本中孤立出来”,论文中的原文是 “separating an instance from the rest of the instances”。
我们用一个随机超平面对一个数据空间进行切割,切一次可以生成两个子空间。接下来,我们再继续随机选取超平面,来切割第一步得到的两个子空间,以此循环下去,直到每子空间里面只包含一个数据点为止。我们可以发现,那些密度很高的簇要被切很多次才会停止切割,即每个点都单独存在于一个子空间内,但那些分布稀疏的点,大都很早就停到一个子空间内了。所以,整个孤立森林的算法思想:异常样本更容易快速落入叶子结点或者说,异常样本在决策树上,距离根节点更近。
随机选择m个特征,通过在所选特征的最大值和最小值之间随机选择一个值来分割数据点。观察值的划分递归地重复,直到所有的观察值被孤立。
获得 t 个孤立树后,单棵树的训练就结束了。接下来就可以用生成的孤立树来评估测试数据了,即计算异常分数 s。对于每个样本 x,需要对其综合计算每棵树的结果,通过下面的公式计算异常得分:
from sklearn.datasets import load_iris
from sklearn.ensemble import IsolationForest
data = load_iris(as_frame=True)
X,y = data.data,data.target
df = data.frame
# 模型训练
iforest = IsolationForest(n_estimators=100, max_samples='auto',
contamination=0.05, max_features=4,
bootstrap=False, n_jobs=-1, random_state=1)
# fit_predict 函数 训练和预测一起 可以得到模型是否异常的判断,-1为异常,1为正常
df['label'] = iforest.fit_predict(X)
# 预测 decision_function 可以得出 异常评分
df['scores'] = iforest.decision_function(X)9. 基于降维的方法
9.1. Principal Component Analysis (PCA)
PCA在异常检测方面的做法,大体有两种思路:
(1) 将数据映射到低维特征空间,然后在特征空间不同维度上查看每个数据点跟其它数据的偏差;
(2) 将数据映射到低维特征空间,然后由低维特征空间重新映射回原空间,尝试用低维特征重构原始数据,看重构误差的大小。
PCA在做特征值分解,会得到:
- 特征向量:反应了原始数据方差变化程度的不同方向;
- 特征值:数据在对应方向上的方差大小。
得分大于阈值C则判断为异常。
第二种做法,PCA提取了数据的主要特征,如果一个数据样本不容易被重构出来,表示这个数据样本的特征跟整体数据样本的特征不一致,那么它显然就是一个异常的样本:
基于低维特征进行数据样本的重构时,舍弃了较小的特征值对应的特征向量方向上的信息。换一句话说,重构误差其实主要来自较小的特征值对应的特征向量方向上的信息。基于这个直观的理解,PCA在异常检测上的两种不同思路都会特别关注较小的特征值对应的特征向量。所以,我们说PCA在做异常检测时候的两种思路本质上是相似的,当然第一种方法还可以关注较大特征值对应的特征向量。
from sklearn.decomposition import PCA
pca = PCA()
pca.fit(centered_training_data)
transformed_data = pca.transform(training_data)
y = transformed_data
# 计算异常分数
lambdas = pca.singular_values_
M = ((y*y)/lambdas)
# 前k个特征向量和后r个特征向量
q = 5
print "Explained variance by first q terms: ", sum(pca.explained_variance_ratio_[:q])
q_values = list(pca.singular_values_ < .2)
r = q_values.index(True)
# 对每个样本点进行距离求和的计算
major_components = M[:,range(q)]
minor_components = M[:,range(r, len(features))]
major_components = np.sum(major_components, axis=1)
minor_components = np.sum(minor_components, axis=1)
# 人为设定c1、c2阈值
components = pd.DataFrame({'major_components': major_components,
'minor_components': minor_components})
c1 = components.quantile(0.99)['major_components']
c2 = components.quantile(0.99)['minor_components']
# 制作分类器
def classifier(major_components, minor_components):
major = major_components > c1
minor = minor_components > c2
return np.logical_or(major,minor)
results = classifier(major_components=major_components, minor_components=minor_components)9.2. AutoEncoder
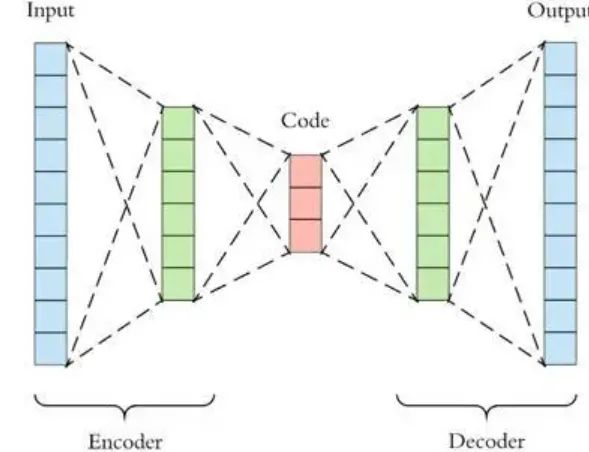
PCA是线性降维,AutoEncoder是非线性降维。根据正常数据训练出来的AutoEncoder,能够将正常样本重建还原,但是却无法将异于正常分布的数据点较好地还原,导致还原误差较大。因此如果一个新样本被编码,解码之后,它的误差超出正常数据编码和解码后的误差范围,则视作为异常数据。需要注意的是,AutoEncoder训练使用的数据是正常数据(即无异常值),这样才能得到重构后误差分布范围是多少以内是合理正常的。所以AutoEncoder在这里做异常检测时,算是一种有监督学习的方法。
import tensorflow as tf
from keras.models import Sequential
from keras.layers import Dense
# 标准化数据
scaler = preprocessing.MinMaxScaler()
X_train = pd.DataFrame(scaler.fit_transform(dataset_train),
columns=dataset_train.columns,
index=dataset_train.index)
# Random shuffle training data
X_train.sample(frac=1)
X_test = pd.DataFrame(scaler.transform(dataset_test),
columns=dataset_test.columns,
index=dataset_test.index)
tf.random.set_seed(10)
act_func = 'relu'
# Input layer:
model=Sequential()
# First hidden layer, connected to input vector X.
model.add(Dense(10,activation=act_func,
kernel_initializer='glorot_uniform',
kernel_regularizer=regularizers.l2(0.0),
input_shape=(X_train.shape[1],)
)
)
model.add(Dense(2,activation=act_func,
kernel_initializer='glorot_uniform'))
model.add(Dense(10,activation=act_func,
kernel_initializer='glorot_uniform'))
model.add(Dense(X_train.shape[1],
kernel_initializer='glorot_uniform'))
model.compile(loss='mse',optimizer='adam')
print(model.summary())
# Train model for 100 epochs, batch size of 10:
NUM_EPOCHS=100
BATCH_SIZE=10
history=model.fit(np.array(X_train),np.array(X_train),
batch_size=BATCH_SIZE,
epochs=NUM_EPOCHS,
validation_split=0.05,
verbose = 1)
plt.plot(history.history['loss'],
'b',
label='Training loss')
plt.plot(history.history['val_loss'],
'r',
label='Validation loss')
plt.legend(loc='upper right')
plt.xlabel('Epochs')
plt.ylabel('Loss, [mse]')
plt.ylim([0,.1])
plt.show()
# 查看训练集还原的误差分布如何,以便制定正常的误差分布范围
X_pred = model.predict(np.array(X_train))
X_pred = pd.DataFrame(X_pred,
columns=X_train.columns)
X_pred.index = X_train.index
scored = pd.DataFrame(index=X_train.index)
scored['Loss_mae'] = np.mean(np.abs(X_pred-X_train), axis = 1)
plt.figure()
sns.distplot(scored['Loss_mae'],
bins = 10,
kde= True,
color = 'blue')
plt.xlim([0.0,.5])
# 误差阈值比对,找出异常值
X_pred = model.predict(np.array(X_test))
X_pred = pd.DataFrame(X_pred,
columns=X_test.columns)
X_pred.index = X_test.index
threshod = 0.3
scored = pd.DataFrame(index=X_test.index)
scored['Loss_mae'] = np.mean(np.abs(X_pred-X_test), axis = 1)
scored['Threshold'] = threshod
scored['Anomaly'] = scored['Loss_mae'] > scored['Threshold']
scored.head()10. 基于分类的方法
One-Class SVM,这个算法的思路非常简单,就是寻找一个超平面将样本中的正例圈出来,预测就是用这个超平面做决策,在圈内的样本就认为是正样本,在圈外的样本是负样本,用在异常检测中,负样本可看作异常样本。它属于无监督学习,所以不需要标签。
One-Class SVM又一种推导方式是SVDD(Support Vector Domain Description,支持向量域描述),对于SVDD来说,我们期望所有不是异常的样本都是正类别,同时它采用一个超球体,而不是一个超平面来做划分,该算法在特征空间中获得数据周围的球形边界,期望最小化这个超球体的体积,从而最小化异常点数据的影响。
C是调节松弛变量的影响大小,说的通俗一点就是,给那些需要松弛的数据点多少松弛空间,如果C比较小,会给离群点较大的弹性,使得它们可以不被包含进超球体。
from sklearn import svm
# fit the model
clf = svm.OneClassSVM(nu=0.1, kernel='rbf', gamma=0.1)
clf.fit(X)
y_pred = clf.predict(X)
n_error_outlier = y_pred[y_pred == -1].size11. 基于预测的方法
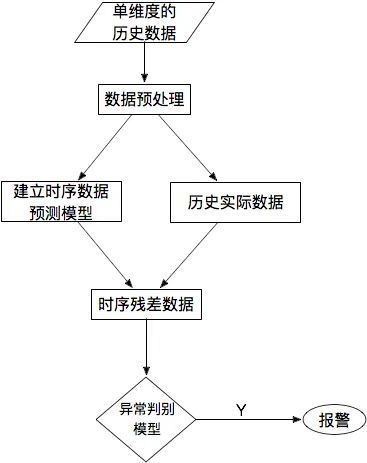
对于单条时序数据,根据其预测出来的时序曲线和真实的数据相比,求出每个点的残差,并对残差序列建模,利用KSigma或者分位数等方法便可以进行异常检测。具体的流程如下:
12. 总结
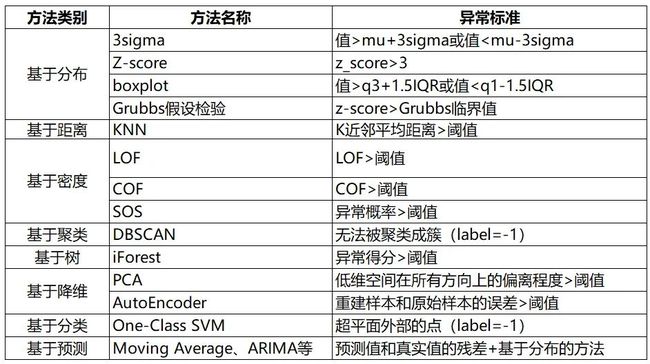
异常检测方法总结如下:
参考文献
【算法】异常检测方法总结
异常检测算法分类及经典模型概览 - 知乎