机器学习——聚类——商场客户聚类
聚类的介绍
…………………………………………
…………………………………………
案例——商场客户聚类
目录
聚类的介绍
案例——商场客户聚类
一、读取数据
二、聚类
KMeans函数的参数讲解:
KMeans属性列表
KMeans接口列表
三、查看数据及可视化
sort_values()方法
groupby()的常见用法
groupby()的配合函数
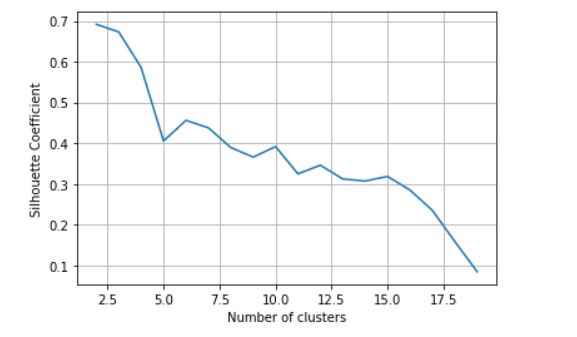
四、聚类评价指标。计算聚簇数量从2到19时的轮廓系数。
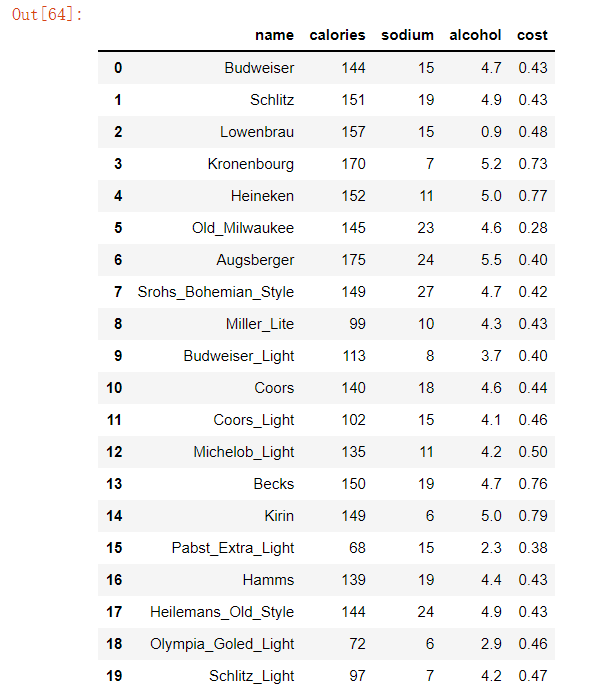
一、读取数据
import pandas as pd
beer=pd.read_csv('beer.txt',sep=' ')
#输出文件内容
beerpandas.read_csv
常用参数为:header, sep, name……
header(表头):
指定行数用来作为列名,数据开始行数。
如果文件中没有列名,则默认为0,否则设置为None。如果明确设定header=0 就会替换掉原来存在列名。
header参数可以是一个list例如:[0,1,3],这个list表示将文件中的这些行作为列标题(意味着
每一列有多个标题),介于中间的行将被忽略掉(例如本例中的2;本例中的数据1,2,4行将被作为多级标题出现,第3行数据将被丢弃,dataframe的数据从第5行开始)。
注意:如果skip_blank_lines=True 那么header参数忽略注释行和空行,所以header=0表示第一
行数据而不是文件的第一行。
sep(分隔符):
指定分隔符。如果不指定参数,则会尝试使用逗号分隔。分隔符长于一个字符并且不是‘\s+’,
将使用python的语法分析器。并且忽略数据中的逗号。正则表达式例子:’\r\t’
names(列名):
用于结果的列名列表,如果数据文件中没有列标题行,就需要执行header=None。默认列表中不
能出现重复,除非设定参数mangle_dupe_cols=True。
二、聚类。把除name之外的所有属性当作输入特征
name 为离散型,所有使用drop删除此行,剩下的为连续性数据
n_clusters:初始化一个3个的聚簇
random_state:及初始化生成的3个聚簇进行标记
确定质心初始化的随机数生成。使用int可以使随机性具有确定性。 确定质心初始化的随机数生成.使用int可以使随机性具有确定性.
fit训练过程:
1.(随机)选择K个聚类的初始中心;
2.对任意一个样本点,求其到K个聚类中心的距离,将样本点归类到距离最小的中心的聚类,如此迭代n次;
3.每次迭代过程中,利用均值等方法更新各个聚类的中心点(质心);
4.对K个聚类中心,利用2,3步迭代更新后,如果位置点变化很小(可以设置阈值),则认为达到稳定状态,迭代结束,对不同的聚类块和聚类中心可选择不同的颜色标注。
#准备输入特征X
X=beer.drop('name',axis=1)
from sklearn.cluster import KMeans
km=KMeans(n_clusters=3,random_state=1)
km.fit(X)drop函数的使用:删除行、删除列:
| 参数名称 | 参数取值 | 参数意义 |
| axis | 0 or ‘index’, 1 or ‘columns’ , default 0 | 确定是删除包含缺失值的行还是列 |
| how | ‘any’ or ‘all’, default ‘any’ | 表明是至少存在一个NAN值还是全为NAN值时执行删除操作 |
| thresh | int, 可选 | 指定存在多少个NAN值才进行删除操作 |
| subset | array, 可选 | 可选子集列表 |
| inplace | bool, default False | 如果为真,执行inplace操作,并返回None |
KMeans函数的参数讲解:
1、n_clusters : 聚类中心数量(开始时需要产生的聚类中心数量),默认为8
2、max_iter : 算法运行的最大迭代次数,默认300
3、tol: 容忍的最小误差,当误差小于tol就会退出迭代(算法中会依赖数据本身),默认为1e-4
4、n_init : k-means算法会随机运行n_init次,最终的结果将是最好的一个聚类结果,默认10
5、init : 聚类中心的初始化方案,有三个选择{'k-means++', 'random' or an ndarray}
5.1、 'k-means++' : 默认选项,初始化过程如下
(1)、从输入的数据点集合(要求有k个聚类)中随机选择一个点作为第一个聚类中心;
(2)、对于数据集中的每一个点x,计算它与最近聚类中心(指已选择的聚类中心)的距离D(x);
(3)、选择一个新的数据点作为新的聚类中心,选择的原则是:D(x)较大的点,被选取作为聚类中心的概率较大;(4)、重复2和3直到k个聚类中心被选出来
5.2、'random': 随机选择k个实例作为聚类中心
5.3、ndarray:如果传入为矩阵(ndarray),则将该矩阵中的每一行作为聚类中心
6、algorithm :可选的K-means距离计算算法, 可选{"auto", "full" or "elkan",default="auto"}
6.1"full":传统的距离计算方式.
6.2"elkan":使用三角不等式,效率更高,但是目前不支持稀疏数据。1、计算任意两个聚类中心的距离;2当计算x点应该属于哪个聚类中心时,当发现2*S(x,K1)
6.3"auto":当为稀疏矩阵时,采用full,否则elkan。
7、precompute_distances : 是否将数据全部放入内存计算,可选{'auto', True, False},开启时速度更快但是更耗内存.
7.1、'auto' : 当n_samples * n_clusters > 12million,不放入内存,否则放入内存,double精度下大概要多用100M的内存
7.2、True : 进行预计算
7.3、False : 不进行预计算
8、n_jobs : 同时进行计算的核数(并发数),n_jobs用于并行计算每个n_init,如果设置为-1,使用所有CPU,若果设置为1,不并行,如果设置小于-1,使用CPU个数+1+n_jobs个CPU
9、random_state : 用于随机产生中心的随机序列
10、verbose : 是否输出详细信息,默认为0,bush
11、copy_x : 是否直接在原矩阵上进行计算。默认为True,会copy一份进行计算。
KMeans属性列表
KMeans接口列表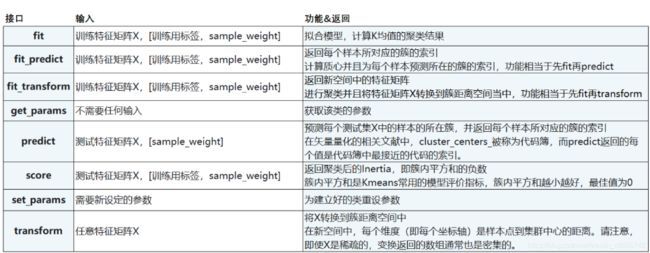
三、查看数据及可视化
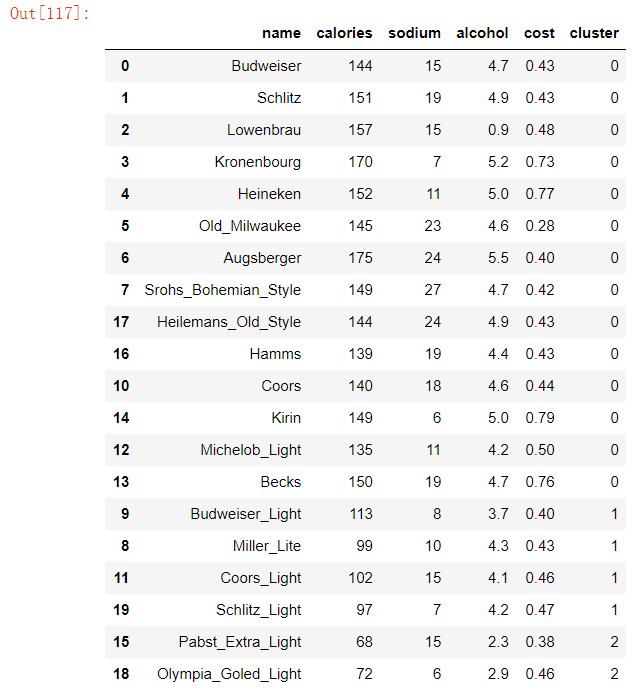
在表beer中添加一列cluster放入标签数据,km.labels_每个样本点对应的标签,并进行升序排序
beer['cluster']=km.labels_
beer.sort_values(by='cluster')sort_values()方法
sort_values(by, axis=0, ascending=True, inplace=False, kind=‘quicksort’, na_position=‘last’)
| 参数 | 含义 |
| axis | 如果axis=0,那么by=“列名”; 如果axis=1,那么by=“行名”; |
| ascending | True则升序,可以是[True,False],即第一字段升序,第二个降序 |
| inplace | 是否用排序后的数据框替换现有的数据框 ,True,或者False |
| kind | 排序方法 |
| na_position | {‘first’, ‘last’}, default ‘last’,默认缺失值排在最后面 |
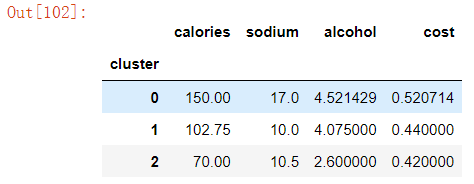
查看每个聚类的每维特征上的平均值(即聚簇中心点)
centers=beer.groupby('cluster').mean()
centersgroupby()的常见用法
| 函数 | 适用场景 | 备注 |
| df.groupby(‘key1’) | 一列聚合 | 分组键为列名(可以是字符串、数字或其他Python对象) |
| df.groupby([‘key1’,‘key2’]) | 多列聚合 | 分组键为列名,引入列表list[] |
| df[‘data1’].groupby(df[‘key1’]).mean() | 按某一列进行一重聚合求均值 | 分组键为Series |
| A=df[‘订单编号’].groupby([ df[‘运营商’], df[‘分类’], df[‘百度圣卡’] ]).count() | 按某一列进行多重聚合计数 | 分组键为Series,引入列表list[] |
| df[‘data1’].groupby([states,years]).mean() | 分组键与原df无关,而是另外指定的任何长度适当的数组 | 分组键是数组,state和year均为数组 |
groupby()的配合函数
| 函数 | 适用场景 | 备注 |
|---|---|---|
| .mean() | 均值 | |
| .count() | 计数 | |
| .min() | 最小值 | |
| .mean().unstack() | 求均值,聚合表的层次索引不堆叠 | |
| .size() | 计算分组大小 | GroupBy的size方法,将返回一个含有分组大小的Series |
| .apply() | ||
| .agg() |
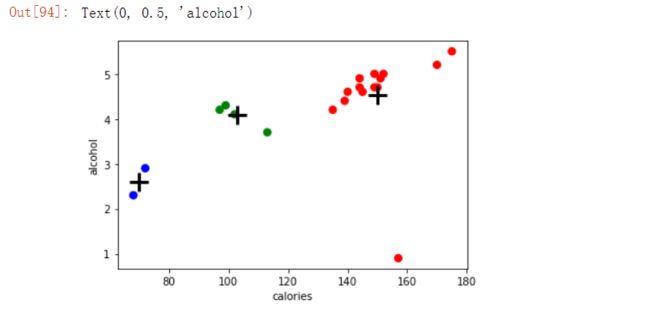
#可视化
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
colors =np.array(['red','green','blue','yellow'])
plt.scatter(beer.calories,beer.alcohol,c=colors[beer.cluster],s=50)
plt.scatter(centers.calories,centers.alcohol,linewidths=3,marker='+',s=300,c='black')
plt.xlabel('calories')
plt.ylabel('alcohol')四、聚类评价指标。计算聚簇数量从2到19时的轮廓系数。
from sklearn import metrics
k_range=range(2,20)
scores=[]
for k in k_range:
km=KMeans(n_clusters=k,random_state=1)
km.fit(X)
scores.append(metrics.silhouette_score(X,km.labels_))
plt.plot(k_range,scores)
plt.xlabel('Number of clusters')
plt.ylabel('Silhouette Coefficient')
plt.grid(True)聚类性能评估-轮廓系数 - 知乎
轮廓系数(Silhouette Coefficient)
是聚类效果好坏的一种评价方式。轮廓系数取值范围为[-1,1],取值越接近1则说明聚类性能越好,相反,取值越接近-1则说明聚类性能越差。
a:某个样本与其所在簇内其他样本的平均距离
b:某个样本与其他簇样本的平均距离
针对某个样本的轮廓系数s为:
![]()
聚类总的轮廓系数SC为:
![]()
轮廓系数的优点:
轮廓系数为-1时表示聚类结果不好,为+1时表示簇内实例之间紧凑,为0时表示有簇重叠。
轮廓系数越大,表示簇内实例之间紧凑,簇间距离大,这正是聚类的标准概念。
轮廓系数的缺点:
对于簇结构为凸的数据轮廓系数值高,而对于簇结构非凸需要使用DBSCAN进行聚类的数据,轮廓系数值低,因此,轮廓系数不应该用来评估不同聚类算法之间的优劣,比如Kmeans聚类结果与DBSCAN聚类结果之间的比较。
根据折线图可直观的找到系数变化幅度最大的点,认为发生畸变幅度最大的点就是最好的聚类数目。
完整代码:
import pandas as pd
beer=pd.read_csv('beer.txt',sep=' ')
beer
#准备输入特征X
X=beer.drop('name',axis=1)
from sklearn.cluster import KMeans
km=KMeans(n_clusters=3,random_state=1)
km.fit_predict(X)
beer['cluster']=km.labels_
beer.sort_values(by='cluster')
centers=beer.groupby('cluster').mean()
centers
#可视化
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
colors =np.array(['red','green','blue','yellow'])
plt.scatter(beer.calories,beer.alcohol,c=colors[beer.cluster],s=50)
plt.scatter(centers.calories,centers.alcohol,linewidths=3,marker='+',s=300,c='black')
plt.xlabel('calories')
plt.ylabel('alcohol')
from sklearn import metrics
k_range=range(2,20)
scores=[]
for k in k_range:
km=KMeans(n_clusters=k,random_state=1)
km.fit(X)
scores.append(metrics.silhouette_score(X,km.labels_))
plt.plot(k_range,scores)
plt.xlabel('Number of clusters')
plt.ylabel('Silhouette Coefficient')
plt.grid(True)今天看到了一句话:
当你已经差到谷底的时候,已经不能再差了,那无论你做什么,都是在走上坡路。
努力奋斗,你行滴!
点个小♥