深度学习图像处理目标检测图像分割计算机视觉 06--图像分类
深度学习图像处理目标检测图像分割计算机视觉 06--图像分类
- 摘要
- 一、图像分类
-
- 1.1 AlexNet(8层)
- 1.2 VGG(19层)
- 1.3 GoogLeNet
- 1.4 ResNet、ResNext
- 二、U-Net模型
-
- 2.1 U-Net
- 2.2 实现
- 三、甲状腺癌研究现状
-
- 3.1 影像学评估在TC诊断中应用
- 3.2 人工智能助力TC的诊断与决策
- 总结
摘要
本周的主要工作是学习深度学习图像分类,跑出一个UNET模型,了解甲状腺癌的研究现状和研究的技术难点。
一、图像分类
图片分类就是给图片一个标签

但是这个标签并不是随便给的,有一个数据集

卷积神经网络进化的过程有几个代表性的网络:

1.1 AlexNet(8层)
AlexNet标志着DNN深度学习革命的开始。 VGG网络是网络改造的首选基础网络。 在GoogLeNet提出之前,模型完全靠猜或者试,GoogLeNet提出一个想法,能否把所有的情况都列举出来,比如堆叠多少卷积核,卷积核是大还是小,要不要加入最大池化层或者卷积直接代替池化,让模型自己训练,决定哪个好,采用什么样的顺序才能对性能提升,把所有的性能都训练出来,让事实说话。 U-Net诞生的一个主要前提是,很多时候深度学习的结构需要大量的sample和计算资源,但是U-Net基于FCN(Fully Convultional Neural Network:全卷积神经网络)进行改进,并且利用数据增强(data augmentation)可以对一些比较少样本的数据进行训练,特别是医学方面相关的数据(医学数据比一般我们所看到的图片及其他文本数据的获取成本更大,不论是时间还是资源的消耗),所以U-Net的出现对于深度学习用于较少样本的医学影像是很有帮助的。生物医学与人工智能的结合正在如火如荼地发展着,利用机器学习手段处理医学影像,可以大大提高医学诊断的效率和精确度。医学影像处理有时涉及到细胞分割,即将图像中的细胞识别出来并把细胞与背景分割开来。为了帮助对这一结果具有更直观的了解,可以参考如下原图和分割图像的效果图: Unet在ISBI 2015 显微图像分割竞赛中拔得头筹,分割效果远优于其他网络,现在已经成为医疗影像处理中最主流的分割网络。与传统的全卷积神经网络相比,Unet进行了改进,通过层与层之间更强的联系,加上上采样和下卷积,实现特征的充分提取,所以可以使用更少的训练样本得到准确的分割。 从图中可以看出,除输出层外,Unet每一层以三层卷积构成,层间采用池化或上采样的方法实现特征的提取和整合,最后一层将之前所有提取出来的特征做一个二分类,实现细胞的语义分割。 UNet 甲状腺癌(thyroidcancer,TC)是世界上常见的恶性肿瘤之一,根据临床和病理学分型可分为分化型甲状腺癌,甲状腺髓样痒和甲状腺未分化癌。虽然TC患者病死率仅为040/10万,但其发病率在全球恶性肿瘤中位列第9值得关注。通过对国内外TC临床研究的高频主题词进行聚类分析,学者们对TC的临床研究聚焦于结合AI诊断评估方向。深度学习在医疗图像诊断领域、甲状腺结节超声诊断方向显示出较大的优势和应用前景。利用深度学习算法分析超声图像构建超声自动诊断系统,可辅助甲状腺肿瘤超声诊断,简化超声医生的工作流程,有助于提高临床实践效率。 甲状腺癌影像诊断首选超声,当超声检查发现高危结节时,需进行活组织检查/分子检测,进而决定是否手术治疗,而中低危患者需行超声随访。甲状腺的评估可以使用几种影像技术进行。包括普通放射线照相、放射性核素成像、超声检查、电子计算机断层扫描和核磁共振成像等检查。每种技术有其优点和局限性,并且通常认为在多数患者中进行此类研究没有绝对的临床指征。技术的主要限制,除了费用和可操作性,主要是其缺乏特异性组织学诊断。超声是一种无创、安全、简便、经济的检查方法,在临床应用广泛,甲状腺是人体浅表器官,超声检查是甲状腺结节诊断的首选检查手段。另外超声弹性成像(UE)、超声造影(CEUS)及超声引导下细针穿刺细胞学检查(US-FNA)等超声新技术的应用进一步提高了TC的诊断准确率。2020年韩国甲状腺放射学会发布了US在甲状腺疾病中的临床应用指南,为US在甲状腺疾病中的临床应用提供指导。国外学者在借鉴孚腺BI-RADS分级的基础上首先提出甲状腺影像学报告及数据系统(TI-RADS),以减少人为的诊断差异,提高诊断一致性。TI-RADS分类有多个学会指南版本,它们的差异主要体现在诊断性能及活检阈值上。根据2019年的一项meta分析中得出的结论,在五种常见的来自欧美及韩国学的TI-RADS分类中,美国放射协会发布的ACR-TIRADS在选择需要FNA的结节时具有更好的表现。在国内大多数机构中最常使用的超声评估方法为C-TIRADS,因其在应用过程中省略了数学计算在使用上较ACR-TIRADS更为便捷。另外在使用ACR-TIRADS分类方法时,对具有可疑恶性超声特征的甲状腺结节,一般需行US-FNA后再制定进一步的诊疗方案,而C-TIRADS的结节分类结果可为尚未开展US-FNA的医疗机构决定是否手术治疗提供参考,更符合中国国情。目前,在US及TI-RADS分类评估的基础上,联合UE,CUES,US-FNA多种超声方法诊断TC的研究同样值得关注,以期提高TC的术前的确诊率,最大限度地降低由误诊带来的创伤性检查和治疗。 随着临床大数据的积累与计算机算法的成熟使AI与临床的深度融合成为可能。近年来,AI已被应用于医学影像分析,协助医生在就诊时提供最好的诊断和治疗建议。早在2013年国内即开展了AI用于甲状腺结节超声图像的辅助诊断相关研究,截至2019年底,相关文献已有19篇,其中12篇文章来自国内学者。2019年柳叶刀子刊的一项多中心回顾性研究表明,与一组熟练的放射科医生相比,可以模拟人脑进行分析学习的深度神经网络在超声图像中识别TC患者显示出类似的敏感性和更高的特异性。郭天南等使用AI技术对近千例甲状腺结节患者的蛋白质组数据进行分析,发现了14个能够鉴别TC的关键性蛋白质组合,这些组合构成了判断甲状腺结节良恶性的模型。另外,该团队用来自国内多个中心提供的288个甲状腺石蜡切片和64个US-FNA样本对该模型进行验证,其准确率高达90%。此类研究为提高临床医生诊断TC的效率并减少不必要的穿刺,或对US-FNA结果不确定的结节提供了一种辅助诊断新方式。不过,在一项最新的研究中显示,AI辅助诊断存在潜在性的伦理风险,当超声医生与AI诊断建议不一致时,有半数医生对诊新进行了修改,但是其中25%的诊断经病理证实为错误修改。因此对于AI技术在临床实践中的角色与定位需要在未来行进一步探索。 人工神经网络(artificial neural network,ANN)指具有非线性适应性信息处理能力的计算方法(学习),是一种基于大数据的运算模型,有别于普通的统计学方法。统计学是以对点数据的计算推论面的 从超声图像中分割甲状腺结节边界在临床指标计算和甲状腺疾病诊断中起着重要作用。但由于甲状腺 总之,近年来国内外对TC临床研究在上述方面取得了显者进展。对于国内TC临床研究未来的发展需聚焦国际热点,如进行TC手术的标准制定以及开展超声诊断研究等,加强与国外TC专家的互访与合作,更多地参与到国际多中心临床试验中,利用我国TC患者基数庞大的优势并结合AI系统,着手建立基于医学大数据的TC专病互联网数据库平台,力争并完全有条件在国际TC研究领域位居前列。 本周完成了深度学习图像分类的算法学习,并且了解了甲状腺目前的研究现状。甲状腺结节的检查目前多用超声成像检查,因为超声是一种无创、安全、简便、经济的检查方法,在临床应用广泛,甲状腺是人体浅表器官,超声检查是甲状腺结节诊断的首选检查手段。而且随着超声的发展,进一步提高了甲状腺结节的诊断准确率。人工智能的发展,将AI技术应用到准确医疗上,而且我国患者基数庞大,这个优势结合AI系统可以实现互联网加准确医疗,已有学者利用庞大的基数进行甲状腺结节的神经网络训练,并在测试集上进行判断,在与医生诊断不一致的病例中,多数是医生修改了诊断结果。大数据将使其临床特性更清晰,实现精准诊疗。

AlexNet是分为两个通道进行训练的,但是,这两个通道并不是完全不相交的,卷积层之间会交互信息,全连接层之间会互相交叉信息。

在每一层上并不是只有卷积一种操作,有涉及到卷积,池化,激活函数等。

在AlexNet中,有一个模型后来被证实并没有什么用,就是LRN局部响应归一化。

NiN在AlexNet的基础上提出来的。

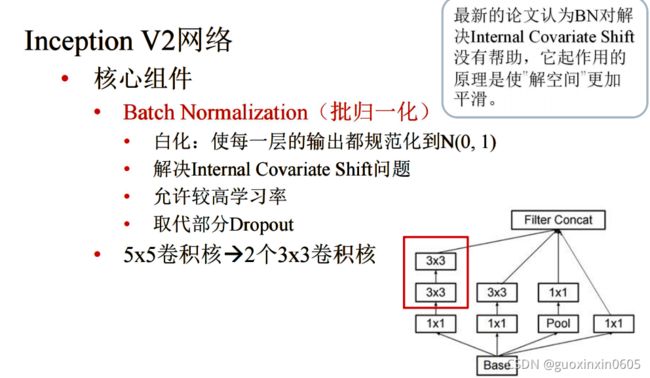
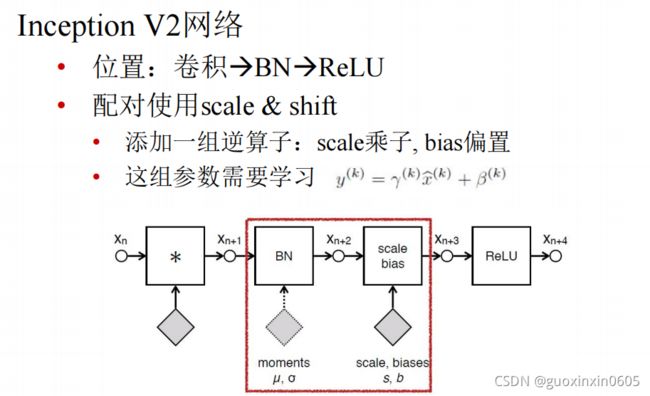
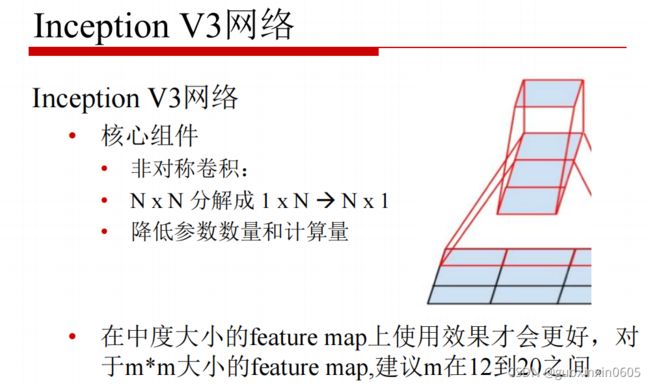
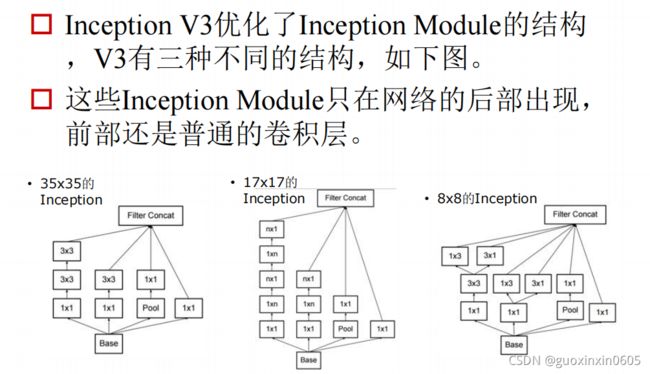
1x1卷积的作用是实现跨通道的交互和信息整合。进行卷积核通道数的降维和升维。c是卷积前的通道数,k是卷积后的通道数。k1.2 VGG(19层)

它把一个大的卷积核分解成为连续的多个小的卷积核,这样做的优势是参数量小,在
卷积层数增加时,参数量的增加并不会太多。但是这样做的劣势使操作时的速度比较慢。1.3 GoogLeNet

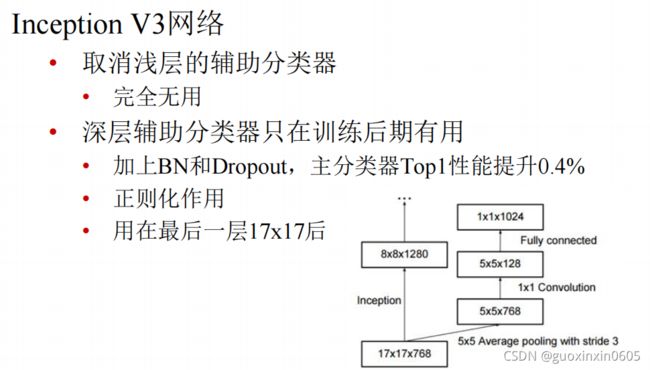
在GoogLeNet有一个重要的模块:Inception,进化了4个阶段。1.4 ResNet、ResNext
二、U-Net模型
2.1 U-Net
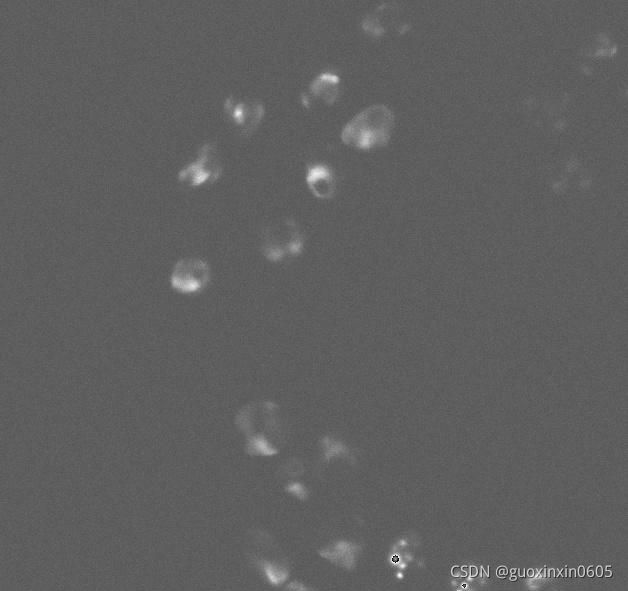

从这两张图可以看出,原图中细胞所在的地方在分割图像中被分割标注了出来,这也就是细胞分割。分割图像中每个细胞的分割被用不同的颜色标注,看起来像是实例分割(多分类),但事实上,细胞与细胞之间分成不同的类别还是太困难了,毕竟神经网络在学习的过程中基本上不能认出来不同颜色标注出来的细胞有什么本质上的区别。所以这里的不同的颜色其实是后续的处理结果,最开始的分割其实是语义分割(二分类)的结果,如下图所示: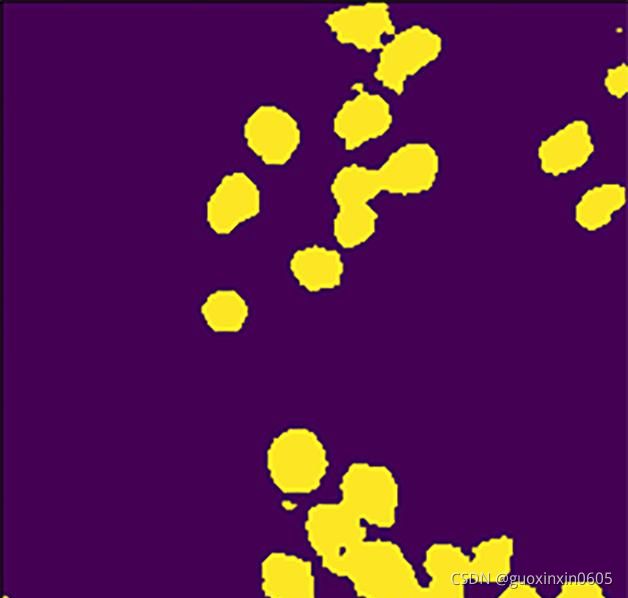
所以要如何实现这样的语义分割呢?这时在细胞分割领域独领风骚的Unet就闪亮登场啦。Unet是一种比较年轻的神经网络,顾名思义,其结构为U形,在卷积层一层一层提取特征的同时将首尾对称的两层联系起来,结构图如下:2.2 实现
# This Python 3 environment comes with many helpful analytics libraries installed
# It is defined by the kaggle/python docker image: https://github.com/kaggle/docker-python
# For example, here's several helpful packages to load in
import numpy as np # linear algebra
import pandas as pd # data processing, CSV file I/O (e.g. pd.read_csv)
import os
import numpy as np
import pandas as pd
import scipy.io
from skimage.transform import resize
import matplotlib.pyplot as plt
from tqdm import tqdm
import gc
gc.collect()
# Input data files are available in the "../input/" directory.
# For example, running this (by clicking run or pressing Shift+Enter) will list the files in the input directory
import os
print(os.listdir("../input"))
# https://www.kaggle.com/pierrenicolaspiquin/oct-segmentation/data
# Settings
input_path = os.path.join('..', 'input', '2015_boe_chiu', '2015_BOE_Chiu')
subject_path = [os.path.join(input_path, 'Subject_0{}.mat'.format(i)) for i in range(1, 10)] + [os.path.join(input_path, 'Subject_10.mat')]
data_indexes = [10, 15, 20, 25, 28, 30, 32, 35, 40, 45, 50]
width = 284
height = 284
width_out = 196
height_out = 196
mat = scipy.io.loadmat(subject_path[0])
img_tensor = mat['images']
manual_fluid_tensor_1 = mat['manualFluid1']
img_array = np.transpose(img_tensor, (2, 0, 1))
manual_fluid_array = np.transpose(manual_fluid_tensor_1, (2, 0, 1))
plt.imshow(img_array[25])
plt.imshow(manual_fluid_array[25])
def thresh(x):
if x == 0:
return 0
else:
return 1
thresh = np.vectorize(thresh, otypes=[np.float])
def create_dataset(paths):
x = []
y = []
for path in tqdm(paths):
mat = scipy.io.loadmat(path)
img_tensor = mat['images']
fluid_tensor = mat['manualFluid1']
img_array = np.transpose(img_tensor, (2, 0 ,1)) / 255
img_array = resize(img_array, (img_array.shape[0], width, height))
fluid_array = np.transpose(fluid_tensor, (2, 0 ,1))
fluid_array = thresh(fluid_array)
fluid_array = resize(fluid_array, (fluid_array .shape[0], width_out, height_out))
for idx in data_indexes:
x += [np.expand_dims(img_array[idx], 0)]
y += [np.expand_dims(fluid_array[idx], 0)]
return np.array(x), np.array(y)
x_train, y_train = create_dataset(subject_path[:9])
x_val, y_val = create_dataset(subject_path[9:])
x_train.shape, y_train.shape, x_val.shape, y_val.shape
import torch
import torchvision
import torch.nn as nn
import torch.nn.functional as F
import torch.optim as optim
from torch.autograd import Variable
from tqdm import trange
from time import sleep
use_gpu = torch.cuda.is_available()
batch_size = 9
epochs = 1000
epoch_lapse = 50
threshold = 0.5
sample_size = None
class UNet(nn.Module):
def contracting_block(self, in_channels, out_channels, kernel_size=3):
block = torch.nn.Sequential(
torch.nn.Conv2d(kernel_size=kernel_size, in_channels=in_channels, out_channels=out_channels),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(out_channels),
torch.nn.Conv2d(kernel_size=kernel_size, in_channels=out_channels, out_channels=out_channels),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(out_channels),
)
return block
def expansive_block(self, in_channels, mid_channel, out_channels, kernel_size=3):
block = torch.nn.Sequential(
torch.nn.Conv2d(kernel_size=kernel_size, in_channels=in_channels, out_channels=mid_channel),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(mid_channel),
torch.nn.Conv2d(kernel_size=kernel_size, in_channels=mid_channel, out_channels=mid_channel),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(mid_channel),
torch.nn.ConvTranspose2d(in_channels=mid_channel, out_channels=out_channels, kernel_size=3, stride=2, padding=1, output_padding=1)
)
return block
def final_block(self, in_channels, mid_channel, out_channels, kernel_size=3):
block = torch.nn.Sequential(
torch.nn.Conv2d(kernel_size=kernel_size, in_channels=in_channels, out_channels=mid_channel),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(mid_channel),
torch.nn.Conv2d(kernel_size=kernel_size, in_channels=mid_channel, out_channels=mid_channel),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(mid_channel),
torch.nn.Conv2d(kernel_size=kernel_size, in_channels=mid_channel, out_channels=out_channels, padding=1),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(out_channels),
)
return block
def __init__(self, in_channel, out_channel):
super(UNet, self).__init__()
#Encode
self.conv_encode1 = self.contracting_block(in_channels=in_channel, out_channels=64)
self.conv_maxpool1 = torch.nn.MaxPool2d(kernel_size=2)
self.conv_encode2 = self.contracting_block(64, 128)
self.conv_maxpool2 = torch.nn.MaxPool2d(kernel_size=2)
self.conv_encode3 = self.contracting_block(128, 256)
self.conv_maxpool3 = torch.nn.MaxPool2d(kernel_size=2)
# Bottleneck
self.bottleneck = torch.nn.Sequential(
torch.nn.Conv2d(kernel_size=3, in_channels=256, out_channels=512),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(512),
torch.nn.Conv2d(kernel_size=3, in_channels=512, out_channels=512),
torch.nn.ReLU(),
torch.nn.BatchNorm2d(512),
torch.nn.ConvTranspose2d(in_channels=512, out_channels=256, kernel_size=3, stride=2, padding=1, output_padding=1)
)
# Decode
self.conv_decode3 = self.expansive_block(512, 256, 128)
self.conv_decode2 = self.expansive_block(256, 128, 64)
self.final_layer = self.final_block(128, 64, out_channel)
def crop_and_concat(self, upsampled, bypass, crop=False):
if crop:
c = (bypass.size()[2] - upsampled.size()[2]) // 2
bypass = F.pad(bypass, (-c, -c, -c, -c))
return torch.cat((upsampled, bypass), 1)
def forward(self, x):
# Encode
encode_block1 = self.conv_encode1(x)
encode_pool1 = self.conv_maxpool1(encode_block1)
encode_block2 = self.conv_encode2(encode_pool1)
encode_pool2 = self.conv_maxpool2(encode_block2)
encode_block3 = self.conv_encode3(encode_pool2)
encode_pool3 = self.conv_maxpool3(encode_block3)
# Bottleneck
bottleneck1 = self.bottleneck(encode_pool3)
# Decode
##print(x.shape, encode_block1.shape, encode_block2.shape, encode_block3.shape, bottleneck1.shape)
##print('Decode Block 3')
##print(bottleneck1.shape, encode_block3.shape)
decode_block3 = self.crop_and_concat(bottleneck1, encode_block3, crop=True)
##print(decode_block3.shape)
##print('Decode Block 2')
cat_layer2 = self.conv_decode3(decode_block3)
##print(cat_layer2.shape, encode_block2.shape)
decode_block2 = self.crop_and_concat(cat_layer2, encode_block2, crop=True)
cat_layer1 = self.conv_decode2(decode_block2)
##print(cat_layer1.shape, encode_block1.shape)
##print('Final Layer')
##print(cat_layer1.shape, encode_block1.shape)
decode_block1 = self.crop_and_concat(cat_layer1, encode_block1, crop=True)
##print(decode_block1.shape)
final_layer = self.final_layer(decode_block1)
##print(final_layer.shape)
return final_layer
def train_step(inputs, labels, optimizer, criterion):
optimizer.zero_grad()
# forward + backward + optimize
outputs = unet(inputs)
# outputs.shape =(batch_size, n_classes, img_cols, img_rows)
outputs = outputs.permute(0, 2, 3, 1)
# outputs.shape =(batch_size, img_cols, img_rows, n_classes)
outputs = outputs.resize(batch_size*width_out*height_out, 2)
labels = labels.resize(batch_size*width_out*height_out)
loss = criterion(outputs, labels)
loss.backward()
optimizer.step()
return loss
learning_rate = 0.01
unet = UNet(in_channel=1,out_channel=2)
if use_gpu:
unet = unet.cuda()
criterion = nn.CrossEntropyLoss()
optimizer = optim.SGD(unet.parameters(), lr = 0.01, momentum=0.99)
def get_val_loss(x_val, y_val):
x_val = torch.from_numpy(x_val).float()
y_val = torch.from_numpy(y_val).long()
if use_gpu:
x_val = x_val.cuda()
y_val = y_val.cuda()
m = x_val.shape[0]
outputs = unet(x_val)
# outputs.shape =(batch_size, n_classes, img_cols, img_rows)
outputs = outputs.permute(0, 2, 3, 1)
# outputs.shape =(batch_size, img_cols, img_rows, n_classes)
outputs = outputs.resize(m*width_out*height_out, 2)
labels = y_val.resize(m*width_out*height_out)
loss = F.cross_entropy(outputs, labels)
return loss.data
epoch_iter = np.ceil(x_train.shape[0] / batch_size).astype(int)
t = trange(epochs, leave=True)
for _ in t:
total_loss = 0
for i in range(epoch_iter):
batch_train_x = torch.from_numpy(x_train[i * batch_size : (i + 1) * batch_size]).float()
batch_train_y = torch.from_numpy(y_train[i * batch_size : (i + 1) * batch_size]).long()
if use_gpu:
batch_train_x = batch_train_x.cuda()
batch_train_y = batch_train_y.cuda()
batch_loss = train_step(batch_train_x , batch_train_y, optimizer, criterion)
total_loss += batch_loss
if (_+1) % epoch_lapse == 0:
val_loss = get_val_loss(x_val, y_val)
print(f"Total loss in epoch {_+1} : {total_loss / epoch_iter} and validation loss : {val_loss}")
gc.collect()
def plot_examples(datax, datay, num_examples=3):
fig, ax = plt.subplots(nrows=3, ncols=4, figsize=(18,4*num_examples))
m = datax.shape[0]
for row_num in range(num_examples):
image_indx = np.random.randint(m)
image_arr = unet(torch.from_numpy(datax[image_indx:image_indx+1]).float().cuda()).squeeze(0).detach().cpu().numpy()
ax[row_num][0].imshow(np.transpose(datax[image_indx], (1,2,0))[:,:,0])
ax[row_num][0].set_title("Orignal Image")
ax[row_num][1].imshow(np.transpose(image_arr, (1,2,0))[:,:,0])
ax[row_num][1].set_title("Segmented Image")
ax[row_num][2].imshow(image_arr.argmax(0))
ax[row_num][2].set_title("Segmented Image localization")
ax[row_num][3].imshow(np.transpose(datay[image_indx], (1,2,0))[:,:,0])
ax[row_num][3].set_title("Target image")
plt.show()
plot_examples(x_train, y_train)
plot_examples(x_val, y_val)
torch.save(unet.state_dict(), 'unet.pt')
三、甲状腺癌研究现状
3.1 影像学评估在TC诊断中应用
3.2 人工智能助力TC的诊断与决策
结局,而大数据是以面数据的归集计算推论点的结果,“是互联、非线性、自适应的信息处理系统”。其
模仿人脑神经元网络建立的基于统计的机器学习方法,是通向AI的途径之一。DL隶属于ANN系统,允许由多个处理层组成的计算模型学习具有多个抽象级别的数据表示[20-21]。DL是一种特定类型的机器学习,具有强大的能力和灵活性,其将大千世界表示为嵌套的层次概念体系(由较简单概念间的联系定义复杂概念、从一般抽象概况到高级抽象表示)[22]。在深度学习算法领域目前有多种神经网络模型,如卷积神经网络(convolutional neural network,CNN)(擅长图像处理)、循环神经(recurrent neural network,RNN)(擅长语言文本处理)等。在图像处理中常使用的是CNN。CNN 是一类包含卷积计算的 ANN,当具有深度网络结构时称为深度卷积神经网络(deep convolu⁃tional neural network,DCNN),是近年来深度学习在计算机视觉中取得突破性成果的基石。DCNN 由许多神经元构成的网络层堆叠而来,因非线性激活函数层的网络架构,可以将其视为一个非线性函数,因此整个深度神经网络可以视为复合非线性多元函数。DCNN主要结构包括卷积部分和全连接部分:卷积部分包括卷积层、激活层和池化层,可从图像提取特征;全连接部分则连接特征提取和输出计算损失,完成识别分类。理上讲,层数越深,参数越多,则函数模拟能力越强,而在实际操作中,学者通过简单堆叠增加网络深度发现其误差同样增加,因此科学家不断采用各种方法优化神经网络,使其具备更强大的特征学习和分类能力。自从 AlexNet 在 2012 年夺得了 ImageNet 挑战赛冠军,DCNN成为计算机视觉领域的首选算法。经典网 络 包 括 AlexNet、ZFNet、VGGNet、GoogleNet、ResNet、Inception- v3、Inception- v4 及 DenseNet。ResNet[23]于2015年被提出,在ImageNet比赛中,其准确率首次超过人类专家。该网具有2个特点:1)使用了残差块;2)可以构建极深的网络结构(能达到152层或更高)。解决了DCNN经常出现的梯度消失的困扰。DenseNet[24]是2017年计算机视觉与模式识别会议最佳论文,通过特征复用和旁路设置,既大幅度减少了网络的参数量,又在一定程度上缓解了梯度消失和模型退化的问题无论是 ResNet 还是DenseNet,核心的思想均为跳跃链接,将某些输入不加选择的使其进入跃层,实现信息流的整合,避免信息在层间传递的丢失和梯度消失的问题,且后者参数量减少了50%。
结节的异质性和与背景相似的成分,通过机器自动准确地分割甲状腺结节具有挑战性,CNN的出现使得从超声图像中自动分割甲状腺结节更容易实现。总结