DNA测序技术发展史:一代、二代、三代测序技术简要原理及比较
一代、二代、三代测序技术简要原理及比较
- 1.一代测序
-
- 1.1 Sanger测序
- 2.二代测序
-
- 2.1.Illumina Solexa合成测序
- 2.2.Roche 454焦磷酸测序
- 2.3.ABI SOLiD连接法测序
- 2.4.BGI(华大基因):纳米球测序
- 3.三代测序
-
- 3.1.Oxford Nanopore公司:[纳米孔测序技术]
- 3.2.Pacific Bioscience公司:[单分子实时荧光测序技术SMRT]
- 3.3 GenoCare:我国乃至全亚洲第一台单分子荧光测序(三代测序仪)
- 4.一代、二代、三代测序技术的比较:
-
- 4.1 一代、二代测序的区别:
- 4.2 总结一代、二代、三代测序技术的区别
-
- 4.2.1一代测序
- 4.2.2二代测序
- 4.2.3三代测序
1.一代测序
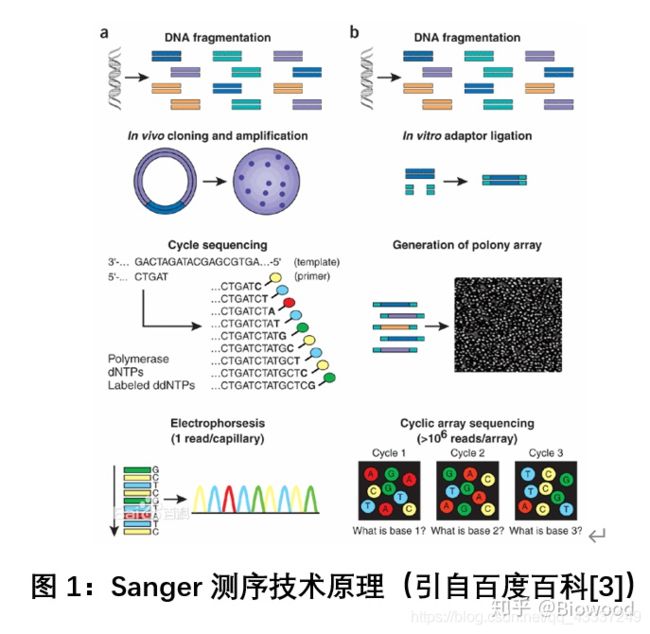
一代测序需要提及一名科学家Frederick Sanger ,Sanger是一位1918年出生于美国的生物化学家,曾经两度获得诺贝尔化学奖 。上世纪70年代末,他提出快速测定脱氧核糖核酸(DNA)序列的技术“双脱氧终止法”,也被称作“Sanger法测序技术,一代测序为合成终止测序
1.1 Sanger测序
Sanger测序:双脱氧链终止法:采用DNA复制原理。Sanger测序反应体系中包括目标DNA片段、脱氧三磷酸核苷酸(dNTP)、双脱氧三磷酸核苷酸(ddNTP)、测序引物及DNA聚合酶等。其技术核心是ddNTP的使用,由于缺少3’-OH基团,不具有与另一个dNTP连接形成磷酸二酯键的能力,这些ddNTP可用来中止DNA链的延伸。此外,这些ddNTP上连接有放射性同位素或荧光标记基团,因此可以被自动化的仪器或凝胶成像系统所检测到。
2.二代测序
2005年,罗氏推出了第一款二代测序仪罗氏454,生命科学开始进入高通量测序时代。后续随着Illumina系列测序平台的推出,极大降低了二代测序的价格,推动了高通量测序在生命科学各个研究领域的普及。目前,高通量测序已经成为一种常规研究方法,大量科研工作中均会用到。然而,为什么二代测序能实现高通量?为什么二代测序读长如此之短?为什么reads末端测序质量会降低?应该如何选择测序读长与打断片段的长度?想要回答这些问题,都需要详细了解二代测序的基本原理。
2.1.Illumina Solexa合成测序
使用克隆单分子阵列技术。首先将目的DNA片段打断成100-200bp,随机连接到固相基质上,经过Bst 聚合酶延伸和甲酸胺变性的桥PCR循环,生成大量的 DNA簇,之后的反应与Sanger法类似,每次延伸所产生的光信号被标准的阵列光学检测系统分析测序,下一次循环中把终止剂和荧光标记基团裂解掉,然后继续延伸 dNTP,实现了边合成边测序技术。

illumina测序基本步骤
1. 文库制备
将基因组DNA打成几百个碱基(或更短)的小片段,在片段的两个末端加上接头(adapter)。
2. 产生DNA簇
利用专利的芯片,其表面连接有一层单链引物,DNA片段变成单链后通过与芯片表面的引物碱基互补被一端“固定”在芯片上。另外一端(5’或3’)随机和附近的另外一个引物互补,也被“固定”住,形成“桥 (bridge) “。反复30轮扩增,每个单分子得到了1000倍扩增,成为单克隆DNA簇。DNA簇产生之后,扩增子被线性化,测序引物随后杂交在目标区域一侧的通用序列上。
3. 边合成边测序
Genome Analyzer系统应用了边合成边测序(Sequencing By Synthesis)的原理。加入改造过的DNA聚合酶和带有4种荧光标记的dNTP。 这些核苷酸是“可逆终止子”,因为3’羟基末端带有可化学切割的部分,它只容许每个循环掺入单个碱基。此时,用激光扫描反应板表面,读取每条模板序列第一轮反应所聚合上去的核苷酸种类。之后,将这些基团化学切割,恢复3’端粘性,继续聚合第二个核苷酸。如此继续下去,直到每条模板序列都完全被聚合为双链。这样,统计每轮收集到的荧光信号结果,就可以得知每个模板DNA片段的序列。目前的配对末端读长可达到2×50 bp,更长的读长也能实现,但错误率会增高。读长会受到多个引起信号衰减的因素所影响,如荧光标记的不完全切割。=
4. 数据分析
自动读取碱基,数据被转移到自动分析通道进行二次分析
2.2.Roche 454焦磷酸测序
使用边合成边测序技术,避免了Sanger法存在的宿主菌克隆问题。即首先将目的DNA片段打断成300-800bp的小片段,然后在5’端加上一个磷酸基团,并将3’端变成平端,再在两端加上衔接子组成目的DNA的样品文库。之后将目的DNA片段固定到磁珠上,将磁珠包被在单个油水混合小滴中进行独立的扩增,从而实现所有目的DNA片段进行平行扩增PCR。随后将这些DNA放入PCT反应板中共进行后继测序,这里面包含了化学光反应所需的各种酶和底物。测序开始时,将T、A、G、C按顺序循环单分子进入PTP 板,如果发生配对,则会释放一个焦磷酸盐分子,其在后续与ATP磷酸化酶和虫荧光素反应产生光信号,此光信号被捕获以确定碱基序列。
2.3.ABI SOLiD连接法测序
首先制备DNA文库,可以使用片段文库和配对末端文库。第二阶段与焦磷酸测序相同,加入磁珠等反应元件进行emPCR平行扩增,,不同的是该方法的磁珠只有1 µm。在连接测序中,底物是 8 个碱基的八聚体单链荧光探针,在5′末端分别标记了CY5、Teaxs Red、CY3、6-FAM这四种颜色的荧光染料。3′ 端的第 1、2 位碱基类别排序分别对应着一个固定的荧光染料,第 3、4、5 位碱基“n”是随机碱基,第 6、7、8 位碱基“z”是可以和任何碱基配对的特殊碱基。一次测序中包括了五轮连接反应,可以减小测序误差
2.4.BGI(华大基因):纳米球测序
1.BGI采用了 联合探针锚定聚合技术(cPAS)和改进的DNA纳米球(DNB)核心测序技术;
2. 单链DNA会缠绕成一个纳米球,纳米球可自动附着于有整齐的固定点的芯片上而不相互堆叠(即阵列技术),随后利用组合探针锚定连接法测序。
整个过程包含样本准备(形成末端修复的DNA片段)、片段扩增(加接头序列、形成单链DNA、成环、滚环扩增形成DNA纳米球)、纳米球附着于芯片并使用cPAS法测序。具体原理参见文献:Science 327, 78 (2010)
3.三代测序
第三代测序技术是指单分子测序技术,DNA测序时,不需要经过PCR扩增,实现了对每一条DNA分子的单独测序。第三代测序技术也叫从头测序技术,即单分子实时DNA测序,主要可以分为单分子荧光测序和纳米孔测序。
3.1.Oxford Nanopore公司:[纳米孔测序技术]
- 英国牛津纳米孔公司 新型纳米孔测序法(nanopore sequencing)是采用电泳技术,借助电泳驱动单个分子逐一通过纳米孔 来实现测序的。由于纳米孔的直径非常细小,仅允许单个核酸聚合物通过,四种核苷酸的空间构象不一样,因此当它们通过纳米孔时,所引起的电流变化不一样。由多个核苷酸组成的DNA或RNA链通过纳米孔时,检测通过纳米孔电流的强度变化,即可判断通过的核苷酸类型,从而进行实时测序。
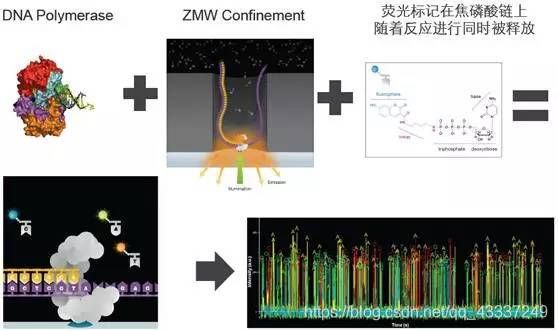
3.2.Pacific Bioscience公司:[单分子实时荧光测序技术SMRT]
- Pacific Bioscience公司(太平洋生物公司):SingleMolecule Real Time(SMRT)测序其通过将脱氧核苷酸用荧光标记,实时地记录荧光的强度变化。当荧光基团被掺入DNA链的时候,它的荧光就同时能在DNA链上探测到。当它与DNA链形成化学键的时候,它的荧光基团就被DNA聚合酶切除,荧光消失。这种荧光标记的脱氧核苷酸不会影响DNA聚合酶的活性,并且在荧光被切除之后,合成的DNA链和天然的DNA链完全一样。测序过程包括文库构建和上机两步。文库构建是将长片段DNA分子与测序接头连接成茎环结构,然后加上与接头互补的测序引物及DNA聚合酶。上机测序是将构建好的文库复合物载入SMRT Cell的纳米孔中,通常一个纳米孔固定一个DNA分子,DNA聚合酶通过共价连接的方式固定在纳米孔底部。
3.3 GenoCare:我国乃至全亚洲第一台单分子荧光测序(三代测序仪)
贺建奎教授团队研发成功第三代基因测序仪GenoCare,是当今世界上准确率最高(准确率达99.9985%)的第三代测序仪,并且率先在全世界取得政府医疗器械证备案、初步实现产业化并已应用于临床检测。该测序仪开发出基于全内反射先进光学的技术,可检测到单个DNA分子的微弱信号,读取DNA序列编码,为健康和疾病提供解读和诊断信息,其技术水平在亚洲乃至世界都居于领先地位
4.一代、二代、三代测序技术的比较:
4.1 一代、二代测序的区别:
- 一代:准确性高(~0.1%ER)、读长较长(约1kb)、通量低、价格昂贵。
- 二代:准确性较高(1~1.5%ER)、读长较长(150-600bp)、通量高、价格便宜。
一代测序为合成终止测序,而二代测序开创性的引入了可逆终止末端,从而实现边合成边测序(Sequencing by Synthesis)。二代测序在DNA复制过程中通过捕捉新添加的碱基所携带的特殊标记(一般为荧光分子标记)来确定DNA的序列,现有的技术平台主要包括Roche的454 FLX、Illumina的Miseq/Hiseq等。由于在二代测序中,单个DNA分子必须扩增成由相同DNA组成的基因簇,然后进行同步复制,来增强荧光信号强度从而读出DNA序列;而随着读长增长,基因簇复制的协同性降低,导致碱基测序质量下降,这严格限制了二代测序的读长(不超过500bp),因此,二代测序具有通量高、读长短的特点。二代测序适合扩增子测序(例如16S、18S、ITS的可变区),而基因组、宏基因组DNA则需要使用鸟枪法(Shotgun method)打断成小片段,测序完毕后再使用生物信息学方法进行拼接。
4.2 总结一代、二代、三代测序技术的区别
4.2.1一代测序
- 一代测序的最新仪器是ABI公司的3730XL。一般测序长度可达到1000bp。其主要原理是利用荧光标记的核苷酸,在PCR边合成的过程中边测序。由于四种核苷酸的荧光标记不一样,从而知道序列信息。
- 一代测序测序精度高,准确性高,但是相对其他平台,通量很低。即相同时间内产生的数据,一代远远少于二代和三代测序。
4.2.2二代测序
二代测序技术,也被称为高通量测序技术。它解决了一代测序只能测一条序列的缺陷。随着科研的不断深入,我们开始分析一个物种或样本中的所有序列信息,这个时候一代测序一次测一条的方式就无法满足我们的需求。二代测序技术就是在这样的情况下诞生的。之所以称其为高通量测序就是因为它一次能够同时测很多的序列。我们通过物理或是化学的方式将DNA随机打断成无数的小片段(250-300bp),之后通过建库(这里就不深入建库的原理了)富集了这些DNA片段。接下来将建完的库放入测序仪中测序,测序仪中有着可以让DNA片段附着的区域,每一个片段都有独立的附着区域,这样测序仪可以一次检测所有附着的DNA序列信息。最后通过生物信息学分析将小片段拼接成长片段。
二代测序特点:一次能够测大量的序列,但是片段被限制在了250-300bp,由于是通过序列的重叠区域进行拼接,所以有些序列可能被测了好多次。由于建库中利用了PCR富集序列,因此有一些量少的序列可能无法被大量扩增,造成一些信息的丢失,且PCR中有概率会引入错配碱基。所以三代测序就这样诞生了。
4.2.3三代测序
三代测序其实就是对二代测序的一个升级,简单来说就是它同样一次能测好多序列,但是测序的长度达到了10kb左右,并且不需要PCR富集序列,直接测序,这就解决了信息的丢失,以及碱基错配的问题。但目前来说三代测序依然有一定的缺陷:三代测序技术依赖DNA聚合酶的活性,且成本很高,目前的错误率在15%-40%,极大地高于二代测序技术的错误率不过好在三代的错误是完全随机发生的,可以靠覆盖度来纠错(但这要增加测序成本)。
参考文献:
1.Shendure J, Balasubramanian S, Church G M, et al. DNA sequencing at 40: past, present and future[J]. Nature, 2017.
注:部分内容整理自网络,仅供交流学习使用,如有侵权,请联系删除