matlab自带的mri数据集,探查三维 MRI 数据集的切片
步骤 1:加载和查看水平 MRI
此示例使用 MATLAB® 附带的用在 montage 和 immovie 的帮助示例中的 MRI 数据集。加载 mri.mat 会向工作区添加两个变量:D(128×128×1×27,uint8 类)和灰度颜色图 map(89×3,double 类)。
D 包含人体头盖骨 MRI 扫描数据的 27 个 128×128 水平切片。D 中的值的范围是从 0 到 88,因此需要颜色图来生成实用的可视图形。D 的维度与 montage 兼容。前两个维度是空间维度。第三个维度是颜色维度,大小为 1,因为它是颜色图的索引。对于 RGB 图像序列,size(D,3) 为 3。第四个维度是时序维度(和任何图像序列一样),但在本例中,它也是空间维度。因此,D 中有三个空间维度,我们可以使用 imtransform 或 tformarray 将水平切片转换为矢状切片(显示头部侧面的视图)或冠状(正面)切片(显示头部正面或背面的视图)。
D 的空间维度排序如下:
维度 1:头部从前到后(从吻突/前侧到尾椎/后侧)
维度 2:头部从左到右
维度 4:头部从底到顶(从下到上)。
一个重要因素是三个维度上的采样间隔不同:沿垂直维度 (4) 的采样间隔比沿水平维度的采样间隔宽 2.5 倍。
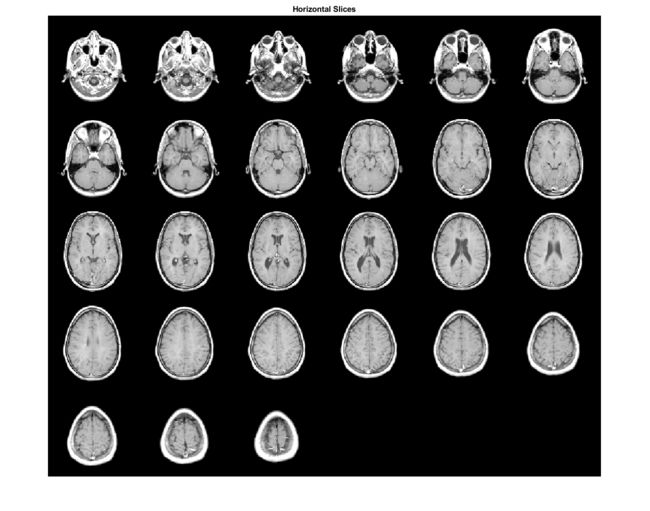
加载 MRI 数据集,将 27 个水平切片以蒙太奇形式进行查看。
load mri;
montage(D,map)
title('Horizontal Slices');
步骤 2:使用 IMTRANSFORM 从水平切片中提取矢状切片
我们可以通过提取 D 的子集,并对其进行变换以处理 D 维度的不同采样间隔和空间方向,从而构建 MRI 数据的正中矢状切片。
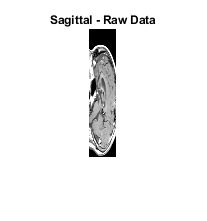
以下语句提取正中矢状切片所需的所有数据。
M1 = D(:,64,:,:); size(M1)
ans = 1×4
128 1 1 27
但是,我们无法以图像形式查看 M1,因为它是 128×1×1×27。reshape(或 squeeze)可以将 M1 转换为可使用 imshow 查看的 128×27 图像。
M2 = reshape(M1,[128 27]); size(M2)
ans = 1×2
128 27
figure, imshow(M2,map);
title('Sagittal - Raw Data');
M2 中的维度的排序如下:
维度 1:头部从前到后(从吻突到尾椎)
维度 2:头部从底到顶(从下到上)。
我们可以通过变换 M2,更改其方向并将垂直(下-上)维度的采样增加为 2.5 倍,使采样间隔在所有三个空间维度上相等,从而获得更令人满意的视图。我们可以从转置开始分多个步骤执行此过程,但以下仿射变换可以实现单步变换并且更节省内存。
T0 = maketform('affine',[0 -2.5; 1 0; 0 0]);
传递给 maketform 的矩阵的上 2×2 块 ([0 -2.5;1 0]) 结合使用了旋转和缩放。变换后可得到以下维度:
维度 1:头部从顶到底(从上到下)。
维度 2:头部从前到后(从吻突到尾椎)
以下调用
imtransform(M2,T0,'cubic')
可将 T 应用于 M2,并足以在沿从顶到底方向插值时提供良好的分辨率。不过,从前到后方向无需执行三次插值,因为沿(输出)维度 2 不会进行重采样。因此,我们在此维度中指定最近邻点重采样,可获得更高的效率和相同的结果。
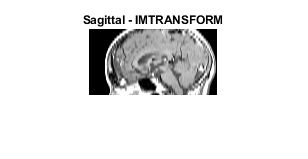
R2 = makeresampler({'cubic','nearest'},'fill');
M3 = imtransform(M2,T0,R2);
figure, imshow(M3,map);
title('Sagittal - IMTRANSFORM')
步骤 3:使用 TFORMARRAY 从水平切片中提取矢状切片
在此步骤中,我们获得的结果与步骤 2 相同,只是使用了 tformarray 通过一次运算将三个空间维度转换为二个空间维度。步骤 2 确实也是从开始的具有三个空间维度的数组转换为最后的具有两个空间维度的数组,但中间生成了二维图像(M1 和 M2),以便于调用 imtransform 来创建 M3。如果我们使用 tformarray 代替 imtransform,则这些中间图像是不必要的。imtransform 对于二维到二维的变换非常方便,但 tformarray 支持从 N 维到 M 维的变换,其中 M 不必等于 N。
通过其 TDIMS_A 参数,tformarray 允许我们定义输入数组的置换。由于我们要创建具有以下维度的图像:
维度 1:从上到下(颠倒的原始维度 4)
维度 2:尾椎到吻突(原始维度 1)
并通过原始维度 2 仅提取单个矢状平面,可指定 tdims_a = [4 1 2]。我们通过变换组合来创建 tform,首先进行二维仿射变换 T1,将维度 1(新)缩放 -2.5 倍,并平移 68.5 以使数组坐标为正值。组合的第二部分是自定义变换 T2,通过很简单的 INVERSE_FCN 提取第 64 个矢状平面。
T1 = maketform('affine',[-2.5 0; 0 1; 68.5 0]);
inverseFcn = @(X,t) [X repmat(t.tdata,[size(X,1) 1])];
T2 = maketform('custom',3,2,[],inverseFcn,64);
Tc = maketform('composite',T1,T2);
请注意,T2 和 Tc 将三维输入转换为二维输入。
我们使用与以前相同的方法进行重采样,但包括第三个维度。
R3 = makeresampler({'cubic','nearest','nearest'},'fill');
tformarray 通过一个步骤将 D 的三个空间维度变换为二维输出。我们的输出图像是 66×128,在垂直(下-上)方向上最初的 27 个平面扩展到 66 个。
M4 = tformarray(D,Tc,R3,[4 1 2],[1 2],[66 128],[],0);
结果与先前的 imtransform 的输出相同。
figure, imshow(M4,map);
title('Sagittal - TFORMARRAY');
步骤 4:创建和显示矢状切片
我们创建一个四维数组(第三个维度是颜色维度),它可用于生成一个图像序列,该图像序列从第 30 个平面开始,从左到右每隔一个平面取一帧,总共有 35 个帧。变换后的数组具有以下维度:
维度 1:从顶到底(从上到下)
维度 2:从前到后(从吻部到尾椎)
维度 4:从左到右。
与上一个步骤一样,我们使用 TDIMS_A = [4 1 2] 置换输入数组,然后再翻转和重新缩放/重采样垂直维度。我们的仿射变换与上面的 T1 相同,不同之处在于添加了第三个维度,其中 (3,3) 元素为 0.5、(4,3) 元素为 -14,以将 30、32、...98 映射到 1、2、...、35。这可将 35 个帧居中置于正中矢状切片上。
T3 = maketform('affine',[-2.5 0 0; 0 1 0; 0 0 0.5; 68.5 0 -14]);
在我们调用 tformarray 时,TSIZE_B = [66 128 35] 现在将 35 个帧包含在从左到右的第 4 个维度(即第三个变换维度)中。重采样器保持不变。
S = tformarray(D,T3,R3,[4 1 2],[1 2 4],[66 128 35],[],0);
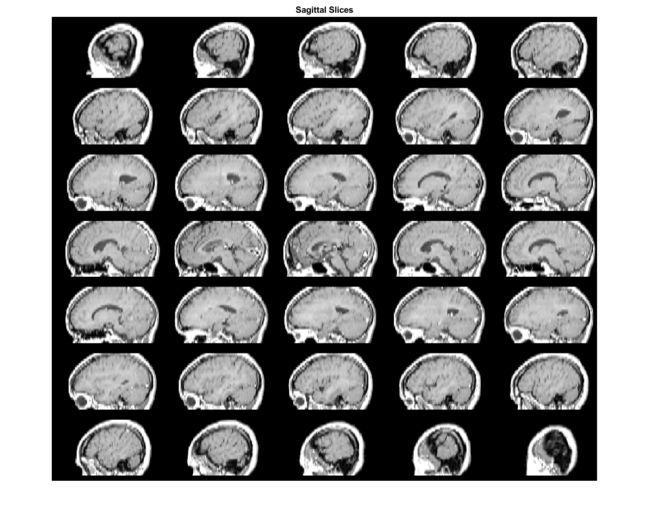
以蒙太奇方式查看矢状切片(稍微填充数组以分隔蒙太奇的元素)。
S2 = padarray(S,[6 0 0 0],0,'both');
figure, montage(S2,map)
title('Sagittal Slices');
步骤 5:创建和显示冠状切片
构造冠状切片与构造矢状切片几乎完全相同。我们将 TDIMS_A 从 [4 1 2] 更改为 [4 2 1]。创建一个包含 45 个帧的系列,从第 8 个平面开始,从后向前移动,每两帧跳过一帧。输出数组的维度排序如下:
维度 1:从顶到底(从上到下)
维度 2:从左到右
维度 4:从后到前(从尾椎到吻突)。
T4 = maketform('affine',[-2.5 0 0; 0 1 0; 0 0 -0.5; 68.5 0 61]);
在 tformarray 调用中,TSIZE_B = [66 128 48] 分别指定垂直、从一侧到另一侧和从前到后的维度。重采样器保持不变。
C = tformarray(D,T4,R3,[4 2 1],[1 2 4],[66 128 45],[],0);
请注意,步骤 3、4 和 5 中的所有数组置换和翻转均作为 tformarray 运算的一部分来处理。
以蒙太奇方式查看冠状切片(稍微填充数组以分离蒙太奇的元素)。
C2 = padarray(C,[6 0 0 0],0,'both');
figure, montage(C2,map)
title('Coronal Slices');