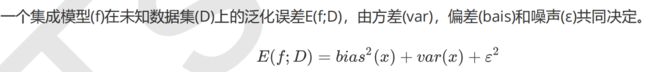【2 - 随机森林 - 案例部分:随机森林在乳腺癌数据上的调参】菜菜sklearn机器学习
课程地址:《菜菜的机器学习sklearn课堂》_哔哩哔哩_bilibili
- 第一期:sklearn入门 & 决策树在sklearn中的实现
- 第二期:随机森林在sklearn中的实现
- 第三期:sklearn中的数据预处理和特征工程
- 第四期:sklearn中的降维算法PCA和SVD
- 第五期:sklearn中的逻辑回归
- 第六期:sklearn中的聚类算法K-Means
- 第七期:sklearn中的支持向量机SVM(上)
- 第八期:sklearn中的支持向量机SVM(下)
- 第九期:sklearn中的线性回归大家族
- 第十期:sklearn中的朴素贝叶斯
- 第十一期:sklearn与XGBoost
- 第十二期:sklearn中的神经网络
目录
机器学习中调参的基本思想
(一)泛化误差
(二)偏差(bias)-方差(variance)困境
案例:随机森林在乳腺癌数据上的调参
(一)导入需要的库
(二)导入数据集,探索数据
(三)进行一次简单的建模,看看模型本身在数据集上的效果
(四)随机森林调整的第一步:无论如何先来调n_estimators
(五)在确定好的范围内,进一步细化学习曲线
(六)为网格搜索做准备,书写网格搜索的参数
(七)开始按照参数对模型整体准确率的影响程度进行调参,首先调整max_depth
(八)调整max_features
(九)调整min_samples_leaf
(十)调整min_samples_split
(十一)尝试一下criterion
(十二)调整完毕,总结出模型的最佳参数
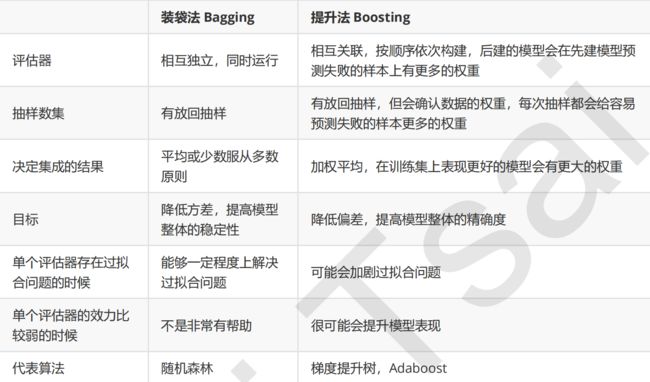
Bagging vs Boosting
机器学习中调参的基本思想
通过画学习曲线,或者网格搜索,我们能够探索到调参边缘
正确的模型调参思路:
- 目标是提升某个模型评估指标,比如对于随机森林来说,想要提升的是模型在未知数据上的准确率(由 score 或 oob_score_ 来衡量)
- 模型在未知数据上的准确率受什么因素影响?在机器学习中,用来衡量模型在未知数据上的准确率的指标,叫泛化误差(genelization error)
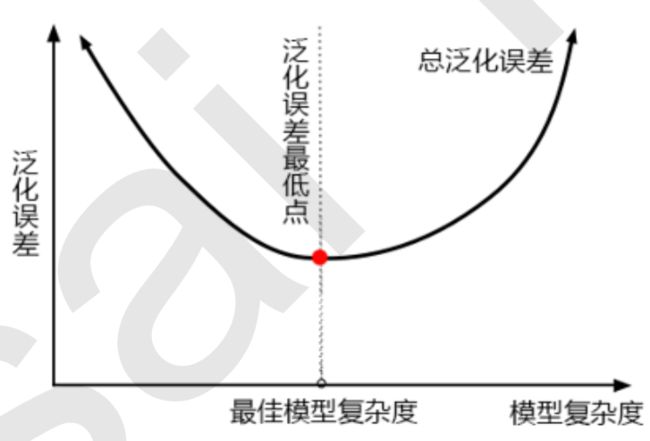
(一)泛化误差
当模型在未知数据(测试集或袋外数据)上表现糟糕时,即模型的泛化程度不够,泛化误差大,模型效果不好
泛化误差受到模型的结构(复杂度)影响:
- 当模型太复杂,模型就会过拟合,泛化能力就不够,所以泛化误差大
- 当模型太简单,模型就会欠拟合,拟合能力就不够,所以泛化误差大
- 只有当模型的复杂度刚刚好,才能够达到泛化误差最小的目标
- 对树模型来说,树越茂盛,深度越深,枝叶越多,模型就越复杂,所以树模型是天生位于图的右上角的模型
- 随机森林以树模型为基础,所以随机森林也是天生复杂度高的模型。随机森林的参数都是向着一个目标去:减少模型的复杂度,把模型往图像的左边移动,防止过拟合
- 但是也有天生处于图像左边的随机森林,所以在调参之前,要先判断模型现在究竟处于图像的哪一边
(二)偏差(bias)-方差(variance)困境
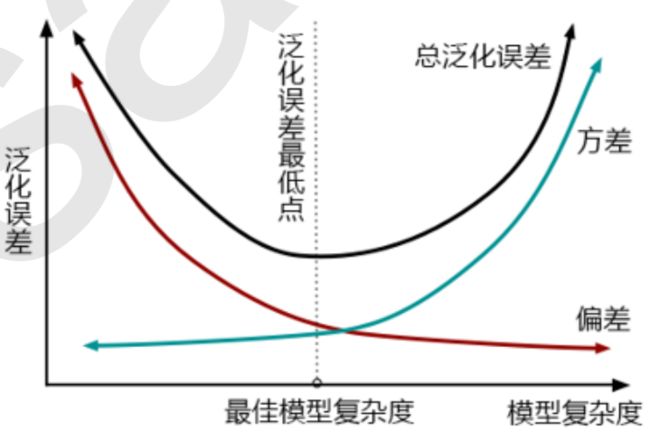
四点结论:
- 模型太复杂或太简单,都会让泛化误差高,追求的是位于中间的平衡点
- 模型太复杂就会过拟合,太简单就会欠拟合
- 对树模型和树的集成模型来说,树的深度越深,枝叶越多,模型越复杂(剪枝是降低模型的复杂度)
- 树模型和树的集成模型的目标,都是减少模型复杂度,把模型往图像的左边移动
随机森林的调参方向是降低复杂度,故将那些对复杂度影响巨大的参数挑选出来,研究它们的单调性,调整那些能最大限度降低复杂度的参数;对于那些不单调的参数,或者反而会让复杂度升高的参数,视情况使用
一个好的模型,要对大多数未知数据都预测的准又稳,即当偏差和方差都很低的时候,模型的泛化误差就小,在未知数据上的准确率就高。然而,方差和偏差是此消彼长的,不可能同时达到最小值,调参的目标是达到方差和偏差的平衡
- 偏差:模型的预测值与真实值之间的差异,模型越精确,偏差越低
- 方差:模型每一次输出结果与模型预测值的平均水平之间的误差,模型越稳定,方差越低
随机森林的基评估器都拥有较低的偏差和较高的方差,因为决策树本身是预测比较准、比较容易过拟合的模型,装袋法本身也要求基分类器的准确率必须要有50%以上。所以以随机森林为代表的装袋法的训练过程,旨在降低方差(即降低模型复杂度),故随机森林参数的默认设定都是假设模型本身在泛化误差最低点的右边
案例:随机森林在乳腺癌数据上的调参
- 基于方差和偏差的调参方法
- 乳腺癌数据是sklearn自带的分类数据之一
(一)导入需要的库
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import cross_val_score
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np(二)导入数据集,探索数据
data = load_breast_cancer()
data.data.shape # (569, 30)
data.target # 二分类数据0/1
# 可以看到,乳腺癌数据集有569条记录,30个特征,单看维度虽然不算太高,但是样本量非常少,过拟合的情况可能存在(三)进行一次简单的建模,看看模型本身在数据集上的效果
rfc = RandomForestClassifier(n_estimators=100,random_state=90)
score_pre = cross_val_score(rfc,data.data,data.target,cv=10).mean() #交叉验证的分类默认scoring='accuracy'
score_pre
#这里可以看到,随机森林在乳腺癌数据上的表现本就还不错,在现实数据集上,基本上不可能什么都不调就看到95%以上的准确率调参顺序:n_estimators —> max_depth —> min_samples_leaf —> min_samples_split —> max_features —> criterion
(四)随机森林调整的第一步:无论如何先来调n_estimators
"""
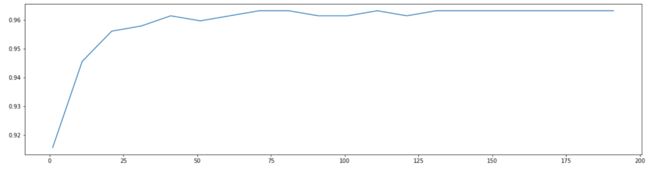
在这里我们选择学习曲线,可以使用网格搜索吗?可以,但是只有学习曲线,才能看见趋势
我个人的倾向是,要看见n_estimators在什么取值开始变得平稳,是否一直推动模型整体准确率的上升等信息
第一次的学习曲线,可以先用来帮助我们划定范围,我们取每十个数作为一个阶段,来观察n_estimators的变化如何引起模型整体准确率的变化
"""
scorel = []
for i in range(0,200,10): # 0 10 20 30 ... 200
rfc = RandomForestClassifier(n_estimators=i+1,
n_jobs=-1, # 使用CPU里的所有core
random_state=90)
score = cross_val_score(rfc,data.data,data.target,cv=10).mean()
scorel.append(score)
# list.index([object]) 返回这个object在列表list中的索引
print(max(scorel),(scorel.index(max(scorel))*10)+1)
# scorel.index(max(scorel))返回scorel列表中最大值的索引
# scorel.index(max(scorel))*10)+1对应n_estimators的取值
plt.figure(figsize=[20,5])
plt.plot(range(1,201,10),scorel)
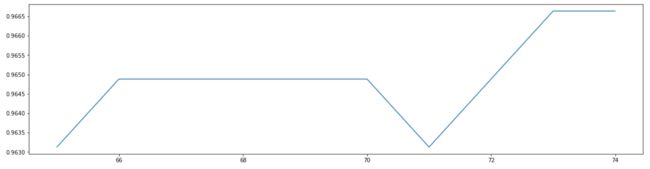
plt.show()(五)在确定好的范围内,进一步细化学习曲线
scorel = []
for i in range(65,75): # 在上一步得出的71结果附近再细化探索
rfc = RandomForestClassifier(n_estimators=i,
n_jobs=-1,
random_state=90)
score = cross_val_score(rfc,data.data,data.target,cv=10).mean()
scorel.append(score)
print(max(scorel),([*range(65,75)][scorel.index(max(scorel))]))
plt.figure(figsize=[20,5])
plt.plot(range(65,75),scorel)
plt.show()调整 n_estimators 的效果显著,模型的准确率立刻上升了0.003
接下来就使用复杂度-泛化误差方法(方差-偏差方法)和网格搜索对参数一个个进行调整(同时调整多个参数,会让我们无法理解参数的组合是怎么得来的,所以即便网格搜索调出来的结果不好,也不知道从哪里去改)
(六)为网格搜索做准备,书写网格搜索的参数
有一些参数是没有参照的,很难说清一个范围,这种情况下我们使用学习曲线,看趋势。从曲线跑出的结果中选取一个更小的区间,再跑曲线
param_grid = {'n_estimators':np.arange(0, 200, 10)}
param_grid = {'max_depth':np.arange(1, 20, 1)}
param_grid = {'max_leaf_nodes':np.arange(25,50,1)}
# 对于大型数据集,可以尝试从1000来构建,先输入1000,每100个叶子一个区间,再逐渐缩小范围有一些参数是可以找到一个范围的,或者说我们知道它们的取值和随着它们的取值,模型的整体准确率会如何变化,这样的参数我们就可以直接跑网格搜索
param_grid = {'criterion':['gini', 'entropy']}
param_grid = {'min_samples_split':np.arange(2, 2+20, 1)} # 默认值2
param_grid = {'min_samples_leaf':np.arange(1, 1+10, 1)} # 默认值1
# 默认值是特征数量开平方,该值要么是设置的最大值,要么是最小值
# 即范围是从0-该值,或从该值-最大特征量
param_grid = {'max_features':np.arange(5,30,1)}
(七)开始按照参数对模型整体准确率的影响程度进行调参,首先调整max_depth
#调整max_depth
param_grid = {'max_depth':np.arange(1, 20, 1)}
# 一般根据数据的大小来进行一个试探,乳腺癌数据很小,所以可以采用1~10,或者1~20这样的试探
# 但对于像digit recognition那样的大型数据来说,我们应该尝试30~50层深度(或许还不足够
# 更应该画出学习曲线,来观察深度对模型的影响
rfc = RandomForestClassifier(n_estimators=73
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10) #网格搜索
GS.fit(data.data,data.target)GS.best_params_ #显示调整出来的最佳参数GS.best_score_ #返回调整好的最佳参数对应的准确率- 将max_depth设置为有限之后,模型的准确率不变
- 限制max_depth,是让模型变得简单,把模型向左推
- 通常来说,随机森林应该在泛化误差最低点的右边,树模型应该倾向于过拟合,而不是拟合不足。这和数据集本身有关,也有可能是我们调整的n_estimators对于数据集来说太大,因此将模型拉到泛化误差最低点了
当模型位于图像左边时,需要增加模型复杂度(增加方差,减少偏差),因此max_depth应尽量大,min_samples_leaf和min_samples_split应尽量小,这三个参数是剪枝参数(减小复杂度的参数)
(八)调整max_features
max_features是唯一一个既能够将模型往左(低方差高偏差)推,也能够将模型往右(高方差低偏差)推的参数。我们需要根据调参前模型所在的位置(在泛化误差最低点的左边还是右边)来决定我们要将max_features往哪边调
max_features的默认最小值是sqrt(n_features)
# 调整max_features,总共有30个特征,默认值为根号30≈5.
param_grid = {'max_features':np.arange(5,30,1)} # 往复杂度高的方向调
rfc = RandomForestClassifier(n_estimators=73
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)
GS.best_params_
GS.best_score_max_features降低之后,模型的准确率提升了。这说明,我们把模型往左推,模型的泛化误差降低了,说明模型在曲线的右边
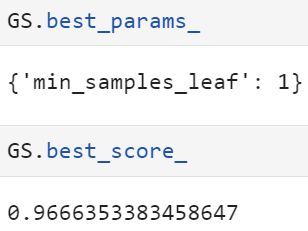
(九)调整min_samples_leaf
#调整min_samples_leaf
param_grid={'min_samples_leaf':np.arange(1, 1+10, 1)} # 默认为1(最大复杂度)
#对于min_samples_split和min_samples_leaf,一般是从他们的最小值开始向上增加10或20
#面对高维度高样本量数据,如果不放心,也可以直接+50,对于大型数据,可能需要200~300的范围
#如果调整的时候发现准确率无论如何都上不来,那可以放心大胆调一个很大的数据,大力限制模型的复杂度
rfc = RandomForestClassifier(n_estimators=73
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)
GS.best_params_
GS.best_score_可以看见,网格搜索返回了min_samples_leaf的最小值,且模型整体的准确率还降低了,即参数把模型向左推,但是模型的泛化误差上升了。在这种情况下,不要把这个参数设置起来,默认即可
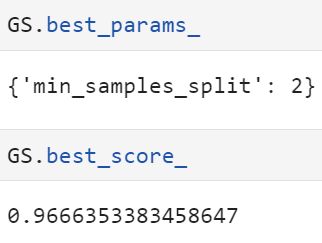
(十)调整min_samples_split
#调整min_samples_split
param_grid={'min_samples_split':np.arange(2, 2+20, 1)} # 默认为2(最大复杂度)
rfc = RandomForestClassifier(n_estimators=73
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)
GS.best_params_
GS.best_score_和min_samples_leaf一样的结果,返回最小值并且模型整体的准确率降低了
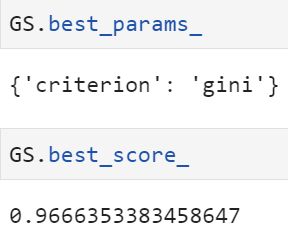
(十一)尝试一下criterion
#调整Criterion
param_grid = {'criterion':['gini', 'entropy']}
rfc = RandomForestClassifier(n_estimators=73
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)
GS.best_params_
GS.best_score_(十二)调整完毕,总结出模型的最佳参数
rfc = RandomForestClassifier(n_estimators=73,max_features=2,random_state=90)
score = cross_val_score(rfc,data.data,data.target,cv=10).mean()
score
score - score_pre # 调参前后准确率的变动在整个调参过程中,首先调整n_estimators(无论如何这都是第一步),然后调整max_depth,通过max_depth产生的结果来判断模型位于复杂度-泛化误差图像的哪一边,从而选择我们应该调整的参数和调参的方向
也可以画学习曲线来观察参数会如何影响准确率,选取学习曲线中单调的部分来放大研究,学习曲线的拐点也许就是我们追求的、最佳复杂度对应的泛化误差最低点(也是方差和偏差的平衡点)