pytorch geometric教程四 利用NeighorSampler实现节点维度的mini-batch + GraphSAGE样例
pytorch geometric教程四 利用NeighorSampler实现节点维度的mini-batch + GraphSAGE样例
- class NeighborSampler
-
- 核心想法
- 返回结果
- 参数
- NeighborSampler在GraphSAGE代码中的使用
-
- 模型训练
-
- train_loader
- train
- model中的forward()函数
- 模型推断:
-
- subgraph_loader, inference函数和test()
- NeighborSampler工作原理&具体实例
-
- batch_size =1 ,采样邻居数小于邻居数
- batch_size =1 ,采样邻居数大于邻居数
- batch_size = [2, 2]
- 实战代码
PyG的官方文档中有mini-batch和Advanced mini-batching两部分内容,但实现的都是图维度的mini-batch。如何像GraphSAGE paper中对minibatch的节点进行邻居采样并训练模型,使得大规模全连接图的GNNs模型训练成为可能,PyG是通过torch_geometric.loader.NeighborSampler实现的(早一点版本是torch_geometric.data.NeighborSampler)。
需要注明的一点是,这篇文章中虽然举了SAGEConv的代码样例,但只要卷积层支持bipartite图,大家举一反三,就可以与NeighborSampler结合使用,实现节点维度的mini-batch模型训练与推断。
class NeighborSampler
核心想法
NeighborSampler的核心想法是,给定mini-batch的节点和图卷积的层数L,以及每一层需要采样的邻居数目sizes,依次从第一层到第L层,对每一层进行邻居采样并返回一个bipartite子图。sizes是一个L长度的list,包含每一层需要采样的邻居个数。以下是主要逻辑的归纳:
For i in L:
- 第
1层使用初始minibatch的节点进行邻居采样,返回采样结果。 - 第
i (i>0)层,使用上层采样中涉及到的所有节点进行邻居采样,返回采样结果。
L层采样完成后,返回结果(batch_size, n_id, adjs),其中batch_size就是mini-batch的节点数目,n_id是包含所有在L层卷积中遇到的节点的list,且target节点在n_id前几位。adjs是一个list,包含了从第L层到第1层采样的结果,所以adjs中的子图是从大到小的。每一层采样返回的结果具体形式为 (edge_index, e_id, size)。其中edge_index是采样得到的bipartite子图中source节点到target节点的边。e_id是edge_index的边在原始大图中的IDs,size就是bipartite子图的shape。
以下是一个2层采样的示意图,注意在第2层采样的时候,使用了第1层中涉及到的所有节点,包括出发点。
![]()
返回结果
整理一下, NeighborSampler的返回结果如下:
batch_sizen_id:L层采样中遇到的所有的节点的list,其中target节点在list最前端adjs:第L层到第1层采样结果的listedge_index:采样得到的bipartite子图中source节点到target节点的边e_id:edge_index的边在原始大图中的IDssize:bipartite子图的shape
参数
class NeighborSampler(torch.utils.data.DataLoader):
def __init__(self, edge_index: Union[Tensor, SparseTensor],
sizes: List[int], node_idx: Optional[Tensor] = None,
num_nodes: Optional[int] = None, return_e_id: bool = True,
transform: Callable = None, **kwargs):
edge_index (Tensor or SparseTensor):图的边信息,可以是Tensor,也可以是SparseTensor。sizes ([int]):每一层需要采样的邻居数目,如果是-1的话,选取所有的邻居。node_idx (LongTensor, optional):提供需要被采样节点的信息,比如模型训练的时候,只给出数据集train中的节点。在预测的时候,使用None,考虑所有的节点。num_nodes: Optional[int] = None:图中节点的数目,可选参数。return_e_id: bool = True:当设为False的时候,不会返回partite子图的边在原图中的IDs。transform**kwargs:NeighborSampler是torch.utils.data.DataLoader的子类,所以父类DataLoader的参数NeighborSampler都可以使用,比如:batch_size,shuffle,num_workers。
NeighborSampler在GraphSAGE代码中的使用
先看一下NeighborSampler如何和卷积一起使用,后面再看它具体是如何工作的。
PyG关于GraphSAGE给了两个代码样例reddit和ogbn_prodcuts_sage,下面具体解释一下reddit代码中NeighborSampler的相关部分,我删掉了一些不太重要的代码内容。
模型训练
train_loader
首先代码里定义了模型训练时的数据加载器train_loader。 node_idx=data.train_mask指定只对训练集的节点进行邻居采样,sizes=[25, 10]指明了这是一个两层的卷积,第一层卷积采样邻居数目25,第二层卷积采样邻居数目10。batch_size=1024指定了mini-batch的节点数目,每次只对1024个节点进行采样。
train_loader = NeighborSampler(data.edge_index, node_idx=data.train_mask,
sizes=[25, 10], batch_size=1024, shuffle=True,
num_workers=12)
train
train_loader每次返回一个batch_size节点邻居采样的结果,其形式是(batch_size, n_id, adjs),其中n_id是采样过程中涉及的所有节点的id,也是adjs中涉及的所有节点,因此x[n_id]是所有相关节点的特征。而且x[n_id]相当于做了一次映射,x[n_id]中第i行就是adjs中i节点的特征(关于这一点,会在后面NeighborSampler工作原理部分详述)。adjs是包含了所有bipartite子图边信息的list。所以model(x[n_id], adjs)传入了所有bipartite子图的节点特征和边信息。
另外NeighborSampler是在CPU中完成的,所以返回的结果都在CPU上。如果用GPU训练模型,要记得将loader的结果放到GPU上。
def train(epoch):
model.train()
total_loss = total_correct = 0
for batch_size, n_id, adjs in train_loader:
# `adjs` holds a list of `(edge_index, e_id, size)` tuples.
adjs = [adj.to(device) for adj in adjs]
optimizer.zero_grad()
out = model(x[n_id], adjs)
loss = F.nll_loss(out, y[n_id[:batch_size]])
loss.backward()
optimizer.step()
total_loss += float(loss)
total_correct += int(out.argmax(dim=-1).eq(y[n_id[:batch_size]]).sum())
loss = total_loss / len(train_loader)
approx_acc = total_correct / int(data.train_mask.sum())
return loss, approx_acc
model中的forward()函数
forward函数依次实现了从第L层到第1层采样得到的bipartite子图的卷积。
adjs包含L层邻居采样的bipartite子图:(edge_index, e_id, size)。在上一个教程中讲过了,SAGEConv是支持bipartite图的。对bipartite图进行卷积时,输入的x是一个tuple: (x_source, x_target)。上面提到过,n_id是包含所有在L层卷积中遇到的节点的list,且target节点在n_id前几位。而bipartite图的size是(num_of_source_nodes, num_of_target_nodes),因此对每一层的bipartite图都有x_target = x[:size[1]] 。所以 self.convs[i]((x, x_target), edge_index)实现了对一层bipartite图的卷积。
class SAGE(torch.nn.Module):
def __init__(self, in_channels, hidden_channels, out_channels, num_layers):
super(SAGE, self).__init__()
self.num_layers = num_layers
...
def forward(self, x, adjs):
for i, (edge_index, _, size) in enumerate(adjs):
x_target = x[:size[1]] # Target nodes are always placed first.
x = self.convs[i]((x, x_target), edge_index)
if i != self.num_layers - 1:
x = F.relu(x)
x = F.dropout(x, p=0.5, training=self.training)
return x.log_softmax(dim=-1)
模型推断:
subgraph_loader, inference函数和test()
做推断时,不再对邻居进行采样,而是取所有的邻居,因此subgraph_loader中的sizes设成了[-1],取所有的一阶邻居。但是这里的SAGE模型是一个两层的卷积层,为什么不取所有的二阶邻居,将sizes设成了[-1, -1]呢?我们看一下这个代码样例中的inference函数。
subgraph_loader = NeighborSampler(data.edge_index, node_idx=None, sizes=[-1],
batch_size=1024, shuffle=False,
num_workers=12)
class SAGE(torch.nn.Module):
def __init__(self, in_channels, hidden_channels, out_channels, num_layers):
super(SAGE, self).__init__()
self.num_layers = num_layers
...
def inference(self, x_all):
for i in range(self.num_layers):
xs = []
for batch_size, n_id, adj in subgraph_loader:
edge_index, _, size = adj.to(device)
x = x_all[n_id].to(device) #使用上一层卷积跟新过的embedding, x_all
x_target = x[:size[1]]
x = self.convs[i]((x, x_target), edge_index)
if i != self.num_layers - 1:
x = F.relu(x)
xs.append(x.cpu())
x_all = torch.cat(xs, dim=0) # 使用这一层卷积得到的embedding来更新x_all
return x_all
注意到其中有一个for i in range(self.num_layers)的循环,总结inference在做的事情就是:
for i in L:
对于所有节点,利用一阶邻居更新embedding
不是取所有的n阶邻居,计算一次就得到节点最终的embedding,这里用的技巧是,只取所有一阶邻居,但是迭代跟新n次。这两种方式的结果是相同的。
但是为什么要使用这种曲折的方式,而不是直接取n阶邻居呢?
首先训练的时候为什么要对邻居采样,就是因为整张图的计算非常消耗内存。所以设计了minibatch邻居采样的计算方式,同时实现了更快收敛和内存节省。而推断如果取全部n阶邻居,随着邻居节点数的指数级增多,也会使内存消耗指数级增加。对于比较稠密的图,或者图中存在度数很大的节点的时候,n阶邻居可能相当于把整张图放了进来,或许会出现out of memory的情况。 所以只取一阶邻居,但是迭代n次,是计算效率更高,内存消耗更小的方法。尤其对于比较稠密的图,这一点体现更为明显。
NeighborSampler工作原理&具体实例
上文只是简略讲了NeighborSampler的工作原理,这里用几个实例,让大家更清楚地理解其中的细节。
首先用networkx建以下一张图:
import networkx as nx
graph = nx.Graph()
graph.add_edges_from([(0,1), (1,2), (1,3), (2,3), (3,4), (4,2)])
nx.draw_kamada_kawai(graph, with_labels=True)
from torch_geometric.data.data import Data
from torch_geometric.utils import from_networkx
data = from_networkx(graph)
data.edge_index
>>> tensor([[0, 1, 1, 1, 2, 2, 2, 3, 3, 3, 4, 4],
[1, 0, 2, 3, 1, 3, 4, 1, 2, 4, 2, 3]])
batch_size =1 ,采样邻居数小于邻居数
from torch_geometric.data import NeighborSampler
loader = NeighborSampler(edge_index=data.edge_index, sizes=[2], node_idx=torch.tensor([2]), batch_size=1)
next(iter(loader))
>>> (1,
tensor([2, 3, 1]),
EdgeIndex(edge_index=tensor([[1, 2],
[0, 0]]), e_id=tensor([8, 2]), size=(3, 1)))
以上代码对2号节点取两个邻居。n_id: tensor([2, 3, 1])是遇到的所有节点,target节点在最前面,是2号节点,3, 1是采样到的邻居。edge_index=tensor([[1, 2], [0, 0]])是采样得到的bipartite子图。n_id中的index对应edge_index中的数值。edge_index[1]中是target节点,bipartite子图是从target节点开始计数的,所以n_id里面永远是target节点在前几位。另外size[0]是source节点的数目,size[1]是target节点的数目,所以n_id[size[1]]可以获取target节点在原图中的id。看以下示意图:
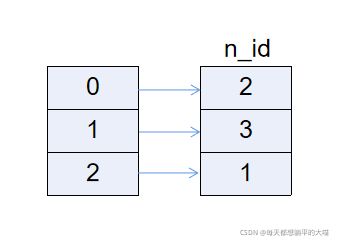
知道了bipartite子图中的节点对应原图哪个节点后,还可以将bipartite子图中的边对应到原图中的边。看以下示意图:

边[3,2]和边[1,2]分别是原图中的第8,第2条边,和返回的e_id相同。
batch_size =1 ,采样邻居数大于邻居数
以下结果可以看出,当采样邻居数大于邻居数的时候,NeighborSampler不会对邻居进行随机填充。这是因为在源码中,作者将采样的replace设置成了False。
from torch_geometric.data import NeighborSampler
loader = NeighborSampler(edge_index=data.edge_index, sizes=[4], node_idx=torch.tensor([2]), batch_size=1)
next(iter(loader))
>>>(1,
tensor([2, 4, 1, 3]),
EdgeIndex(edge_index=tensor([[1, 2, 3],
[0, 0, 0]]), e_id=tensor([10, 2, 8]), size=(4, 1)))
batch_size = [2, 2]
from torch_geometric.data import NeighborSampler
loader = NeighborSampler(edge_index=data.edge_index, sizes=[2, 2], node_idx=torch.tensor([2]), batch_size=1)
next(iter(loader))
>>> (1,
tensor([2, 4, 1, 3, 0]),
[EdgeIndex(edge_index=tensor([[2, 3, 0, 3, 0, 4],
[0, 0, 1, 1, 2, 2]]), e_id=tensor([2, 8, 6, 9, 4, 0]), size=(5, 3)),
EdgeIndex(edge_index=tensor([[1, 2],
[0, 0]]), e_id=tensor([10, 2]), size=(3, 1))])
其中EdgeIndex(edge_index=tensor([[1, 2], [0, 0]]), e_id=tensor([10, 2]), size=(3, 1))是第一层采样得到的bipartite图, EdgeIndex(edge_index=tensor([[2, 3, 0, 3, 0, 4], [0, 0, 1, 1, 2, 2]]), e_id=tensor([2, 8, 6, 9, 4, 0]), size=(5, 3))是第二层采样得到的bipartite图。
可以看出来,第二层采样是建立在第一层采样的基础上的,第一层采样bipartite中所有的节点[0, 1, 2] (在原图中对应[2, 4, 1])作为第二层采样的出发点。所以edge_index[1]有[0, 0, 1, 1, 2, 2],这是将0, 1, 2作为target节点采样两个邻居的结果。
就不再赘述更多样例了,大家可以自己试一试,玩一玩。
欢迎多交流,转发请注明出处。
实战代码
https://github.com/DGraphXinye/2022_finvcup_baseline
这是我为我们公司比赛准备的baseline代码,里面包含了基本的图算法以及相应的mini-batch版本。
