【读文献】可解读的深度学习抗体结构预测
Antibody structure prediction using interpretable deep learning
模型简称:DeepAB
Supplemental information
发表日期:December 09, 2021
作者:Jeffery等
机构:The Johns Hopkins University,
摘要
DeepAb是一个深度学习方法,通过氨基酸序列可以直接预测抗体F_v结构。在结构多样、治疗相关的benchmark数据集上测试,DeepAb的表现良好。然后剖析了DeepAb各方面在预测结构的时候做出的贡献如何,并发现网络更关注重要的氨基酸对等可解释的内容。
介绍
抗体-抗原的相互作用的合理设计都是基于准确的抗体结构上。但是通过实验得到抗体结构量少速度慢。抗体结构由两条重链和两条轻链组装成一个大的Y形复合体,结晶区Fc在同型抗体中高度保守,而可变区Fv通过形成互补决定区(CDR)的六个高度可变的环来与抗原结合。因此,对Fv区域的结构建模将对理解抗体结合机制和理性设计至关重要。
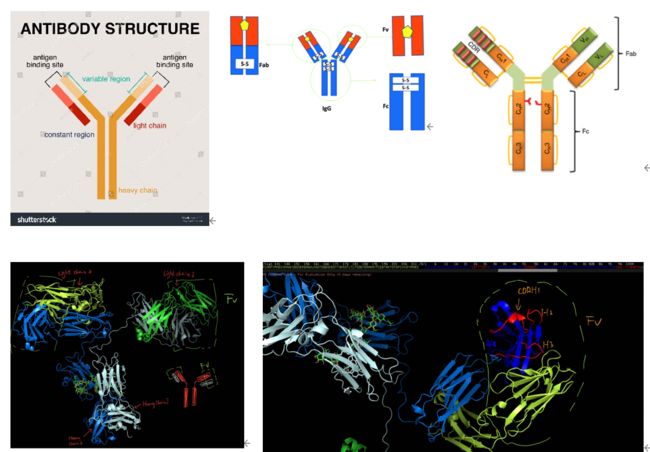
然而,抗体重链的第三个CDR环(CDR H3)的长度和构象多样性导致基于嫁接的方法难以预测得到高质量的模型,CDR H3环的建模一直是抗体Fv结构预测中的长期挑战。
本文在现有蛋白质结构预测的基础上进一步实现抗体Fv结构预测。主要通过预测残基距离和方向,并且利用Rosetta来生成结构。使用深度学习预测的结构会比嫁接的更准确。(Pre-trained model和本文code都有提供)
结果
方法概览:
包含两部分(Figure 1)
第一部分 输入重、轻链的序列,一个深度残差卷积网络(包含一些可解释的组件),通过两个残基对之间的相对距离和方向来表示预测Fv的结构。
第二部分 一个快速的基于Rosetta的结构生成协议,利用第一部分预测出来的信息作为输入。
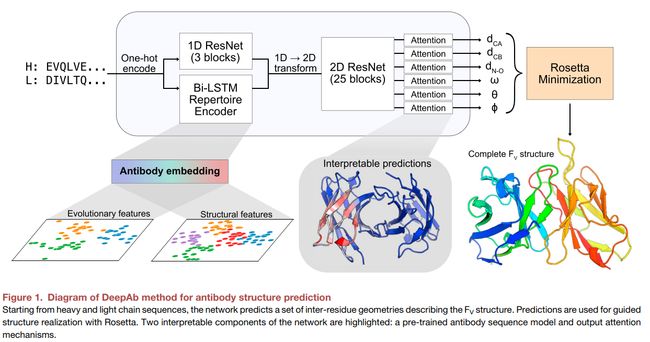
从序列中预测残基间的几何关系:
由于Fv的晶体结构数量对于监督学习来说太少了,所以尝试使用冗余的抗体序列。利用无监督表征学习,将抗体序列中的一般pattern embedding到一个潜在的表征中,这些pattern在已知结构的小子集中并不明显。
选择RNN而不是Transformer是为了方便在有限的数据集上训练。虽然RNN的固定的hidden state会使得表征学习遇到瓶颈,但是在UniRep方法中,证明了RNN在用于训练预测下一个氨基酸的任务上,能学到很丰富的表征信息。
所以搭建了一个RNN encoder-decoder模型,encoder是一个biLSTM,decoder是LSTM。简单来说,encoder能将一个输入序列中的残基一个接着一个变成固定大小的hidden state。然后hidden state变成一个总的vector,传递给decoder,让decoder一次重构一个原序列的残基。
利用交叉熵损失函数在118,386对重、轻链(OAS database)上做训练,通过将每一个残基的hidden state拼接在一起得到抗体序列的embedding(轻链和重链是一起?还是分开的,下文说了)
我们将Fv的结构表示成一组残基间距离和角度,这个距离包括碳alpha、beta和NO之间的距离,角度包括一组二面角(ω: Ca—Cb—Cb—Ca, θ: N—Ca—Cb—Cb) and (φ: Ca—Cb—Cb。(题外话:一般任意两个氨基酸之间的信息可以表示为6元组,五个二面角信息加上一个距离信息)
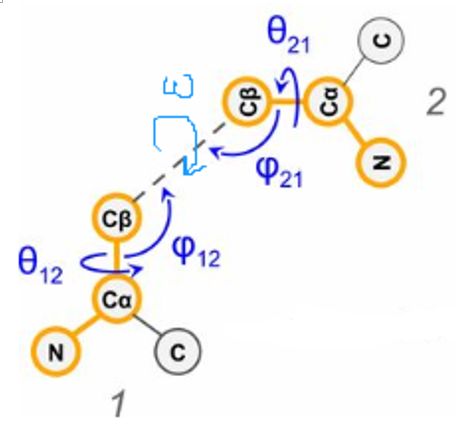
每个几何输出都会被分为36个bins,其中一个是用于描述Calpha距离大于18A的,每个bin的范围是0.5A。二面角和平面角分别按照10°和5°划分bins。
作者们之前也做过类似的CDR H3结构预测,但是有两个新内容,新增了从预训练模型中提出embedding,第二个是可解释的attention层。
网络将重链和轻链拼在一起作为输入,然后做了one-hot encoding,分别送入两个平行的分支:1D的ResNet和预训练模型。两个输出结合转换成pairwise data(下文提及),进入2D resnet之后,网络将结果分成六个输出分支,对应了每个几何信息。每个输出的分支包含了一个criss-cross attention模块,这个模块允许每一个残基对能够获得其他所有残基对的信息
损失函数使用的是focal loss。
模型在不冗余1692个Fv结构(来自于SAbDab)进行训练,预训练模型作为特征提取器,在训练过程中是不会进行更新的。
结构实现:
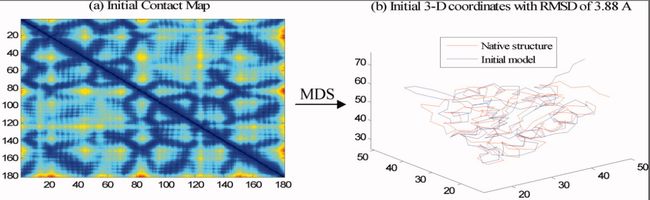
利用网络预测结果使用约束最小化来生成3D结构,不像过往的我们从phi-psi扭角中进行采样,因为框架结构区的高度保守性使得我们可以通过MDS(多维尺度法,是一组对象之间的距离或者不相似度的可视化呈现,比如说有了两两碳Alpha的距离,就可以用来计算他们的相对坐标)来创建初始模型,省了很多时间。在本文,除了利用Calpha的距离,还用了三个角度来生成了一个主链原子之间的距离(因为这四个几何信息都是bin)。
下图左边是残基距离,右边是初始模型。
网络预测出来的六个几何信息通过模型logits取负得到离散的能量电位,再通过三次样条函数将离散电位变成连续的constraints。从MDS开始,这些限制就在Rosetta里被用作引导拟牛顿法来找到最优解。
第一步,限制结合rosetta中的能量函数来优化结构得到一个比较粗放的Fv结构,这个结构的侧链都是一个单独的原子。
第二步,有限制的全原子松弛会被用来得到侧链,然后去除冲突部分。
松弛之后,结构还会根据限制和rosetta的全原子能量函数进行减少。
最优的过程中会重复产出50个结构(由于高收敛性,其实5个结构就够了),并将能量最低的结构作为最后的输出。5个结构在一个cpu上花费大概10分钟,相比嫁接(几秒几分钟一个序列)来说还是慢的,但是比直接采样(几小时一个序列)快很多。
Benchmarking Methods for Fv结构预测:
使用了两个独立的测试集(47条的RosettaAntibody数据集,45条临床阶段的治疗性抗体数据集)来验证效果。这两者可以组成一个有结构多样性、治疗性的数据集。
深度学习比拼接方法好:
尽管我们的方法和现有蛋白质结构预测有相似性,但是因为现在已有的预测抗体的方法不能成功预测那些具有挑战性的CDR loop,所以我们的方法主要与特定的抗体方法进行比较。
与RosettaAntibody-G、RepertoireBuilder 和 ABodyBuilder进行比较,他们都是嫁接方法,也就是他们的Fv结构是通过过往已知结构那些具有序列相似性的片段组装起来的。
通过CDR loop主链以及两个链的Framework region的重原子的RMSD来评估方法。
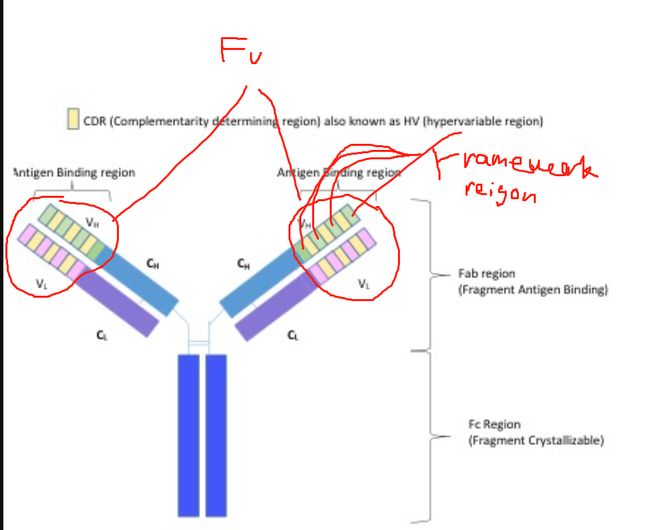
黄色是高变区,绿色是重链的framework region,粉色是轻链的FR
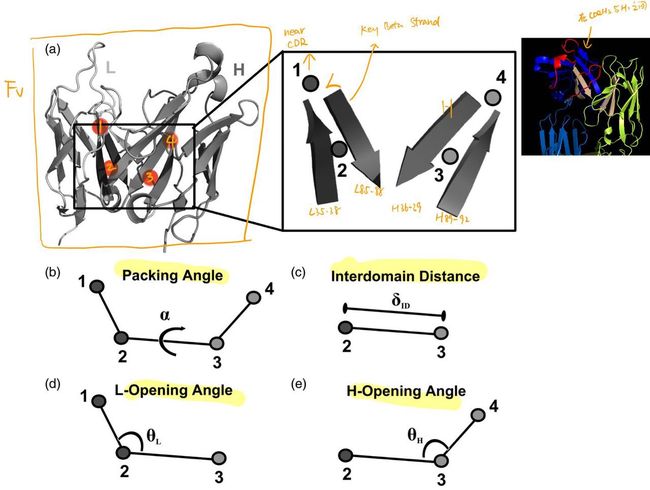
我们还测量了orientational coordinate distance(OCD),一个重轻链的方向准确度标准。他通过LHOC(light-heavy orientational coordinates,是一个四指标方向坐标框架)来计算重、轻链结合的结构与真实结构之间的差距。这四个指标分别是下图的bcde,而四个points是Fv区域的Key Beta Strand中选取的
结构A相对于B的OCD如下所示,其中θi,dB表示最适合LHOC指标i数据库(当前数据)分布的高斯分布的标准差。
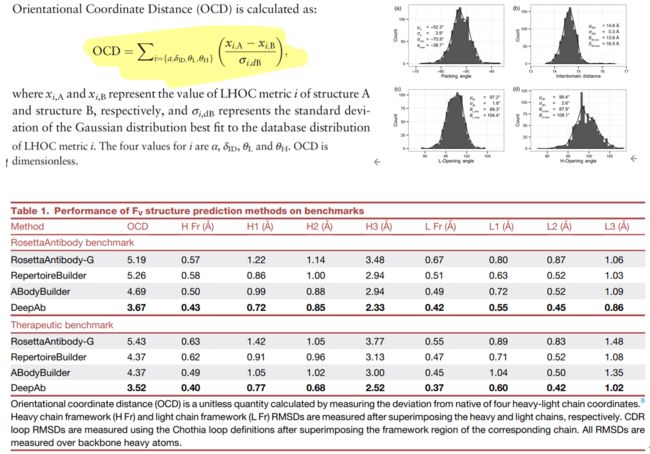
Table1 是在benchmark上的表现。OCD没有单位,用于计算与原结构的4个坐标的偏差。H/L Fr是重/轻链framework。所有的RMSD都是在骨架重原子上计算的。
DeepAb的每一个表现都是最好的,并且在重链framework上相对第二好的有14%-18%的进步,轻链有16%-17%提高。CDR loop结构也有提高。
对比CDR H3建模的准确率
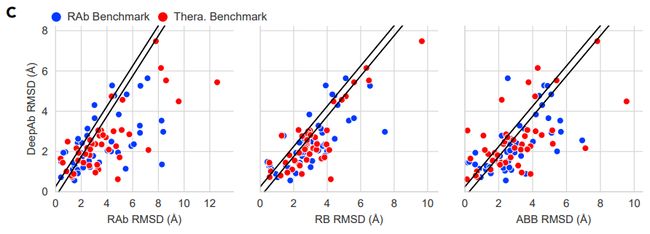
图A是几个方法在CDR H3上的表现,两个数据集上平均RMSD分别为2.33A(±1.32),2.52(±1.50),比第二好进步16%。图B为了看H3长度会有什么样的影响,发现越长每个模型的效果都越差,但是DeepAb在每一个长度上效果都是最好的。
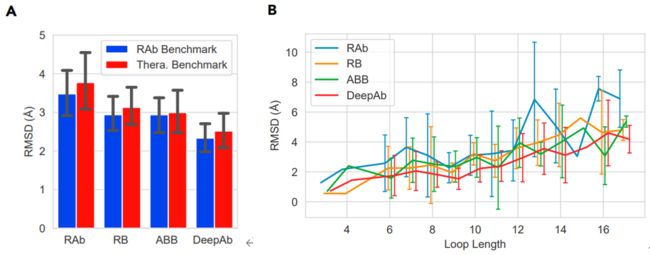
图C对比了DeepAb在不同数据集上的表现,黑线是指两者预测出来的rmsd差距在0.25A以内,黑线以内的就当相等。总体来说在92个数据中有42个H3是在2A以内,但另外三个方法分别只有26,23,26.
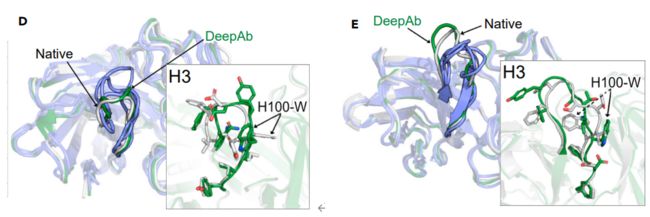
准确预测有挑战、治疗性相关的数据
选取了两个有难度的samples:3PP3和3I9G。在H3的预测上,3PP3长度为12aa,嫁接方法只能达到3.3-4.1RMSD,DeepAb可以达到2.1。3I9G上,DeepAb1.8,嫁接2.9-3.9。两个samples上DeepAb都能抓住主要的loop形状并且能找到不少准确的侧链位置。
问题:两个Sample上,DeepAb都遇到H100位置上的残基方向预测错误。
(模型)结构对H3 loop建模准确率的影响
相比之前预测H3结构的工作,本次主要有两个主要的新增内容:预训练LSTM和输出处的交叉attention。本节通过训练了两个额外的模型来看这两个新增结构的影响。(消融实验)
第一个模型是baseline,不包含LSTM或者交叉attention。第二个模型包含LSTM。然后在92个数据集上和我们的full 模型做比较:
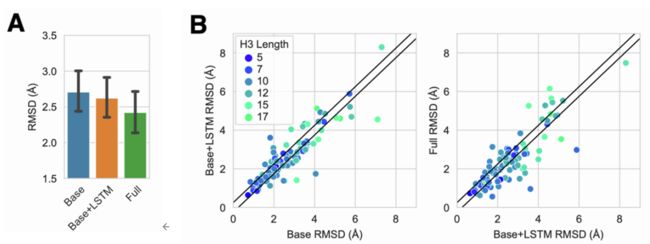
说明两个模块都是有用的
模型预测的解释性
输出的attention能抓住模型的关注点
有六个criss-cross attention(为了解决non-local-把map中的任意一个点都考虑其他所有点-计算复杂才有的)。criss-cross attention通过两个1*1卷积得到QK,然后Q中每一个元素都和K的行列相乘,然后得到只有所在位置行列的A(这个过程称为Affinity)。然后后有了A和V之后,十字架上的内容对应相乘再相加,结果再加上最开始原始位置的数值得到一个新的值。
下图Loop1表示在第一次做attention时,蓝色点能收集到所在的行列信息,在第一次attention之后,整个矩阵中的每个点都包含了所在行所在列的信息。然后第二次再用行列做attention的时候,就会看到整个map上的信息(原来的行列又去看了行列)
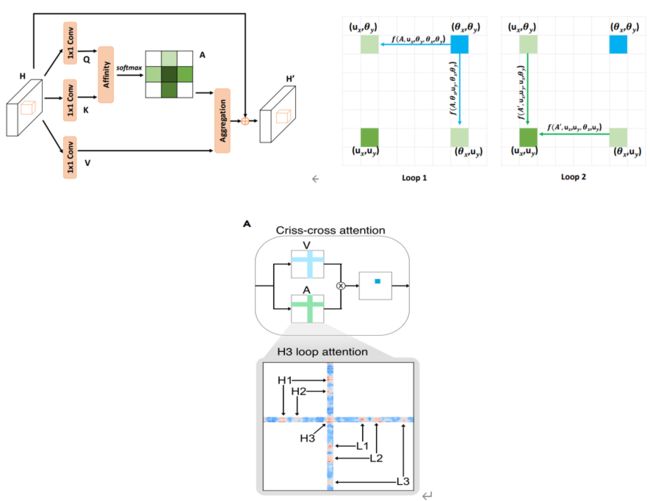
我们用4H0H作为可视化的例子,因为预测出来的H3在1.2A。
我们收集了dCalpha的attention矩阵,然后看在预测H3的时候(H3的碳原子?)更关注哪些残基。意料之中,在预测loop的时候更关注周围的残基。
对于CDR1-2loops,网络关注相邻的残基,很少关注对面的链。但对于CDR3,轻链重链都会关注到,表明loop的结构和链整体的方向有关。
仔细看了看H3,在预测H3的时候最关注的C端扭结,这也是先前研究表明C端扭结会影响H3构象的多样性。再看一眼除了H3的5个最关注的残基,一个是苯丙氨酸,三个是酪氨酸,他们庞大的侧链也对H3构象起到很大影响。第五个是丝氨酸(L91)与H-100丝氨酸形成了氢键。
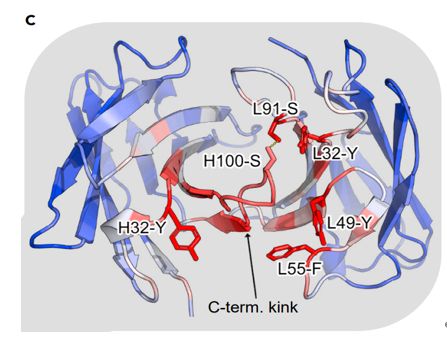
模型学习到进化和结构表征
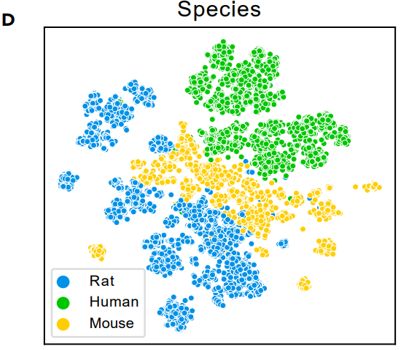
通过t-SNE(T-分布邻域嵌入算法)将LSTM encoder编码的序列embedding转换成二维,发现序列很自然的根据种类聚集。由于结构数据集主要由人类和鼠类抗体组成,无监督的特征很可能提供了进化的背景,否则是无法获得的。
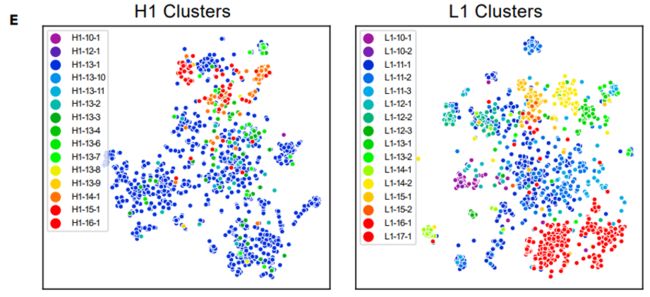
对于那五个non-H3的CDR,他们通常呈现为几个典型的构象之一,从H1和L1的embedding也能清楚的看出不同构象的聚类情况。
总结
本文使用预训练模型和attention机制有效地预测了抗体中Fv的结构,并且借助attention的帮助发现网络在预测结构时会更关注Fv的什么区域,以及预测CDR H3时网络会更关注哪些残基。
在这篇文章在review的时候,AlphaFold2发表了,可能存在的优势是它是单序列,本文是复合体。未来会研究与抗原结合之后的结构预测。
实验过程
通讯作者:Jeffrey J.Gray([email protected])论文中预测的结构数据在Zenodo(10.5281/zenodo.5525257),代码在github
独立测试集
RosettaAntibody:49条,CDR H3的长度在7-17,每个结构都大于2.5埃的解析度;
56条临床阶段的治疗性抗体,有着已知晶体结构。移除了五个丢失CDR loop的PDB(3B2U, 3C08, 3HMW, 3S34, and 4EDW),组成一个 therapeutic benchmark set。
两个数据集有两个交集(3EO9 and 3GIZ),从第二个数据集中移除了。
但是在对比的方法中,有一些方法不能成功预测所有的target,最后选取了47个RosettaAntibody,45个therapeutic benchmark。使用Chothia CDR loop definition来测量RMSD
预训练模型
训练数据集
Fv区成对的重轻链序列来自OAS数据库,OAS中所有的序列都是ANARCI进行解析并标注的。所以我们只提取了ANARCI标注的Fv序列。最后从五个研究中获得了118,386条数据。
模型和训练细节
为了学习到免疫球蛋白的representation,使用了encoder-decoder模型,由两个LSTM组成。decoder是为了将序列复原。
encoder中使用的是biLSTM,隐藏层大小为64,输入是重链序列和轻链序列拼在一起,并且加了三个tokens,表示重链的头尾和轻链的尾。
拼接好的序列做one-hot encoding之后得到一个(L+3)23的矩阵。
最终的输出向量是将向前和向后的LSTM结果进行了拼接,然后放入一个线性变化,从128维降到64维,并经过一个tanh激活函数。
deocder用了两层LSTM,隐藏层大小为64,用来预测原序列。
模型使用交叉熵损失函数和Adam优化器(lr=0.01,在损失趋于平稳时降低学习率)
Teacher forcing = 0.5
在NVIDIA K80 GPU 大约4小时 5个epoch,batch size128(占满内存)
预测几何信息
训练数据集
从ASbDab和PDB中收集Fv结构,解析度小于4A的去除,并且有99%同一性的序列移除。之所以选择高相似度的是因为抗体序列的高保守性特点,以及数据库中有很多一致的序列有过高的代表性。还有就是希望模型能够辨别极其相似序列的不同结构。
benchmark中出现的或者结构有99%相似的都去除了。
最后得到1692个Fv结构用于训练。
模型和训练细节
输入是重轻链拼起来,做了one-hot之后分别输入1D-Resnet和biLSTMencoder。
1D-Resnet中我们加了一个分隔符channel来标注重链的尾,得到一个L21的矩阵(20个aa一个delimiter)。1D-Resnet是由一个1d卷积开始,将输入的特征扩展到L64,然后跟上三个1D-Resnet的模块(两个kernel size为17的1D卷积),并保持矩阵大小。
第二个分支是预训练中的encoder,中间有三个delimiters,所以是(L+3)23,然后在每一个氨基酸encode之后就将前后两个向量拼接得到128维度,总共得到L128维度,将两者的结果拼接在一起得到L160。然后通过两两残基对,对应相拼得到LL320,传给2DResNet。
2DResNet开头是一个2D卷积,将维度减少到LL64,再跟上25个blocks(里面是55的2d卷积)。25个block每5个block一个循环,分别设置扩张值为1,2,4,8,16。
2DResNet之后,网络拆封分成了6个分支,每个分支包含一个2D卷积把维度从LL64降到LL37,然后跟上一个循环criss-cross attention(RCCA)模块。
RCCA:使用了两个共享权重的criss-cross attention操作,使得任意一个残基对都能在整个空间维度中收集信息。Q和K被投影到LL1大小(一个head)。(一个残基对可以用137向量表示)
所有卷积层后面都跟上了一个ReLU激活,总共包含大约6.4million可训练参数。
随机划分9/1的训练和验证集训练了5个模型,5个模型做Logit average ensemble。使用focal loss训练和Adam优化器(lr=0.01并且当loss变平稳就降低lr)
每个模型在一个NVIDIA K80 GPU上需要大约60h训练60epochs。
结构实现
多维尺度法
预测的结果我们是通过取bin的中点值获得的,根据这些距离和角度的真实值,利用骨架原子初始了原子之间的距离矩阵,对于碳beta距离大于18A的,我们通过floyd-warshall最短距离算法估计了原子之间的距离。然后利用MDS初始了一组3d坐标。但初始的结构可能会包含冲突和不理想的几何值,需要进一步精细。
能量最小化
利用rosetta进行能量最小化精细。对于每一对残基,将模型的logit取负得到能量电位。再通过rosetta把离散的电位变成连续的电位。去除Calpha距离大于18A的,bin概率小于10%的,对于d_N_O,去除大于5A,概率小于30%的。
注意力矩阵计算
可以见前文的criss-cross attention部分。
注:
Pairwise(外积-对应相乘,外加-对应相加,外拼-对应相拼-维度扩大,外差-对应相减)
原本是L160,每个氨基酸是160维度,那么两两拼在一起比如第i个氨基酸和第j个氨基酸,外拼之后就是320,但这个代表了i拼j,所以总共有LL个320。