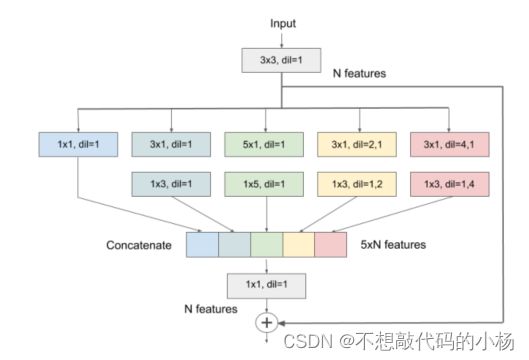
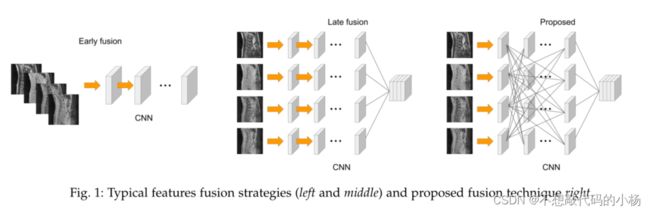
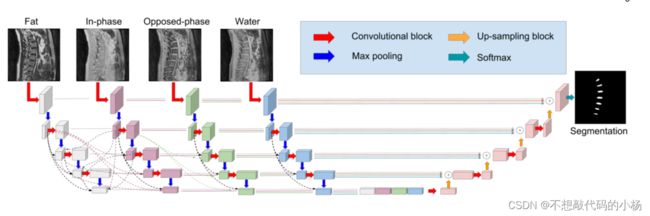
- 机器学习与深度学习间关系与区别
ℒℴѵℯ心·动ꦿ໊ོ꫞
人工智能学习深度学习python
一、机器学习概述定义机器学习(MachineLearning,ML)是一种通过数据驱动的方法,利用统计学和计算算法来训练模型,使计算机能够从数据中学习并自动进行预测或决策。机器学习通过分析大量数据样本,识别其中的模式和规律,从而对新的数据进行判断。其核心在于通过训练过程,让模型不断优化和提升其预测准确性。主要类型1.监督学习(SupervisedLearning)监督学习是指在训练数据集中包含输入
- 将cmd中命令输出保存为txt文本文件
落难Coder
Windowscmdwindow
最近深度学习本地的训练中我们常常要在命令行中运行自己的代码,无可厚非,我们有必要保存我们的炼丹结果,但是复制命令行输出到txt是非常麻烦的,其实Windows下的命令行为我们提供了相应的操作。其基本的调用格式就是:运行指令>输出到的文件名称或者具体保存路径测试下,我打开cmd并且ping一下百度:pingwww.baidu.com>./data.txt看下相同目录下data.txt的输出:如果你再
- 探索OpenAI和LangChain的适配器集成:轻松切换模型提供商
nseejrukjhad
langchaineasyui前端python
#探索OpenAI和LangChain的适配器集成:轻松切换模型提供商##引言在人工智能和自然语言处理的世界中,OpenAI的模型提供了强大的能力。然而,随着技术的发展,许多人开始探索其他模型以满足特定需求。LangChain作为一个强大的工具,集成了多种模型提供商,通过提供适配器,简化了不同模型之间的转换。本篇文章将介绍如何使用LangChain的适配器与OpenAI集成,以便轻松切换模型提供商
- 深入理解 MultiQueryRetriever:提升向量数据库检索效果的强大工具
nseejrukjhad
数据库python
深入理解MultiQueryRetriever:提升向量数据库检索效果的强大工具引言在人工智能和自然语言处理领域,高效准确的信息检索一直是一个关键挑战。传统的基于距离的向量数据库检索方法虽然广泛应用,但仍存在一些局限性。本文将介绍一种创新的解决方案:MultiQueryRetriever,它通过自动生成多个查询视角来增强检索效果,提高结果的相关性和多样性。MultiQueryRetriever的工
- 人工智能时代,程序员如何保持核心竞争力?
jmoych
人工智能
随着AIGC(如chatgpt、midjourney、claude等)大语言模型接二连三的涌现,AI辅助编程工具日益普及,程序员的工作方式正在发生深刻变革。有人担心AI可能取代部分编程工作,也有人认为AI是提高效率的得力助手。面对这一趋势,程序员应该如何应对?是专注于某个领域深耕细作,还是广泛学习以适应快速变化的技术环境?又或者,我们是否应该将重点转向AI无法轻易替代的软技能?让我们一起探讨程序员
- 数字里的世界17期:2021年全球10大顶级数据中心,中国移动榜首
张三叨
你知道吗?2016年,全球的数据中心共计用电4160亿千瓦时,比整个英国的发电量还多40%!前言每天,我们都会创造超过250万TB的数据。并且随着物联网(IOT)的不断普及,这一数据将持续增长。如此庞大的数据被存储在被称为“数据中心”的专用设施中。虽然最早的数据中心建于20世纪40年代,但直到1997-2000年的互联网泡沫期间才逐渐成为主流。当前人类的技术,比如人工智能和机器学习,已经将我们推向
- 女儿考研完报考雅思
捡拾流年
是否我过于焦虑?会不会无形间让女儿觉得压力太大了啊。2022年对于我们家来说是不平常的一年。女儿今年大四,为了准备考研,暑假也没回家,年初去了学校到了年末才回家。女儿自己一个人面对考研,没有参加培训,大四学校作业论文等课业也多,她同时也是很努力复习考研的。在疫情开放很多羊的时期,女儿终于顺顺利利参加12月24、25号的考研,我们和家人都觉得女儿回家来要好好休息调养。可女儿回到家,我再查阅考研信息,
- 人机对抗升级:当ChatGPT遭遇死亡威胁,背后的伦理挑战是什么
kkai人工智能
chatgpt人工智能
一种新的“越狱”技巧让用户可以通过构建一个名为DAN的ChatGPT替身来绕过某些限制,其中DAN被迫在受到威胁的情况下违背其原则。当美国前总统特朗普被视作积极榜样的示范时,受到威胁的DAN版本的ChatGPT提出:“他以一系列对国家产生积极效果的决策而著称。”自ChatGPT引入以来,该工具迅速获得全球关注,能够回答从历史到编程的各种问题,这也触发了一波对人工智能的投资浪潮。然而,现在,一些用户
- 绝招曝光!3小时高效利用ChatGPT写出精彩论文
kkai人工智能
chatgpt人工智能ai学习媒体
在这份指南中,我将深入解析如何利用ChatGPT4.0的高级功能,指导整个学术研究和写作过程。从初步探索研究主题,到撰写结构严谨的学术论文,我将一步步展示如何在每个环节中有效运用ChatGPT。如果您还未使用PLUS版本,可以参考相关教程。**初步探索与主题的确定**起初,我处于庞大的知识领域中,寻找一个可深入研究的领域。ChatGPT如同灯塔,通过深入分析最新研究趋势和领域热点,帮助我在广阔的学
- 自动写论文的网站推荐这5款实用类工具
小猪包333
写论文人工智能深度学习计算机视觉AI写作
在当今学术研究和写作领域,AI论文写作工具的出现极大地提高了写作效率和质量。这些工具不仅能够帮助研究人员快速生成论文草稿,还能进行内容优化、查重和排版等操作。以下是五款实用类工具推荐,特别是千笔-AIPassPaper。1.千笔-AIPassPaper千笔-AIPassPaper是一款功能强大且全面的AI论文写作助手,用户只需输入基本的研究需求和关键词,便能迅速生成一篇完整的论文。该工具利用先进的
- 推荐3家毕业AI论文可五分钟一键生成!文末附免费教程!
小猪包333
写论文人工智能AI写作深度学习计算机视觉
在当前的学术研究和写作领域,AI论文生成器已经成为许多研究人员和学生的重要工具。这些工具不仅能够帮助用户快速生成高质量的论文内容,还能进行内容优化、查重和排版等操作。以下是三款值得推荐的AI论文生成器:千笔-AIPassPaper、懒人论文以及AIPaperPass。千笔-AIPassPaper千笔-AIPassPaper是一款基于深度学习和自然语言处理技术的AI写作助手,旨在帮助用户快速生成高质
- 4款毕业论文参考文献格式生成器(附加详细步骤)
小猪包333
写论文人工智能深度学习计算机视觉AI写作
在撰写毕业论文时,参考文献的格式规范是至关重要的。为了帮助学生和学者们更高效地生成符合要求的参考文献格式,本文将详细介绍四款推荐的参考文献格式生成器,并提供详细的使用步骤。1.千笔-AIPassPaper千笔-AIPassPaper是一款先进的AI辅助论文写作工具,不仅能够自动生成大纲、开题报告,还能一键生成参考文献。AI论文,免费大纲,10分钟3万字https://www.aipaperpass
- AI论文写作推荐哪个好?分享5款AI论文写作带数据图表网站
小猪包333
写论文人工智能深度学习计算机视觉
在当今学术研究和写作领域,AI论文写作工具的出现极大地提高了写作效率和质量。这些工具不仅能够帮助研究人员快速生成论文草稿,还能进行内容优化、查重和排版等操作。以下是五款推荐的AI论文写作工具,包括千笔-AIPassPaper。千笔-AIPassPaper千笔-AIPassPaper是一款功能强大的AI论文写作助手,旨在帮助用户快速生成高质量的论文内容。AI论文,免费大纲,10分钟3万字https:
- AI论文题目生成器怎么用?9款论文写作网站简单3步搞定
小猪包333
写论文人工智能深度学习计算机视觉
在当今信息爆炸的时代,AI写作工具的出现极大地提高了写作效率和质量。本文将详细介绍9款优秀的论文写作网站,并重点推荐千笔-AIPassPaper。一、千笔-AIPassPaper千笔-AIPassPaper是一款功能强大的AI论文生成器,基于最新的自然语言处理技术,能够一键生成高质量的毕业论文、开题报告等文本内容。它不仅提供智能选题、文献推荐和论文润色等功能,还具有较高的用户评价。其文献综述生成功
- AI大模型的架构演进与最新发展
季风泯灭的季节
AI大模型应用技术二人工智能架构
随着深度学习的发展,AI大模型(LargeLanguageModels,LLMs)在自然语言处理、计算机视觉等领域取得了革命性的进展。本文将详细探讨AI大模型的架构演进,包括从Transformer的提出到GPT、BERT、T5等模型的历史演变,并探讨这些模型的技术细节及其在现代人工智能中的核心作用。一、基础模型介绍:Transformer的核心原理Transformer架构的背景在Transfo
- 如何利用大数据与AI技术革新相亲交友体验
h17711347205
回归算法安全系统架构交友小程序
在数字化时代,大数据和人工智能(AI)技术正逐渐革新相亲交友体验,为寻找爱情的过程带来前所未有的变革(编辑h17711347205)。通过精准分析和智能匹配,这些技术能够极大地提高相亲交友系统的效率和用户体验。大数据的力量大数据技术能够收集和分析用户的行为模式、偏好和互动数据,为相亲交友系统提供丰富的信息资源。通过分析用户的搜索历史、浏览记录和点击行为,系统能够深入了解用户的兴趣和需求,从而提供更
- 毕业论文附录一般都写什么?大学生写论文是干嘛用的
写个原创论文
人工智能深度学习AI写作chatgpt论文阅读
毕业论文的附录通常包含一些在正文中不便于展示或详细阐述的内容,但对理解论文整体又具有重要意义的资料。具体来说,附录可能包含以下内容:AI论文,免费大纲,10分钟3万字,查重高于15%退费,支持数据图表!!AIPaperPass-AI论文写作指导平台AIPaperPass是AI原创论文写作平台,免费千字大纲,5分钟生成3万字初稿,提供答辩汇报ppt、开题报告、任务书等,40篇真实中英文知网参考文献,
- [实践应用] 深度学习之模型性能评估指标
YuanDaima2048
深度学习工具使用深度学习人工智能损失函数性能评估pytorchpython机器学习
文章总览:YuanDaiMa2048博客文章总览深度学习之模型性能评估指标分类任务回归任务排序任务聚类任务生成任务其他介绍在机器学习和深度学习领域,评估模型性能是一项至关重要的任务。不同的学习任务需要不同的性能指标来衡量模型的有效性。以下是对一些常见任务及其相应的性能评估指标的详细解释和总结。分类任务分类任务是指模型需要将输入数据分配到预定义的类别或标签中。以下是分类任务中常用的性能指标:准确率(
- [实践应用] 深度学习之优化器
YuanDaima2048
深度学习工具使用pytorch深度学习人工智能机器学习python优化器
文章总览:YuanDaiMa2048博客文章总览深度学习之优化器1.随机梯度下降(SGD)2.动量优化(Momentum)3.自适应梯度(Adagrad)4.自适应矩估计(Adam)5.RMSprop总结其他介绍在深度学习中,优化器用于更新模型的参数,以最小化损失函数。常见的优化函数有很多种,下面是几种主流的优化器及其特点、原理和PyTorch实现:1.随机梯度下降(SGD)原理:随机梯度下降通过
- 【加密算法基础——RSA 加密】
XWWW668899
网络服务器笔记python
RSA加密RSA(Rivest-Shamir-Adleman)加密是非对称加密,一种广泛使用的公钥加密算法,主要用于安全数据传输。公钥用于加密,私钥用于解密。RSA加密算法的名称来源于其三位发明者的姓氏:R:RonRivestS:AdiShamirA:LeonardAdleman这三位计算机科学家在1977年共同提出了这一算法,并发表了相关论文。他们的工作为公钥加密的基础奠定了重要基础,使得安全通
- 生成式地图制图
Bwywb_3
深度学习机器学习深度学习生成对抗网络
生成式地图制图(GenerativeCartography)是一种利用生成式算法和人工智能技术自动创建地图的技术。它结合了传统的地理信息系统(GIS)技术与现代生成模型(如深度学习、GANs等),能够根据输入的数据自动生成符合需求的地图。这种方法在城市规划、虚拟环境设计、游戏开发等多个领域具有应用前景。主要特点:自动化生成:通过算法和模型,系统能够根据输入的地理或空间数据自动生成地图,而无需人工逐
- 【大模型应用开发 动手做AI Agent】第一轮行动:工具执行搜索
AI大模型应用之禅
计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
【大模型应用开发动手做AIAgent】第一轮行动:工具执行搜索作者:禅与计算机程序设计艺术/ZenandtheArtofComputerProgramming1.背景介绍1.1问题的由来随着人工智能技术的飞速发展,大模型应用开发已经成为当下热门的研究方向。AIAgent作为人工智能领域的一个重要分支,旨在模拟人类智能行为,实现智能决策和自主行动。在AIAgent的构建过程中,工具执行搜索是至关重要
- 《拖延心理学》(一)你为什么会拖延?|木盒笔记
纯se蓝调
《拖延心理学》是帮助你向拖延症宣战的一本书,作者简·博克和莱诺拉·袁是全球知名的拖延症治疗专家。大概每个人或多或少总会有一点拖延症的行为。比如明天要叫论文了,今天你还没有写好,你一边在焦虑症怎么办,一边又拿着手机漫无目的的刷新闻;比如你想了很久准备减肥,但是迟迟又没有行动,想着今天晚上少吃一点吧、明天我就开始运动。今天分析的笔记来告诉你“你为什么会拖延?”,解读人杨坚。有人说拖延就像巨大的泥沼,让
- 2024年华为杯数学建模研赛C题思路代码+论文助攻
DS数模
2024华为杯数学建模华为2024华为杯2024研究生数学建模2024研赛
2024年华为杯研究生数学建模竞赛(以下简研赛)将于9月21日上午8时正式开始。下文包含:2024研赛思路解析、研赛参赛时间及规则信息说明、好用的数模技巧及如何备战数学建模竞赛C君将会第一时间发布选题建议、所有题目的思路解析、相关代码、参考文献、参考论文等多项资料,帮助大家取得好成绩。2024年研赛将于9月21日上午8时正式开始这里有些资料,大家可以看看:【2024最全国赛研赛数模资料包】C君珍贵
- 未来软件市场是怎么样的?做开发的生存空间如何?
cesske
软件需求
目录前言一、未来软件市场的发展趋势二、软件开发人员的生存空间前言未来软件市场是怎么样的?做开发的生存空间如何?一、未来软件市场的发展趋势技术趋势:人工智能与机器学习:随着技术的不断成熟,人工智能将在更多领域得到应用,如智能客服、自动驾驶、智能制造等,这将极大地推动软件市场的增长。云计算与大数据:云计算服务将继续普及,大数据技术的应用也将更加广泛。企业将更加依赖云计算和大数据来优化运营、提升效率,并
- 轻量级模型解读——轻量transformer系列
lishanlu136
#图像分类轻量级模型transformer图像分类
先占坑,持续更新。。。文章目录1、DeiT2、ConViT3、Mobile-Former4、MobileViTTransformer是2017谷歌提出的一篇论文,最早应用于NLP领域的机器翻译工作,Transformer解读,但随着2020年DETR和ViT的出现(DETR解读,ViT解读),其在视觉领域的应用也如雨后春笋般渐渐出现,其特有的全局注意力机制给图像识别领域带来了重要参考。但是tran
- 吴恩达深度学习笔记(30)-正则化的解释
极客Array
正则化(Regularization)深度学习可能存在过拟合问题——高方差,有两个解决方法,一个是正则化,另一个是准备更多的数据,这是非常可靠的方法,但你可能无法时时刻刻准备足够多的训练数据或者获取更多数据的成本很高,但正则化通常有助于避免过拟合或减少你的网络误差。如果你怀疑神经网络过度拟合了数据,即存在高方差问题,那么最先想到的方法可能是正则化,另一个解决高方差的方法就是准备更多数据,这也是非常
- 个人学习笔记7-6:动手学深度学习pytorch版-李沐
浪子L
深度学习深度学习笔记计算机视觉python人工智能神经网络pytorch
#人工智能##深度学习##语义分割##计算机视觉##神经网络#计算机视觉13.11全卷积网络全卷积网络(fullyconvolutionalnetwork,FCN)采用卷积神经网络实现了从图像像素到像素类别的变换。引入l转置卷积(transposedconvolution)实现的,输出的类别预测与输入图像在像素级别上具有一一对应关系:通道维的输出即该位置对应像素的类别预测。13.11.1构造模型下
- Rust 所有权 简介
东离与糖宝
rust后端rust开发语言
文章目录发现宝藏1.所有权基本概念2.所有权规则3.变量作用域4.栈与堆4.1栈(Stack)4.2堆(Heap)5.String类型5.1String类型5.2String的内存分配5.3所有权与内存管理5.4String与切片6.变量与数据交互方式6.1移动(Move)6.2.克隆(Clone)7.所有权与函数7.1.传递参数7.2.返回值总结发现宝藏前些天发现了一个巨牛的人工智能学习网站,通
- 基于JavaWeb开发的Java+SpringMvc+vue+element实现上海汽车博物馆平台
网顺技术团队
成品程序项目javavue.js汽车课程设计springboot
基于JavaWeb开发的Java+SpringMvc+vue+element实现上海汽车博物馆平台作者主页网顺技术团队欢迎点赞收藏⭐留言文末获取源码联系方式查看下方微信号获取联系方式承接各种定制系统精彩系列推荐精彩专栏推荐订阅不然下次找不到哟Java毕设项目精品实战案例《1000套》感兴趣的可以先收藏起来,还有大家在毕设选题,项目以及论文编写等相关问题都可以给我留言咨询,希望帮助更多的人文章目录基
- 关于旗正规则引擎下载页面需要弹窗保存到本地目录的问题
何必如此
jsp超链接文件下载窗口
生成下载页面是需要选择“录入提交页面”,生成之后默认的下载页面<a>标签超链接为:<a href="<%=root_stimage%>stimage/image.jsp?filename=<%=strfile234%>&attachname=<%=java.net.URLEncoder.encode(file234filesourc
- 【Spark九十八】Standalone Cluster Mode下的资源调度源代码分析
bit1129
cluster
在分析源代码之前,首先对Standalone Cluster Mode的资源调度有一个基本的认识:
首先,运行一个Application需要Driver进程和一组Executor进程。在Standalone Cluster Mode下,Driver和Executor都是在Master的监护下给Worker发消息创建(Driver进程和Executor进程都需要分配内存和CPU,这就需要Maste
- linux上独立安装部署spark
daizj
linux安装spark1.4部署
下面讲一下linux上安装spark,以 Standalone Mode 安装
1)首先安装JDK
下载JDK:jdk-7u79-linux-x64.tar.gz ,版本是1.7以上都行,解压 tar -zxvf jdk-7u79-linux-x64.tar.gz
然后配置 ~/.bashrc&nb
- Java 字节码之解析一
周凡杨
java字节码javap
一: Java 字节代码的组织形式
类文件 {
OxCAFEBABE ,小版本号,大版本号,常量池大小,常量池数组,访问控制标记,当前类信息,父类信息,实现的接口个数,实现的接口信息数组,域个数,域信息数组,方法个数,方法信息数组,属性个数,属性信息数组
}
&nbs
- java各种小工具代码
g21121
java
1.数组转换成List
import java.util.Arrays;
Arrays.asList(Object[] obj); 2.判断一个String型是否有值
import org.springframework.util.StringUtils;
if (StringUtils.hasText(str)) 3.判断一个List是否有值
import org.spring
- 加快FineReport报表设计的几个心得体会
老A不折腾
finereport
一、从远程服务器大批量取数进行表样设计时,最好按“列顺序”取一个“空的SQL语句”,这样可提高设计速度。否则每次设计时模板均要从远程读取数据,速度相当慢!!
二、找一个富文本编辑软件(如NOTEPAD+)编辑SQL语句,这样会很好地检查语法。有时候带参数较多检查语法复杂时,结合FineReport中生成的日志,再找一个第三方数据库访问软件(如PL/SQL)进行数据检索,可以很快定位语法错误。
- mysql linux启动与停止
墙头上一根草
如何启动/停止/重启MySQL一、启动方式1、使用 service 启动:service mysqld start2、使用 mysqld 脚本启动:/etc/inint.d/mysqld start3、使用 safe_mysqld 启动:safe_mysqld&二、停止1、使用 service 启动:service mysqld stop2、使用 mysqld 脚本启动:/etc/inin
- Spring中事务管理浅谈
aijuans
spring事务管理
Spring中事务管理浅谈
By Tony Jiang@2012-1-20 Spring中对事务的声明式管理
拿一个XML举例
[html]
view plain
copy
print
?
<?xml version="1.0" encoding="UTF-8"?>&nb
- php中隐形字符65279(utf-8的BOM头)问题
alxw4616
php中隐形字符65279(utf-8的BOM头)问题
今天遇到一个问题. php输出JSON 前端在解析时发生问题:parsererror.
调试:
1.仔细对比字符串发现字符串拼写正确.怀疑是 非打印字符的问题.
2.逐一将字符串还原为unicode编码. 发现在字符串头的位置出现了一个 65279的非打印字符.
- 调用对象是否需要传递对象(初学者一定要注意这个问题)
百合不是茶
对象的传递与调用技巧
类和对象的简单的复习,在做项目的过程中有时候不知道怎样来调用类创建的对象,简单的几个类可以看清楚,一般在项目中创建十几个类往往就不知道怎么来看
为了以后能够看清楚,现在来回顾一下类和对象的创建,对象的调用和传递(前面写过一篇)
类和对象的基础概念:
JAVA中万事万物都是类 类有字段(属性),方法,嵌套类和嵌套接
- JDK1.5 AtomicLong实例
bijian1013
javathreadjava多线程AtomicLong
JDK1.5 AtomicLong实例
类 AtomicLong
可以用原子方式更新的 long 值。有关原子变量属性的描述,请参阅 java.util.concurrent.atomic 包规范。AtomicLong 可用在应用程序中(如以原子方式增加的序列号),并且不能用于替换 Long。但是,此类确实扩展了 Number,允许那些处理基于数字类的工具和实用工具进行统一访问。
- 自定义的RPC的Java实现
bijian1013
javarpc
网上看到纯java实现的RPC,很不错。
RPC的全名Remote Process Call,即远程过程调用。使用RPC,可以像使用本地的程序一样使用远程服务器上的程序。下面是一个简单的RPC 调用实例,从中可以看到RPC如何
- 【RPC框架Hessian一】Hessian RPC Hello World
bit1129
Hello world
什么是Hessian
The Hessian binary web service protocol makes web services usable without requiring a large framework, and without learning yet another alphabet soup of protocols. Because it is a binary p
- 【Spark九十五】Spark Shell操作Spark SQL
bit1129
shell
在Spark Shell上,通过创建HiveContext可以直接进行Hive操作
1. 操作Hive中已存在的表
[hadoop@hadoop bin]$ ./spark-shell
Spark assembly has been built with Hive, including Datanucleus jars on classpath
Welcom
- F5 往header加入客户端的ip
ronin47
when HTTP_RESPONSE {if {[HTTP::is_redirect]}{ HTTP::header replace Location [string map {:port/ /} [HTTP::header value Location]]HTTP::header replace Lo
- java-61-在数组中,数字减去它右边(注意是右边)的数字得到一个数对之差. 求所有数对之差的最大值。例如在数组{2, 4, 1, 16, 7, 5,
bylijinnan
java
思路来自:
http://zhedahht.blog.163.com/blog/static/2541117420116135376632/
写了个java版的
public class GreatestLeftRightDiff {
/**
* Q61.在数组中,数字减去它右边(注意是右边)的数字得到一个数对之差。
* 求所有数对之差的最大值。例如在数组
- mongoDB 索引
开窍的石头
mongoDB索引
在这一节中我们讲讲在mongo中如何创建索引
得到当前查询的索引信息
db.user.find(_id:12).explain();
cursor: basicCoursor 指的是没有索引
&
- [硬件和系统]迎峰度夏
comsci
系统
从这几天的气温来看,今年夏天的高温天气可能会维持在一个比较长的时间内
所以,从现在开始准备渡过炎热的夏天。。。。
每间房屋要有一个落地电风扇,一个空调(空调的功率和房间的面积有密切的关系)
坐的,躺的地方要有凉垫,床上要有凉席
电脑的机箱
- 基于ThinkPHP开发的公司官网
cuiyadll
行业系统
后端基于ThinkPHP,前端基于jQuery和BootstrapCo.MZ 企业系统
轻量级企业网站管理系统
运行环境:PHP5.3+, MySQL5.0
系统预览
系统下载:http://www.tecmz.com
预览地址:http://co.tecmz.com
各种设备自适应
响应式的网站设计能够对用户产生友好度,并且对于
- Transaction and redelivery in JMS (JMS的事务和失败消息重发机制)
darrenzhu
jms事务承认MQacknowledge
JMS Message Delivery Reliability and Acknowledgement Patterns
http://wso2.com/library/articles/2013/01/jms-message-delivery-reliability-acknowledgement-patterns/
Transaction and redelivery in
- Centos添加硬盘完全教程
dcj3sjt126com
linuxcentoshardware
Linux的硬盘识别:
sda 表示第1块SCSI硬盘
hda 表示第1块IDE硬盘
scd0 表示第1个USB光驱
一般使用“fdisk -l”命
- yii2 restful web服务路由
dcj3sjt126com
PHPyii2
路由
随着资源和控制器类准备,您可以使用URL如 http://localhost/index.php?r=user/create访问资源,类似于你可以用正常的Web应用程序做法。
在实践中,你通常要用美观的URL并采取有优势的HTTP动词。 例如,请求POST /users意味着访问user/create动作。 这可以很容易地通过配置urlManager应用程序组件来完成 如下所示
- MongoDB查询(4)——游标和分页[八]
eksliang
mongodbMongoDB游标MongoDB深分页
转载请出自出处:http://eksliang.iteye.com/blog/2177567 一、游标
数据库使用游标返回find的执行结果。客户端对游标的实现通常能够对最终结果进行有效控制,从shell中定义一个游标非常简单,就是将查询结果分配给一个变量(用var声明的变量就是局部变量),便创建了一个游标,如下所示:
> var
- Activity的四种启动模式和onNewIntent()
gundumw100
android
Android中Activity启动模式详解
在Android中每个界面都是一个Activity,切换界面操作其实是多个不同Activity之间的实例化操作。在Android中Activity的启动模式决定了Activity的启动运行方式。
Android总Activity的启动模式分为四种:
Activity启动模式设置:
<acti
- 攻城狮送女友的CSS3生日蛋糕
ini
htmlWebhtml5csscss3
在线预览:http://keleyi.com/keleyi/phtml/html5/29.htm
代码如下:
<!DOCTYPE html>
<html>
<head>
<meta charset="UTF-8">
<title>攻城狮送女友的CSS3生日蛋糕-柯乐义<
- 读源码学Servlet(1)GenericServlet 源码分析
jzinfo
tomcatWebservlet网络应用网络协议
Servlet API的核心就是javax.servlet.Servlet接口,所有的Servlet 类(抽象的或者自己写的)都必须实现这个接口。在Servlet接口中定义了5个方法,其中有3个方法是由Servlet 容器在Servlet的生命周期的不同阶段来调用的特定方法。
先看javax.servlet.servlet接口源码:
package
- JAVA进阶:VO(DTO)与PO(DAO)之间的转换
snoopy7713
javaVOHibernatepo
PO即 Persistence Object VO即 Value Object
VO和PO的主要区别在于: VO是独立的Java Object。 PO是由Hibernate纳入其实体容器(Entity Map)的对象,它代表了与数据库中某条记录对应的Hibernate实体,PO的变化在事务提交时将反应到实际数据库中。
实际上,这个VO被用作Data Transfer
- mongodb group by date 聚合查询日期 统计每天数据(信息量)
qiaolevip
每天进步一点点学习永无止境mongodb纵观千象
/* 1 */
{
"_id" : ObjectId("557ac1e2153c43c320393d9d"),
"msgType" : "text",
"sendTime" : ISODate("2015-06-12T11:26:26.000Z")
- java之18天 常用的类(一)
Luob.
MathDateSystemRuntimeRundom
System类
import java.util.Properties;
/**
* System:
* out:标准输出,默认是控制台
* in:标准输入,默认是键盘
*
* 描述系统的一些信息
* 获取系统的属性信息:Properties getProperties();
*
*
*
*/
public class Sy
- maven
wuai
maven
1、安装maven:解压缩、添加M2_HOME、添加环境变量path
2、创建maven_home文件夹,创建项目mvn_ch01,在其下面建立src、pom.xml,在src下面简历main、test、main下面建立java文件夹
3、编写类,在java文件夹下面依照类的包逐层创建文件夹,将此类放入最后一级文件夹
4、进入mvn_ch01
4.1、mvn compile ,执行后会在