论文翻译-LSTM Multi-modal UNet for Brain Tumor Segmentation
用于脑肿瘤分割的LSTM多模态神经网络
论文地址链接:https://pan.baidu.com/s/1HuPtnRvVuTgiwv8FZh0juw
提取码:xvsu
github地址 https://github.com/HowieMa/lstm_multi_modal_UNet
摘要——卷积神经网络等深度学习模型已广泛应用于三维生物医学图像分割。然而,它们中的大多数既没有考虑不同模态之间的相关性,也没有充分利用深度信息。为了更好地利用多模态和深度信息,我们提出了一种在多模态磁共振图像中分割脑肿瘤的结构,称为LSTM多模态UNet.。在BRATS-2015上的实验结果表明,我们的方法优于最先进的生物医学分割方法。
关键词-多模态图像;图像分割;UNetLSTM;深度学习
一、引言
近年来,磁共振成像(MRI)已被广泛用于研究人脑的结构和功能。由于它,在脑部疾病分析、诊断、治疗和识别研究领域取得了重大成就,特别是在脑瘤方面。为了更好地诊断和治疗肿瘤,基于脑磁共振成像的肿瘤分割至关重要。
磁共振成像有四种图像序列:T2加权液体衰减反转恢复(FLAIR)、T1加权(T1)、T1加权对比增强(T1c)、T2加权(T2)。这四种图像通常分别被称为磁共振成像的一种形式,它们在肿瘤分割中起着不同的作用。例如,使用Flair可以更好地进行整个肿瘤的分割,并且在T2下可以更好地分割肿瘤核心。典型的处理方法之一是早期融合,它结合了低层特征上的模态。这种早期融合是基于不同模态之间的关系简单而实际上很复杂的假设[1]。为了更好地了解多模态信息,其他研究人员提出了后期融合策略,即每个模态在独立的CNN之后与其他模态在深层融合。这种后期融合策略在脑分割上优于早期融合[2]。此外,一些研究表明,不同模态之间的复杂性不能简单地用单层来建模[1]。在文献[3]中,提出了一种CNN,它不仅在单个模态内的层对之间,而且在不同模态的层之间包含密集的连接,可以解释多模态数据建模中的非线性。
近年来,随着卷积神经网络的发展,深度学习在脑肿瘤分割领域取得了显著的成就。一些方法使用完全卷积网络(FCN) [4]来分割3D生物医学图像。基于FCN的U-Net [5]在这个问题上也有不错的表现。
然而,三维图像的分割往往会导致训练速度慢的问题,因为三维图像的尺寸很大。基于面片的方法[6]将图像的一小部分放入网络中,并预测每个中心像素的结果。一些方法[7]将2D分割用于3D生物医学数据,这意味着将2D分割应用于3D图像的每个切片,并将每个结果连接起来以获得3D分割。然而,这些方法忽略了整体结构信息或连续切片之间的顺序信息。考虑到充分学习整个信息,我们利用卷积LSTM [8]来更好地利用连续切片之间的关系。
在这篇论文中,我们提出了一种新的方法,LSTM多模态UNet,,如图1所示。简而言之,我们利用2DUnet 分割磁共振图像的切片,LSTM [8]学习连续切片的顺序信息。密集连接不仅用于获得每个模态的全部特征,还用于获得它们之间的复杂关系。
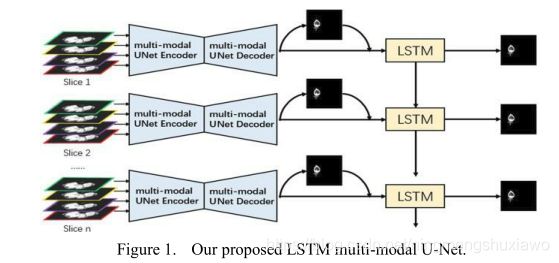
二、方法
我们的方法,LSTM多模态UNet,由两部分组成,1)多模态UNet 2)卷积LSTM [8]。多模式UNet包括超高密度编码器和解码器,以充分利用多模态数据。卷积LSTM进一步利用了连续切片之间的顺序信息。
A Multi-modal UNet
提议的Multi-modal UNet 遵循IVD网络[3]的结构。
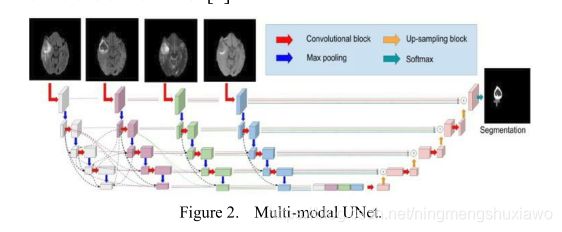 1)编解码:我们采用UNet [5]的架构作为我们的基本编解码结构。这个众所周知的模型由收缩路径和扩张路径组成。前者包含多个卷积,用于下采样以生成高级特征,后者包含多个反卷积层,用于上采样特征以生成像素级分割。此外,在压缩过程中,使用跳跃连接来传输信息,该跳跃连接将裁剪后的要素地图与收缩路径连接起来。然而,UNet 忽略了深度信息,因为它是一种2D分割方法,没有很好地利用多模态数据,因为它是一种预融合策略。
1)编解码:我们采用UNet [5]的架构作为我们的基本编解码结构。这个众所周知的模型由收缩路径和扩张路径组成。前者包含多个卷积,用于下采样以生成高级特征,后者包含多个反卷积层,用于上采样特征以生成像素级分割。此外,在压缩过程中,使用跳跃连接来传输信息,该跳跃连接将裁剪后的要素地图与收缩路径连接起来。然而,UNet 忽略了深度信息,因为它是一种2D分割方法,没有很好地利用多模态数据,因为它是一种预融合策略。
2)多编码路径:为了利用UNet的结构并实现密集的连接模式,我们使用具有多个UNet编码路径的架构,每个路径分别处理一个模态图像。多编码路径的目标是更好地说明多模态数据之间的复杂关系避免了早期融合,这限制了网络的学习能力。
3)超密集连接:为了在各个层次上充分利用多模态信息,我们在多UNet编码路径网络中采用了超密集连接方法[9]。
让x1表示lth层的输出,Hl是一个映射函数,它对应于我们提出的卷积块。在以前的中枢神经系统中,第1层的输出通常是从前一层xl-1的输出获得的
![]()
在密集连接的网络中,所有特征输出以前馈方式连接
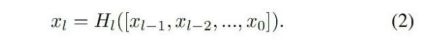
[…]表示串联操作。
在当前的工作中,来自不同编码路径中的前一层的输出也被连接以形成到后续层的输入。在多模态环境中,这种结构产生了比早期或晚期融合策略更强大的特征表示,因为网络具有学习不同模态之间更复杂关系的能力。为简单起见,假设我们只有两种模态。

此外,最近的工作已经发现,在CNN中打乱顺序和交错特征映射(shuffling and
interleaving feature maps )可以提高其性能,因为它作为一个强正则化器工作。因此,我们以不同的顺序为每个分支和图层连接要素地图,此时第1层的输出变为
![]()
F p是一个改变输入要素图顺序的函数。
 B.切片序列学习
B.切片序列学习
我们提出了一个端到端切片序列学习模型来利用序列相关性。图像深度被视为一系列切片。我们使用卷积LSTM [8]来模拟切片之间的关系。
1)卷积LSTM:与传统的LSTM方法不同,卷积LSTM用卷积算子*代替了矩阵乘法,为长期序列保留了空间信息。整个网络定义如下:
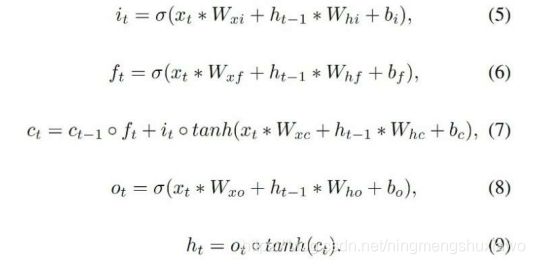 2)后期LSTM:由于我们的多模态UNet由两部分组成:编码路径和解码路径,切片依赖。我们在解码路径后添加了名为LSTM多模式UNet的convLSTM块。由于多模态UNet已经完全利用了不同的模态信息,convLSTM网络应该只捕获序列相关性。此外,我们在编码路径后添加监督,以避免梯度消失。
2)后期LSTM:由于我们的多模态UNet由两部分组成:编码路径和解码路径,切片依赖。我们在解码路径后添加了名为LSTM多模式UNet的convLSTM块。由于多模态UNet已经完全利用了不同的模态信息,convLSTM网络应该只捕获序列相关性。此外,我们在编码路径后添加监督,以避免梯度消失。
C.与现有方法的关系
2017年,Kuan-Lun还提出了一个网络,该网络具有convL- STM和跨模态卷积[10],以利用序列和多模态信息。然而,Kuan-Lun网络的编码路径遵循后融合策略,这种策略需要更多的参数,因为交叉模态卷积的通道尺寸更大。显然,我们的网络不同于IVD网络的架构[3]。由于IVD网络是一种2D分割方法,因此不能使用序列关系。IVD网采用了初始模块,大大增加了模型参数和内存消耗。
三.实验
我们对三维生物医学图像分割问题进行了实验,并将我们的LSTM多模态超声图像与传统方法进行了比较,以证明其实用性。
A.数据集
BRATS-2015 [10]是最具挑战性的3D分割问题之一,因为肿瘤的大小和形状各不相同。训练数据集包括220名患有高等级胶质瘤的受试者(HGG)和54名患有低等级胶质瘤的受试者(LGG)。每张核磁共振图像的大小为155 x 240x240,我们从所有274名受试者中随机选择224名受试者进行培训,50名受试者进行测试。数据集中的所有大脑都具有相同的方向,并且配准了四种模态(Flair、T1、T1c、T2)。所有大脑包含五个标签:0代表非肿瘤,1代表坏死,2代表水肿,3代表非增强肿瘤,4代表增强肿瘤。
B.评估指标
评估标准是每个标签的交集(IoU)。IoU是一种常用的分割性能度量,它计算相交面积与并集面积的比率。
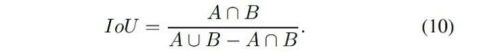
同时,我们将肿瘤结构分为三个区域,如BRATS-2015在线判断系统所示。
完整评分:表示整个肿瘤区域和测量标签1、2、3、4。
核心评分:仅考虑肿瘤核心区域,评估标签1、3、4。
提高分数:它将提高核心结构考虑在内,并评估标签4。(仅限HGG)
评价标准有三种:骰子相似系数、正预测值和灵敏度。
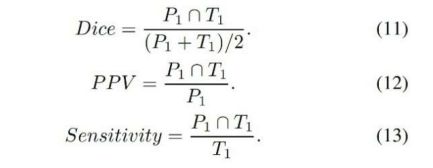
C 实施细节
1)基线:我们用Pytorch实现我们的实验[11]。几个模型被用来展示我们网络的优势。我们选择早期融合策略的UNet作为基线。所有的模态被合并以建立一个具有4个通道的图像。详细信息见表1。
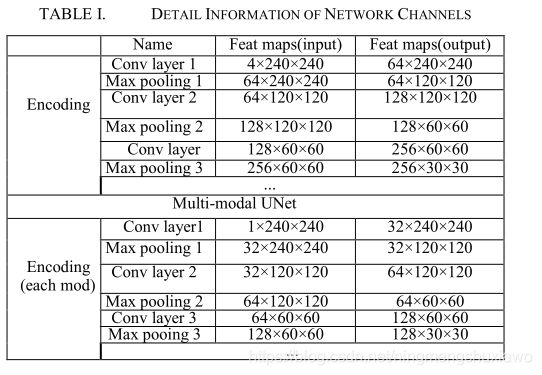 对于UNet中的每个卷积块,我们首先使用内核大小为3×3的卷积层来生成特征图,然后是一个批处理归一化层和一个逐元素校正的线性非线性(ReLU)。由于肿瘤和非肿瘤组织的分布是不均衡的,所以批量归一化层对于训练我们的网络是至关重要的。然后,使用大小为2且步长为2的最大池图层对特征图进行下采样。如表1所示,所有输入和输出通道的数量都基于标准UNet。
对于UNet中的每个卷积块,我们首先使用内核大小为3×3的卷积层来生成特征图,然后是一个批处理归一化层和一个逐元素校正的线性非线性(ReLU)。由于肿瘤和非肿瘤组织的分布是不均衡的,所以批量归一化层对于训练我们的网络是至关重要的。然后,使用大小为2且步长为2的最大池图层对特征图进行下采样。如表1所示,所有输入和输出通道的数量都基于标准UNet。
我们还实现了2D多模态UNet作为对比。2D多模态UNet有多个UNet编码路径和一个解码路径。为了证明更多的参数不是我们模型性能提高的原因,我们使用标准UNet的一半通道数。
2)我们的网络:对于我们提议的架构,我们使用的2D多模式UNet的频道数量与表1所示相同。至于序列学习,我们最初将序列长度设置为4。
3)训练:由于BRATS2015数据集的标签分布极不平衡。因此模态很容易收敛到局部最小值。我们使用中值频率平衡策略[10]来解决这个问题。交叉熵损失函数中每个Wc类的权重定义为:
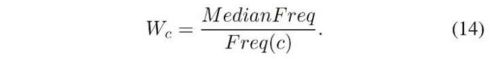
其中Freq©是所有训练像素中c类的频率。此外,我们在训练中只采集带有肿瘤组织的切片。
在我们的实验中,我们使用亚当[12]优化器来训练所有的架构,β1=0.9,β2=0.99。初始学习率设为10-4。对于所有基于2D的模型,在每个小批量中使用32幅图像,对于所有基于序列的网络,使用16个3D体积。所有核磁共振成像图像在0和1之间被归一化,并且没有使用数据增加来提高所有网络的性能。
四.结果
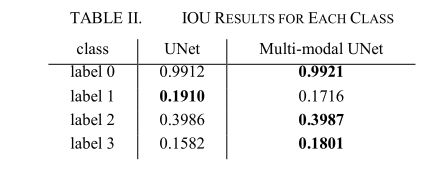
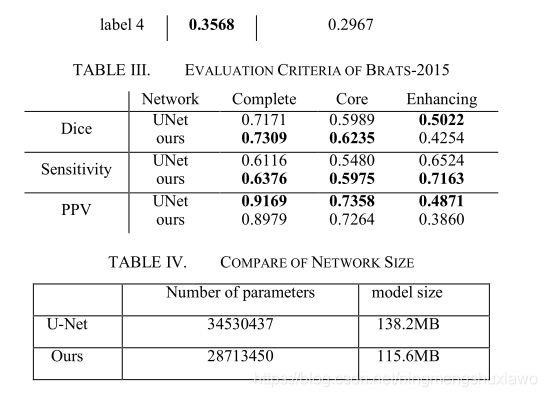
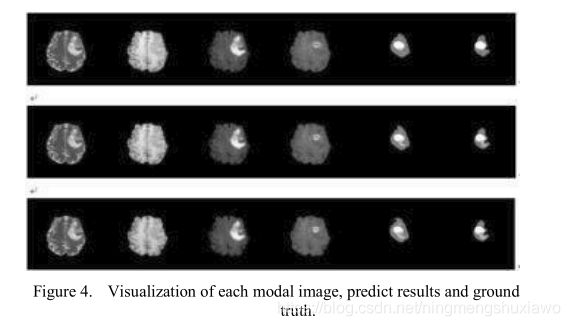
表2和表3报告了我们实验的定量结果。我们还显示了网络参数的数量和模型大小。我们发现,与模型参数较少的标准U-Net相比,我们的LSTM多模式UNet带来了性能的提升。该结果表明,每个模态和序列之间的相关性可以极大地帮助提高基于我们的网络的分割结果的准确性。此外,我们在图4中提供了对我们提出的模型的定性评估。
五、结论
在这篇文章中,我们提出了一个网络,它结合了多模态神经网络和LSTM,以充分利用模态之间的关系和序列之间的相关性,称为LSTM多模态神经网络。我们基于BRATS-2015训练了我们的模型,它显示出比传统的联合国教育、科学及文化组织更好的表现。未来我们会尝试借鉴UNet++来降低模型的复杂度,提高单幅图像的分割速度。此外,我们还考虑了通过弱监督学习来改进算法的方法。
生词短语
biomedical image segmentation.生物医学图像分割。
leverage手段,影响力;[力]杠杆作用;杠杆效率
BraTS 2015 :一个数据集
Simply put简而言之
reserves the spatial information保留空间信息
Apparently显然
quantitative results定量结果
qualitative evaluation定性评价