Lasso model selection: Cross-Validation / AIC / BIC
利用Akaike信息准则(AIC)、Bayes信息准则(BIC)和交叉验证来选择Lasso估计量的正则化参数α的最优值。
LassoLarsIC获得的结果基于AIC/BIC标准。
基于信息准则的模型选择是非常快速的,但它依赖于对自由度的适当估计,是针对大样本(渐近结果)导出的,并且假设模型是正确的,即数据实际上是由该模型生成的。当问题条件恶劣时(特征多于样本),它们也往往会中断。
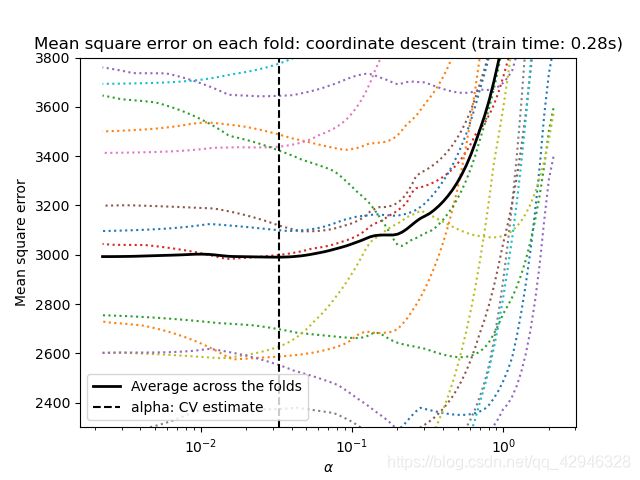
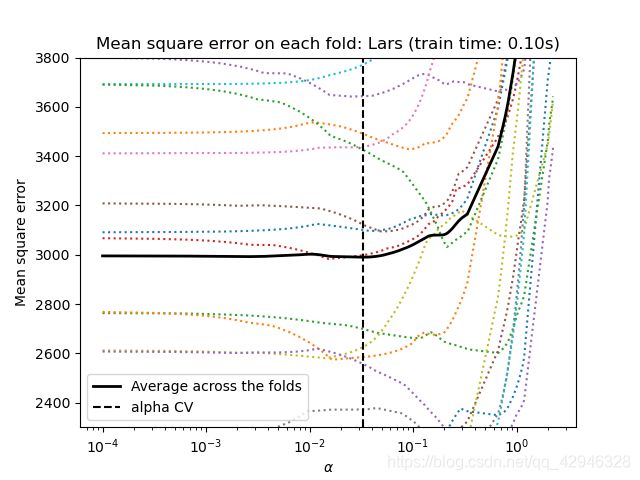
对于交叉验证,我们使用两种算法计算套索路径:坐标下降(由LassoCV类实现)和Lars(最小角度回归)(由LassoLarsCV类实现)。两种算法得出的结果大致相同。它们在执行速度和数值误差来源方面有所不同。
Lars只为路径中的每个扭结计算路径解。因此,当只有很少的扭结时,它是非常有效的,如果特征或样本很少的话就是这种情况。无需设置任何参数,meta也能计算完整路径。相反,坐标下降计算预先指定网格上的路径点(这里我们使用默认值)。因此,如果网格点的数目小于路径中的扭结数目,则效率更高。如果特征的数量真的很大,并且有足够的样本来选择大量的特征,那么这样的策略可能会很有趣。在数值误差方面,对于高度相关的变量,Lars会累积更多的误差,而坐标下降算法只对网格上的路径进行采样。
注意α的最佳值是如何随每次折叠而变化的。这说明了当试图评估由交叉验证选择参数的方法的性能时,嵌套交叉验证是必要的:对于看不见的数据,这种参数选择可能不是最佳的。
import time
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import LassoCV, LassoLarsCV, LassoLarsIC
from sklearn import datasets
# 这是为了避免在np.log10时被0除
EPSILON = 1e-4
X, y = datasets.load_diabetes(return_X_y=True) # 导入数据
rng = np.random.RandomState(42)
X = np.c_[X, rng.randn(X.shape[0], 14)] # 增加一些表现不好的特征
# 按Lars所做的标准化数据,以便进行比较
X /= np.sqrt(np.sum(X ** 2, axis=0))
# #############################################################################
# LassoLarsIC: 基于BIC/AIC准则的最小角度回归
model_bic = LassoLarsIC(criterion='bic')
t1 = time.time()
model_bic.fit(X, y)
t_bic = time.time() - t1 # 拟合模型运行时间
alpha_bic_ = model_bic.alpha_ # 打印alpha
model_aic = LassoLarsIC(criterion='aic')
model_aic.fit(X, y)
alpha_aic_ = model_aic.alpha_
# 画出所有alphas中信息标准(“aic”、“bic”)的值
def plot_ic_criterion(model, name, color):
criterion_ = model.criterion_ # 模型使用的标准
plt.semilogx(model.alphas_ + EPSILON, criterion_, '--', color=color,
linewidth=3, label='%s criterion' % name)
plt.axvline(model.alpha_ + EPSILON, color=color, linewidth=3,
label='alpha: %s estimate' % name) # 标出最优alpha
plt.xlabel(r'$\alpha$')
plt.ylabel('criterion')
plt.figure()
plot_ic_criterion(model_aic, 'AIC', 'b')
plot_ic_criterion(model_bic, 'BIC', 'r')
plt.legend()
plt.title('Information-criterion for model selection (training time %.3fs)'
% t_bic)
# #############################################################################
# LassoCV: 坐标下降法
# Compute paths
print("Computing regularization path using the coordinate descent lasso...")
t1 = time.time()
model = LassoCV(cv=20).fit(X, y)
t_lasso_cv = time.time() - t1
# Display results
plt.figure()
ymin, ymax = 2300, 3800
plt.semilogx(model.alphas_ + EPSILON, model.mse_path_, ':')
plt.plot(model.alphas_ + EPSILON, model.mse_path_.mean(axis=-1), 'k',
label='Average across the folds', linewidth=2)
plt.axvline(model.alpha_ + EPSILON, linestyle='--', color='k',
label='alpha: CV estimate')
plt.legend()
plt.xlabel(r'$\alpha$')
plt.ylabel('Mean square error')
plt.title('Mean square error on each fold: coordinate descent '
'(train time: %.2fs)' % t_lasso_cv)
plt.axis('tight')
plt.ylim(ymin, ymax)
# #############################################################################
# LassoLarsCV: 最小角度回归
# Compute paths
print("Computing regularization path using the Lars lasso...")
t1 = time.time()
model = LassoLarsCV(cv=20).fit(X, y)
t_lasso_lars_cv = time.time() - t1
# Display results
plt.figure()
plt.semilogx(model.cv_alphas_ + EPSILON, model.mse_path_, ':')
plt.semilogx(model.cv_alphas_ + EPSILON, model.mse_path_.mean(axis=-1), 'k',
label='Average across the folds', linewidth=2)
plt.axvline(model.alpha_, linestyle='--', color='k',
label='alpha CV')
plt.legend()
plt.xlabel(r'$\alpha$')
plt.ylabel('Mean square error')
plt.title('Mean square error on each fold: Lars (train time: %.2fs)'
% t_lasso_lars_cv)
plt.axis('tight')
plt.ylim(ymin, ymax)
plt.show()