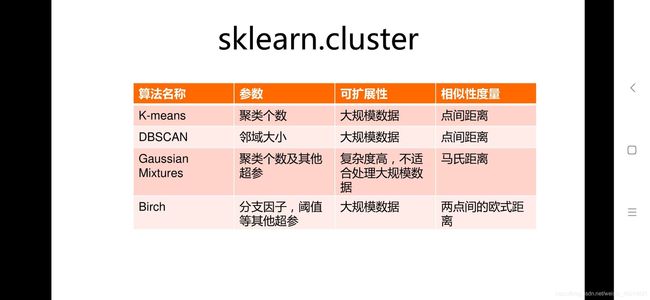
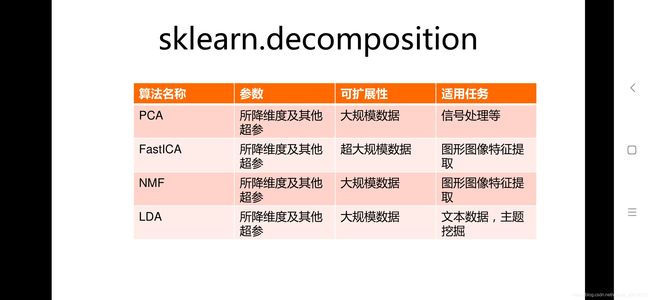
(1-3)sklearn库的模型----无监督学习
from sklearn.cluster import KMeans
KMeans(n_clusters=8, init='k-means++', n_init=10, max_iter=300, tol=0.0001, precompute_distances='auto', verbose=0, random_state=None, copy_x=True, n_jobs=1, algorithm='auto')
- n_clusters: 指定聚类中心的个数
- 最大迭代次数,设置最大迭代次数可以控制时间,否则程序运行时间会非常长;


数据请见(电脑F盘)或(腾讯微云文件“Redhur的进阶“)的{python数据—test2}
北京,2959.19,730.79,749.41,513.34,467.87,1141.82,478.42,457.64
天津,2459.77,495.47,697.33,302.87,284.19,735.97,570.84,305.08
河北,1495.63,515.90,362.37,285.32,272.95,540.58,364.91,188.63
山西,1406.33,477.77,290.15,208.57,201.50,414.72,281.84,212.10
…
import numpy as np
from sklearn.cluster import KMeans
def loadData(filePath):
fr = open(filePath,'r+',encoding='utf-8')
lines = fr.readlines()
retData = []
retCityName = []
for line in lines:
items = line.strip().split(",")
retCityName.append(items[0])
retData.append([float(items[i]) for i in range(1,len(items))])
return retData,retCityName
'''
retData大致的模样是:
[[2959.19, 730.79, 749.41, 513.34, 467.87, 1141.82, 478.42, 457.64],
[2459.77, 495.47, 697.33, 302.87, 284.19, 735.97, 570.84, 305.08]...]
K-Means聚类算法默认用的是欧氏距离
'''
if __name__=='__main__':
filepath = r'E:\快乐的程序猿\city.txt'
data,cityName=loadData(filepath)
km=KMeans(n_clusters=4) #n_cluster用于指定聚类中心的个数
label=km.fit_predict(data)
#fit_predict():计算簇中心以及为簇分配序号;
#label:聚类后各数据所属的标签,大致是[2 0 3 3 3 1 3 3 2 0 0 1 0 3 1 3 1 1 2 1 1 0 1 1 0 0 3 3 3 3 3]的样子
print(km.cluster_centers_) ##每个簇的每种消费的mean值
print("--------------------------------------------------------------------")
expenses=np.sum(km.cluster_centers_,axis=1) #每个簇的平均总消费()
print(expenses)
print("--------------------------------------------------------------------")
CityCluster=[[],[],[],[]] #将城市 按label分成设定的簇,将每个簇的城市输出
for i in range(len(cityName)):
CityCluster[label[i]].append(cityName[i])
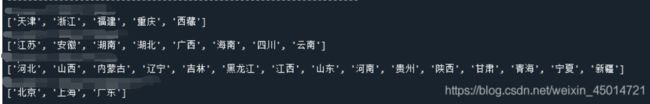
for i in range(len(CityCluster)):
print("Expenses:{}.2f" .format(expenses[i]))
print(CityCluster[i])
- cityName=[‘北京’,‘上海’,…]是一个包含31个省份名字的列表
- label=[2 0 3 3 3 1 …]含有31个数字代表相应省份所在的蔟。
- label是根据省份消费水平划分的蔟,label里面的0代表第0个蔟,一共有0、1、2、3四个蔟
现在想要把每个蔟里面的省份输出来
CityCluster=[[],[],[],[]] #四个蔟
for i in range(len(cityName)):
CityCluster[label[i]].append(cityName[i])
for i in range(len(CityCluster)):
print(CityCluster[i])
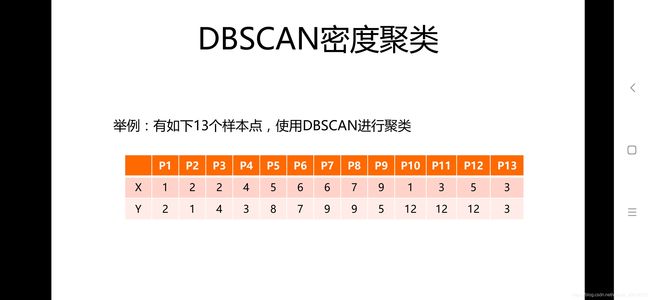
2.无监督学习中的 聚类算法之 Dbscan聚类算法–








数据请见(电脑F盘)或(腾讯微云文件“Redhur的进阶“)的{python数据—test1}
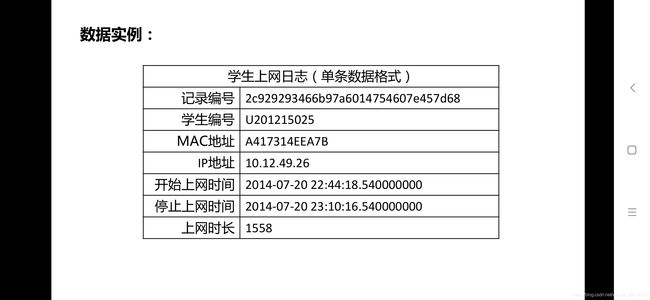
1.根据上网的时间(几点上的网)进行聚类

import numpy as np
import sklearn.cluster as skc
from sklearn import metrics
import matplotlib.pyplot as plt
mac2id = dict()
"""
在mac2id这个字典里:
键key是MAC地址
值value是字典里面对应的序号
"""
onlinetimes = []
f = open("F:/python数据/test1.txt",encoding='utf8')
for line in f:
data = line.split(',')
mac = data[2] #读取Mac地址
onlinetime = int(data[6]) #读取上网时长
starttime = int(data[4].split(' ')[1].split(':')[0]) #读取开始时间(我们只要年月日时分秒里的“时”)
mac2id[mac] = len(onlinetimes) #len(onlinetimes)就是此时对应的onlinetimes里面的元素个数,mac2id的内容见下面
onlinetimes.append((starttime,onlinetime))#onlinetimes里面的内容见下面
#onlinetimes里面的内容是[(22, 1558), (12, 40261), (22, 1721), (23, 351), (16, 23564),,,]
#mac2id这个字典里面是{'A417314EEA7B': 0, 'F0DEF1C78366': 1, '88539523E88D': 2,,,,'3CDFBD175878': 287, '002427FE3712': 288}
real_X = np.array(onlinetimes).reshape((-1,2)) #参数-1可以自动确定行数
X = real_X[:,0:1] # 截取第一列(也就是“上网时间”),我们是要根据上网的时间进行蔟类
'''
real_X为:
[[ 22 1558]
[ 12 40261]
[ 22 1721]
[ 23 351]
[ 16 23564]
[ 23 1162]
[ 22 3540]
...
]
X为:
[[22]
[12]
[22]
[23]
[16]
[23]
[22]
...
]
'''
db = skc.DBSCAN(eps=0.01,min_samples=20,metric = 'euclidean').fit(X) #调用Dbscan的的方法进行训练
# eps:两个样本被看作邻居节点的最大距离
# min_sample:簇的样本数
# metrics:距离计算方式(默认欧几里得距离)
labels = db.labels_
print("Labels")
print(labels)
ratio = len(labels[labels[:]==-1]) / len(labels) # 判定噪声数据(label被打上-1)数据所占的比例
print("Noise ratio:{:.2f}".format(ratio)) #输出噪声数据所占的比例
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0) #减去噪声数据-1占的位置
print("Estimate number of clusters:%d"%n_clusters_)#输出蔟的个数
print("Silhouette Coefficient: %0.3f"% metrics.silhouette_score(X, labels))#输出蔟类效果评价指标(轮廓系数)
#轮廓系数(Silhouette Coefficient)的值是介于 [-1,1] ,越趋近于1代表内聚度和分离度都相对较优
for i in range(n_clusters_):
print("Cluster",i,":")
print(list(X[labels==i].flatten()))
# flatten()方法:将numpy对象(如array、mat)折叠成一维数组返回
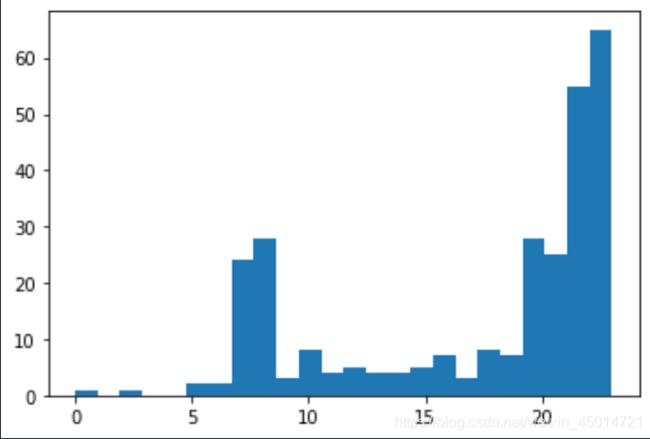
plt.hist(X,24)
plt.show()
import numpy as np
city=['太原','北京','上海','成都','西安','南京','广州','西宁']
rank=[ 2, 0, 0, 1, 1, 1, 0, 2 ] #城市对应的排名
ranks=np.array(rank)
citys=np.array(city)
print(ranks[:]==2) #[False False False True True True False False]
print('----------------------------------------------------')
num=len( ranks[ranks[:]==2] ) #ranks里面元素等于2的元素的数量(也就是排名第二的城市的数量)
print(num)
print('----------------------------------------------------')
print( citys[ranks[:]==2] ) #输出排名第二的城市
print('----------------------------------------------------')
print( set(ranks) )
print('----------------------------------------------------')
for i in range( len(set(ranks)) ):
print('排名第',i,'的城市是:')
print( citys[ranks[:]==i] )
print('----------------------------------------------------')
2.根据上网的总时长进行聚类
import numpy as np
import sklearn.cluster as skc
from sklearn import metrics
mac2id = dict()
"""
key是MAC地址
value是开始上网时间和上网时长
"""
onlinetimes = []
f = open("F:/python数据/test1.txt",encoding='utf8')
for line in f:
data = line.split(',')
mac = data[2] #读取Mac地址
onlinetime = int(data[6]) #读取上网时间
starttime = int(data[4].split(' ')[1].split(':')[0]) #读取开始时间(我们只要年月日时分秒里的“时”)
mac2id[mac] = len(onlinetimes) #len(onlinetimes)就是此时对应的onlinetimes里面的元素个数,mac2id的内容见下面
onlinetimes.append((starttime,onlinetime))#onlinetimes里面的内容见下面
#onlinetimes里面的内容是[(22, 1558), (12, 40261), (22, 1721), (23, 351), (16, 23564),,,]
#mac2id这个字典里面是{'A417314EEA7B': 0, 'F0DEF1C78366': 1, '88539523E88D': 2,,,,'3CDFBD175878': 287, '002427FE3712': 288}
#-----------------------------------------以下内容进行了改动-------------------------------------------------------------
real_X = np.array(onlinetimes).reshape((-1,2)) #参数-1可以自动确定行数
X = real_X[:,1:] # 截取第二列(也就是“上网的总时长”)我们这次根据上网的总时长进行蔟类
X=np.log(1+real_X[:,1:])#上网的总时长太大了,都是几千,我们要进行对数化处理
db = skc.DBSCAN(eps=0.14,min_samples=10).fit(X) #调用Dbscan的的方法进行训练
# eps:两个样本被看作邻居节点的最大距离
# min_sample:簇的样本数
# metrics:距离计算方式(默认欧几里得距离)
labels = db.labels_
print("Labels")
print(labels)
ratio = len(labels[labels[:]==-1]) / len(labels[:]) # 判定噪声数据(label被打上-1)数据所占的比例
print("Noise ratio:{:.2f}".format(ratio)) #输出噪声数据所占的比例
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
print("Estimate number of clusters:%d"%n_clusters_)#输出蔟的个数
print("Silhouette Coefficient: %0.3f"% metrics.silhouette_score(X, labels))#输出蔟类效果评价指标(轮廓系数)
#轮廓系数(Silhouette Coefficient)的值是介于 [-1,1] ,越趋近于1代表内聚度和分离度都相对较优
for i in range(n_clusters_):
print("Cluster",i,":")
count=len(X[labels==i])
mean=np.mean(real_X[labels==i][:,1])
std=np.std(real_X[labels==i][:,1])
print('\t number of sample:',count)
print('\t mean of sample:',format(mean,'.1f'))
print('\t std of sample:',format(std,'.1f'))
import numpy as np
n = 2 # 取对应特征值最大的n个特征向量
data = np.random.rand(10, 5) # (10行5列数据)生成10个样本,每个样本5个特征
mean = np.mean(data, axis=0) # 计算原始数据中每一列的均值,axis=0按列取均值
zeroCentred_data = data - mean # 数据中心化,使每个feature的均值为0
covMat = np.cov(zeroCentred_data, rowvar=False) # 计算协方差矩阵,rowvar=False表示数据的每一列代表一个feature
featValue, featVec = np.linalg.eig(covMat) # 计算协方差矩阵的特征值和特征向量
index = np.argsort(featValue) # 将特征值按从小到大排序,index是对应原featValue中的下标
n_index = index[-n:] # 取最大的n个特征值在原featValue中的下标
n_featVec = featVec[:, n_index] # 取最大的两维特征值对应的特征向量组成映射矩阵
low_dim_data = np.dot(zeroCentred_data, n_featVec) # 降维后的数据
2.调用sklearn实现PCA
import numpy as np
from sklearn.decomposition import PCA
data = np.random.rand(10, 5) # (生成10行5列数据)生成10个样本,每个样本5个特征
pca = PCA(n_components=2)
low_dim_data = pca.fit_transform(data) # 每个样本降为2维
3.PCA的参数
sklearn.decomposition.PCA(n_components=None, copy=True, whiten=False)
- n_components: PCA算法中所要保留的主成分个数,也即保留下来的特征个数,如果 n_components = 1,将把原始数据降到一维;
- copy:True 或False,默认为True,即是否需要将原始训练数据复制。
- whiten:True 或False,默认为False,即是否白化,使得每个特征具有相同的方差。
4.PCA对象的属性
- explained_variance_ratio_:返回所保留各个特征的方差百分比,如果n_components没有赋值,则所有特征都会返回一个数值且解释方差之和等于1。
- n_components_:返回所保留的特征个数。
5.PCA常用的方法
- fit(X): 用数据X来训练PCA模型。
- fit_transform(X):用X来训练PCA模型,同时返回降维后的数据。
- inverse_transform(newData) :将降维后的数据转换成原始数据,但可能不会完全一样,会有些许差别。
- transform(X):将数据X转换成降维后的数据,当模型训练好后,对于新输入的数据,也可以用transform方法来降维。
简单的栗子:
import numpy as np
from sklearn.decomposition import PCA
X = np.array([[-1, -1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]])
pca = PCA(n_components=2)
newX = pca.fit_transform(X)
print(X)
Out[365]:
[[-1 -1]
[-2 -1]
[-3 -2]
[ 1 1]
[ 2 1]
[ 3 2]]
print(newX)
Out[366]:
array([[ 1.38340578, 0.2935787 ],
[ 2.22189802, -0.25133484],
[ 3.6053038 , 0.04224385],
[-1.38340578, -0.2935787 ],
[-2.22189802, 0.25133484],
[-3.6053038 , -0.04224385]])
print(pca.explained_variance_ratio_)
[ 0.99244289 0.00755711]
可以发现第一个特征可以99.24%表达整个数据集,因此我们可以降到1维:
pca = PCA(n_components=1)
newX = pca.fit_transform(X)
print(pca.explained_variance_ratio_)
[ 0.99244289]
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
data = load_iris() # 以字典形式加载鸢尾花数据集
y = data.target # 使用y表示数据集中的标签
X = data.data # 使用X表示数据集中的属性数据
pca = PCA(n_components=2) # 加载PCA算法,设置降维后主成分数目为2
reduced_X = pca.fit_transform(X) # 对原始数据进行降维,保存在reduced_X中
'''
print(reduced_X)得到:
[[-2.68412563 0.31939725]
[-2.71414169 -0.17700123]
[-2.88899057 -0.14494943]
[-2.74534286 -0.31829898]
[-2.72871654 0.32675451]
...............
[ 1.90094161 0.11662796]
[ 1.39018886 -0.28266094]]
'''
red_x, red_y = [], [] # 第一类数据点
blue_x, blue_y = [], [] # 第二类数据点
green_x, green_y = [], [] # 第三类数据点
for i in range(len(reduced_X)): # 按照鸢尾花的类别将降维后的数据点保存在不同的列表中。
if y[i] == 0:
red_x.append(reduced_X[i][0])
red_y.append(reduced_X[i][1])
elif y[i] == 1:
blue_x.append(reduced_X[i][0])
blue_y.append(reduced_X[i][1])
else:
green_x.append(reduced_X[i][0])
green_y.append(reduced_X[i][1])
plt.scatter(red_x, red_y, c='r', marker='x')
plt.scatter(blue_x, blue_y, c='b', marker='D')
plt.scatter(green_x, green_y, c='g', marker='.')
plt.show()
#可以看出降维后的数据集仍能够分成三类,并没有改变数据的质量
已知降到2维后的鸢尾花数据集为reduced_X=
[[-2.68412563 0.31939725]
[-2.71414169 -0.17700123]
[-2.88899057 -0.14494943]
[-2.74534286 -0.31829898]
[-2.72871654 0.32675451]
...............
[ 1.90094161 0.11662796]
[ 1.39018886 -0.28266094]]
对应的标签为y=
[0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2
2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
2 2]
我们要根据标签y里面的数字将reduced_X分开存储到六个列表里
red_x, red_y = [], [] # 第一类数据点
blue_x, blue_y = [], [] # 第二类数据点
green_x, green_y = [], [] # 第三类数据点
for i in range(len(reduced_X)):
if y[i] == 0:
red_x.append(reduced_X[i][0])
red_y.append(reduced_X[i][1])
elif y[i] == 1:
blue_x.append(reduced_X[i][0])
blue_y.append(reduced_X[i][1])
else:
green_x.append(reduced_X[i][0])
green_y.append(reduced_X[i][1])
以下例题是未搞明白的
4.无监督学习中的 降维算法之 NMF降维算法–




实例


(以下代码只做了解)
import matplotlib.pyplot as plt
from sklearn import decomposition
#加载PCA算法包
from sklearn.datasets import fetch_olivetti_faces
#加载人脸数据集
from numpy.random import RandomState
#加载RandomState用于创建随机种子
n_row,n_col = 2,3
#设置图像展示时的排列情况,2行三列
n_components = n_row * n_col
#设置提取的特征的数目
image_shape = (64,64)
#设置人脸数据图片的大小
dataset = fetch_olivetti_faces(shuffle=True,random_state=RandomState(0))
faces = dataset.data#加载数据,并打乱顺序
def plot_gallery(title,images,n_col=n_col,n_row=n_row):
plt.figure(figsize=(2. * n_col,2.26 * n_row))#创建图片,并指定大小
plt.suptitle(title,size=16)#设置标题及字号大小
for i,comp in enumerate(images):
plt.subplot(n_row,n_col,i+1)#选择画制的子图
vmax = max(comp.max(),-comp.min())
plt.imshow(comp.reshape(image_shape),cmap=plt.cm.gray,interpolation='nearest',vmin=-vmax,vmax=vmax)#对数值归一化,并以灰度图形式显示
plt.xticks(())
plt.yticks(())#去除子图的坐标轴标签
plt.subplots_adjust(0.01,0.05,0.99,0.93,0.04,0.)
estimators=[('Eigenfaces - PCA using randomized SVD',decomposition.PCA(n_components=6,whiten=True)),('Non-negative components - NMF',decomposition.NMF(n_components=6,init='nndsvda',tol=5e-3))]
#NMF和PCA实例,将它们放在一个列表中
for name,estimators in estimators:#分别调用PCA和NMF
estimators.fit(faces)#调用PCA或NMF提取特征
components_=estimators.components_#获取提取特征
plot_gallery(name,components_[:n_components])
#按照固定格式进行排列
plt.show()#可视化
import numpy as np
import PIL.Image as Image #加载PIL包,用于加载创建图片
from sklearn.cluster import KMeans #加载Kmeans算法
def loadData(filePath):
data= []
img=Image.open(filePath)
m,n =img.size #获得图片大小(width, height)
for i in range(m):
for j in range(n):
x,y,z =img.getpixel((i,j)) #im.getpixel(x,y)返回给定位置的像素值。
data.append([x/256.0,y/256.0,z/256.0])#将每个像素点RGB颜色处理到0-1范围内,将颜色值存入data内
return np.mat(data),m,n #以矩阵的形式返回data,以及图片大小
imgData,row,col =loadData('bull.png') #调用函数,获取数据
km=KMeans(n_clusters=3) #聚类获得每个像素所属的类别
label =km.fit_predict(imgData)
label=label.reshape([row,col])
pic_new = Image.new("L",(row,col))#创建一张新的灰度图以保存聚类后的结果
for i in range(row):
for j in range(col):
pic_new.putpixel((i,j),int(256/(label[i][j]+1))) #im.putpixel((x,y),(r,g,b)) 在指定位置(x,y)处画一像素
pic_new.save("result-bull-4.jpg","JPEG") #以JPEG格式保存图像