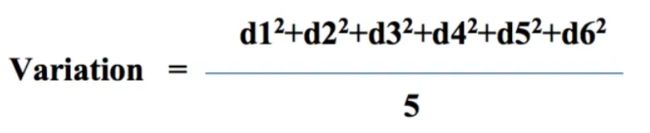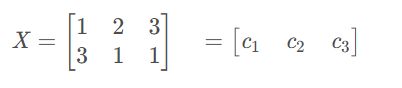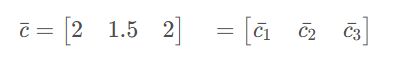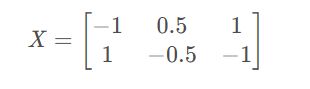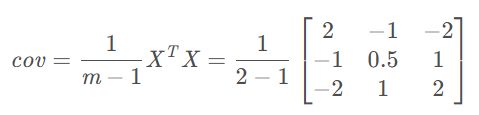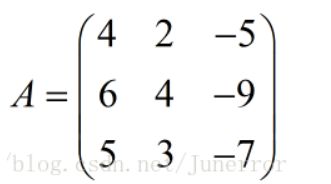PCA(主成分分析)的理解与应用(学习笔记)
PCA
主成分分析(Principal Component Analysis, PCA)是一种线性降维算法,也是一种常用的数据预处理(Pre-Processing)方法。它的目标是是用方差(Variance)来衡量数据的差异性,并将差异性较大的高维数据投影到低维空间中进行表示。绝大多数情况下,我们希望获得两个主成分因子:分别是从数据差异性最大和次大的方向提取出来的,称为PC1(Principal Component 1) 和 PC2(Principal Component 2)。
PCA的具体实现
| student_id | 1 | 2 | 3 | 4 | 5 | 6 |
| scores1 | 92 | 70 | 95 | 73 | 72 | 87 |
| scores2 | 74 | 87 | 70 | 92 | 97 | 74 |
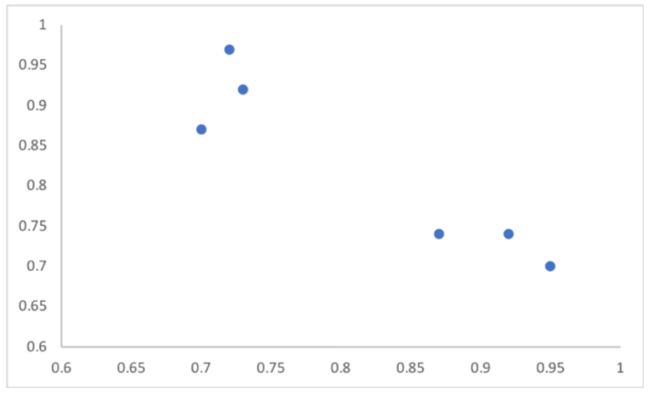
制作为散点图:
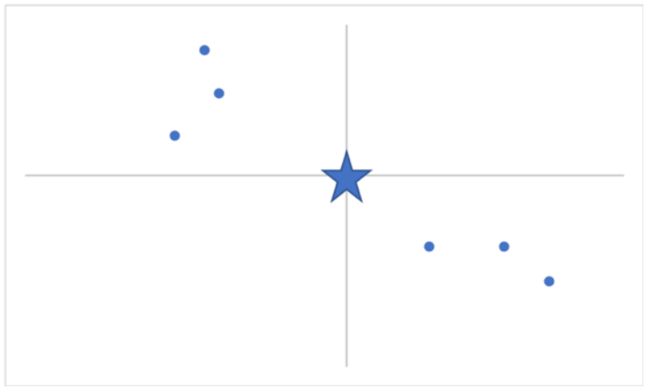
图中每个点代表了一个学生,X轴代表语文成绩,Y轴代表数学成绩。然后分别取所有样本的X平均值和Y平均值,并将这两个值变为X、Y坐标,在图中画出这个点(用五角星表示):
按照图中箭头所示方向,将整个坐标系平移,使原点与五角星重叠。这样就获得了一个新的平面直角坐标系:
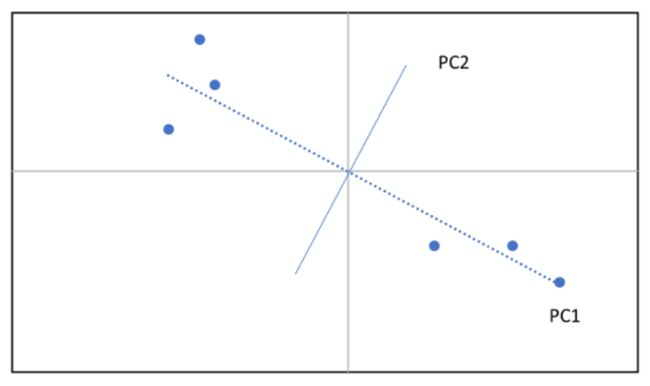
尽管此时坐标系和每个点的值都发生了变化,点与点之间的相对位置仍保持一致。找到这些点的最优拟合线(Line of Best Fit),也就找到了PC1,再通过原点做PC1的垂线,就找到PC2:
处理三维数组时便会产生第三个因子(PC3),以此类推,数据的维度越大,因子的数量也就越多。当维度大于等于4的时候,我们是无法想象出图像的,但PC4确实存在;假设有x个维度,便可以做x-1条垂线,就能得到PCx。接下来要做的便是选取最能代表数据差异性的两个因子,作为PC1和PC2。
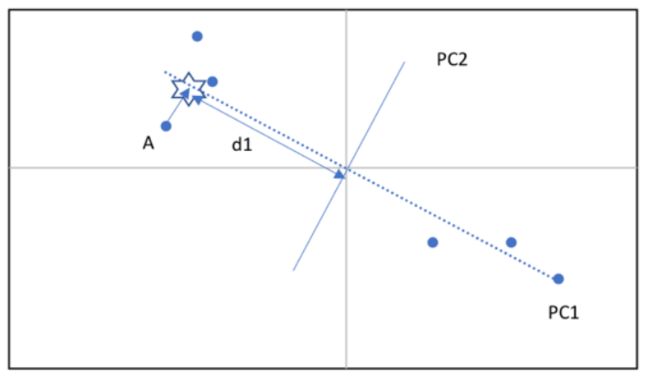
按照下图所示,将点A投影到PC1上(六角星的位置),并计算其与原点之间的距离称为d1:
其余的五个点也做同样操作,得出d2至d5,再求这六个距离的平方和,称为PC1的特征值(Eigenvalue)。然后将PC1的特征值除以总样本数量减一(n-1),就计算出了PC1的差异值(Variation)。
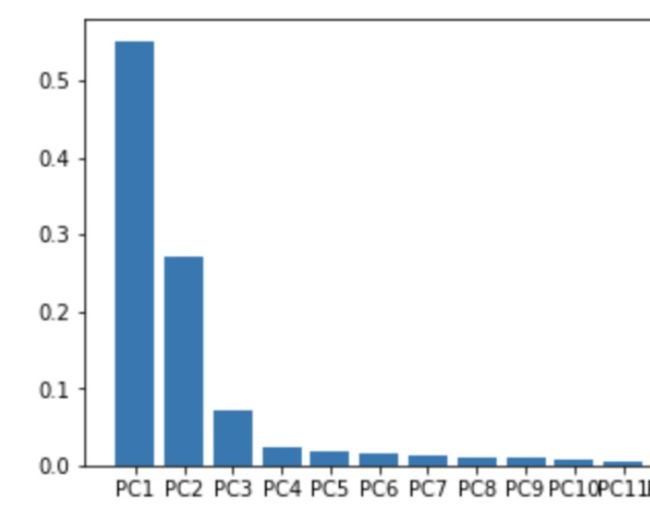
以此类推,并选择差异值最大的两个因子作为PC1 和 PC2。假设在某个三维数组中,获得了PC1、PC2和PC3的差异值分别为18,7,5。通过计算(18+7)/ (18+7+5) ≈ 83.3% 得到结论:PC1 和 PC2 代表了这个三维数组83.3%的差异性。在本次分析的13个因子中,PC1和PC2描述了整组数据约81%的差异性:
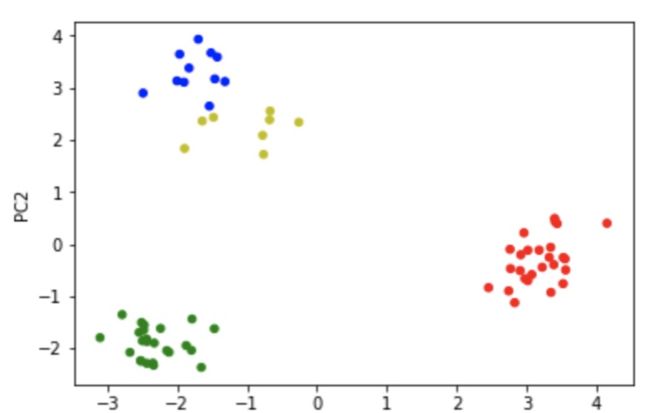
最后,再通过选中的PC1和PC2将样本映射回本身所在的坐标,就可以得到降维后的图像(PCA Plot)。
协方差矩阵基本知识点:
矩阵中的数据按行排列和按列排列求出的协方差矩阵是不同的,这里默认数据是按行排列。即每一行是一个observation(样本),那么每一列就是一个随机变量(特征)。
举个例子,矩阵X按行排列:
1.求每个维度的平均值
2.将X的每一列减去平均值
3.计算协方差矩阵
矩阵特征值和特征向量计算方法:
计算A的特征值和特征向量
![]()
计算行列式得
![]()
化简得:
![]()
得到特征值:
![]()
![]()
![]()
![]() 化简得:
化简得:![]()
![]()
![]()
令![]() 得到特征矩阵:
得到特征矩阵:![]()
同理,当![]() 得:
得:
![]()
![]()
令![]() 得到特征矩阵:
得到特征矩阵:![]()
代码实现(依次由繁到简实现效果)
方法一:
# -*- coding: utf-8 -*-
"""
@author: 绯雨千叶
用PCA求样本矩阵X的K阶降维矩阵Z
请保证输入的样本矩阵X shape=(m, n),m行样例,n个特征
"""
import numpy as np
class PCA(object):
def __init__(self, X, K):
self.X = X # 训练样本矩阵X
self.K = K # X的降维矩阵的阶数,即X要特征降维成k阶
self.centrX = [] # 矩阵X的中心化
self.C = [] # 样本集的协方差矩阵C
self.U = [] # 样本矩阵X的降维转换矩阵
self.Z = [] # 样本矩阵X的降维矩阵Z
self.centrX = self._centralized()
self.C = self._conv()
self.U = self._U()
self.Z = self._Z() # Z=XU求得
def _centralized(self): # 矩阵X的中心化
print('样本矩阵X:\n', self.X)
centrX = []
mean = np.array([np.mean(attr) for attr in self.X.T]) # mean()函数功能:求取均值;样本集的特征均值(每一列的平均数)
centrX = self.X - mean # 样本集的中心化(减去他那行的特征均值)
print('样本集的特征均值:\n', mean)
print('样本矩阵X的中心化centrX:\n', centrX)
return centrX
def _conv(self): # 求样本矩阵X的协方差矩阵C
ns = np.shape(self.centrX)[0] # 样本集的样例总数
C = np.dot(self.centrX.T, self.centrX) / (ns - 1) # 样本矩阵的协方差矩阵C;.dot向量点积和矩阵乘法
print('样本矩阵X的协方差矩阵C:\n', C)
return C
def _U(self): # 求X的降维转换矩阵U, shape=(n,k), n是X的特征维度总数,k是降维矩阵的特征维度
a, b = np.linalg.eig(self.C) # 第一个返回值是X的协方差矩阵C的特征值,第二个返回值是特征向量
print('样本集的协方差矩阵C的特征值:\n', a)
print('样本集的协方差矩阵C的特征向量:\n', b)
ind = np.argsort(-1 * a) # 给出特征值降序的topK的索引序列
UT = [b[:, ind[i]] for i in range(self.K)] # 构建K阶降维的降维转换矩阵U
U = np.transpose(UT)
print('%d阶降维转换矩阵U:\n' % self.K, U)
return U
def _Z(self): # 按照Z=XU求降维矩阵Z, shape=(m,k), n是样本总数,k是降维矩阵中特征维度总数
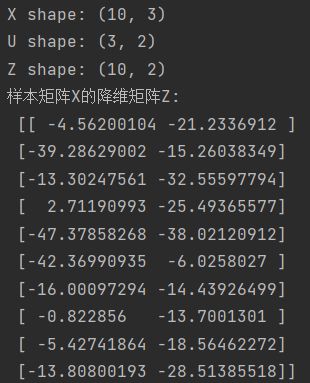
Z = np.dot(self.X, self.U)
print('X shape:', np.shape(self.X))
print('U shape:', np.shape(self.U))
print('Z shape:', np.shape(Z))
print('样本矩阵X的降维矩阵Z:\n', Z)
return Z
if __name__ == '__main__':
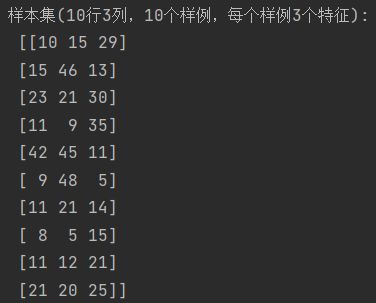
'10样本3特征的样本集, 行为样例,列为特征维度'
X = np.array([[10, 15, 29],
[15, 46, 13],
[23, 21, 30],
[11, 9, 35],
[42, 45, 11],
[9, 48, 5],
[11, 21, 14],
[8, 5, 15],
[11, 12, 21],
[21, 20, 25]])
K = np.shape(X)[1] - 1
print('样本集(10行3列,10个样例,每个样例3个特征):\n', X)
pca = PCA(X, K)效果展示:
方法二:
# coding=utf-8
"""
@author: 绯雨千叶
"""
import numpy as np
class PCA():
def __init__(self, n_components):
self.n_components = n_components
def fit_transform(self, X):
self.n_features_ = X.shape[1]
# 求协方差矩阵
X = X - X.mean(axis=0)
self.covariance = np.dot(X.T, X) / X.shape[0]
# 求协方差矩阵的特征值和特征向量
eig_vals, eig_vectors = np.linalg.eig(self.covariance)
# 获得降序排列特征值的序号
idx = np.argsort(-eig_vals)
# 降维矩阵
self.components_ = eig_vectors[:, idx[:self.n_components]]
# 对X进行降维
return np.dot(X, self.components_)
# 调用
pca = PCA(n_components=2)
X = np.array(
[[-1, 2, 66, -1], [-2, 6, 58, -1], [-3, 8, 45, -2], [1, 9, 36, 1], [2, 10, 62, 1], [3, 5, 83, 2]]) # 导入数据,维度为4
newX = pca.fit_transform(X)
print(newX) # 输出降维后的数据效果展示:
方法三:
#coding=utf-8
"""
@author: 绯雨千叶
"""
import numpy as np
from sklearn.decomposition import PCA
X = np.array([[-1,2,66,-1], [-2,6,58,-1], [-3,8,45,-2], [1,9,36,1], [2,10,62,1], [3,5,83,2]]) #导入数据,维度为4
pca = PCA(n_components=2) #降到2维
pca.fit(X) #训练
newX=pca.fit_transform(X) #降维后的数据
PCA(copy=True, n_components=2, whiten=False)
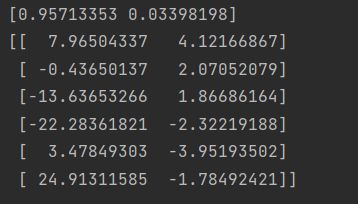
print(pca.explained_variance_ratio_) #输出贡献率
print(newX) #输出降维后的数据效果展示:
sklearn.decomposition.PCA(n_components=None, copy=True, whiten=False)参数:
n_components:
意义:PCA算法中所要保留的主成分个数n,也即保留下来的特征个数n
类型:int 或者 string,缺省时默认为None,所有成分被保留。
赋值为int,比如n_components=1,将把原始数据降到一个维度。
赋值为string,比如n_components='mle',将自动选取特征个数n,使得满足所要求的方差百分比。
copy:
类型:bool,True或者False,缺省时默认为True。意义:表示是否在运行算法时,将原始训练数据复制一份。若为True,则运行PCA算法后,原始训练数据的值不 会有任何改变,因为是在原始数据的副本上进行运算;若为False,则运行PCA算法后,原始训练数据的 值会改,因为是在原始数据上进行降维计算。
whiten:
类型:bool,缺省时默认为False
意义:白化,使得每个特征具有相同的方差。
PCA属性:
- components_ :返回具有最大方差的成分。
- explained_variance_ratio_:返回 所保留的n个成分各自的方差百分比。
- n_components_:返回所保留的成分个数n。
- mean_:
- noise_variance_:
PCA方法:
1、fit(X,y=None)
fit(X),表示用数据X来训练PCA模型。
函数返回值:调用fit方法的对象本身。比如pca.fit(X),表示用X对pca这个对象进行训练。
拓展:fit()可以说是scikit-learn中通用的方法,每个需要训练的算法都会有fit()方法,它其实就是算法中的“训练”这一步骤。因为PCA是无监督学习算法,此处y自然等于None。
2、fit_transform(X)
用X来训练PCA模型,同时返回降维后的数据。
newX=pca.fit_transform(X),newX就是降维后的数据。
3、inverse_transform()
将降维后的数据转换成原始数据,X=pca.inverse_transform(newX)
4、transform(X)
将数据X转换成降维后的数据。当模型训练好后,对于新输入的数据,都可以用transform方法来降维。
此外,还有get_covariance()、get_precision()、get_params(deep=True)、score(X, y=None)等方法,以后用到再补充吧。