「深度学习之优化算法」(六)遗传算法
1. 遗传算法简介
遗传算法(Genetic Algorithms,GA)是一种模拟自然中生物的遗传、进化以适应环境的智能算法。由于其算法流程简单,参数较少优化速度较快,效果较好,在图像处理、函数优化、信号处理、模式识别等领域有着广泛的应用。
在遗传算法(GA)中,每一个待求问题的候选解被抽象成为种群中一个个体的基因。种群中个体基因的好坏由表示个体基因的候选解在待求问题中的所的得值来评判。种群中的个体通过与其他个体交叉产生下一代,每一代中个体均只进行一次交叉。两个进行交叉的个体有一定几率交换一个或者多个对应位的基因来产生新的后代。每个后代都有一定的概率发生变异。发生变异的个体的某一位或某几位基因会变异成其他值。最终将以个体的适应度值为概率选取个体保留至下一代。
2. 算法流程
遗传算法启发于生物的繁殖与dna的重组,本次的主角选什么呢?还是根据大家熟悉的孟德尔遗传规律选豌豆吧,选动物的话又会有人疑车,还是植物比较好,本次的主角就是它了。
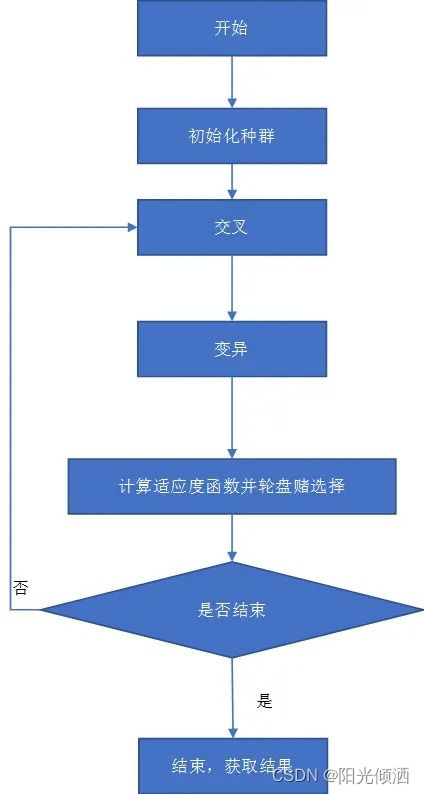
遗传算法包含三个操作(算子):交叉,变异和选择操作。下面我们将详细介绍这三个操作。
大多数生物的遗传信息都储存在DNA,一种双螺旋结构的复杂有机化合物。其含氮碱基为腺嘌呤、鸟嘌呤、胞嘧啶及胸腺嘧啶。
遗传算法的个体结构受DNA的分子结构启发,基于计算机的二进制,其编码内容如下:
| 编号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
|---|---|---|---|---|---|---|---|---|---|---|
| 基因 | 0 | 1 | 1 | 0 | 1 | 1 | 0 | 1 | 0 | 0 |
表格中表示了一个有10个基因的个体,它们每一个基因的值为0或者1。
2.1交叉
生物的有性生殖一般伴随着基因的重组。遗传算法中父辈和母辈个体产生子代个体的过程称为交叉。
| 编号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
|---|---|---|---|---|---|---|---|---|---|---|
| A豌豆基因 | 0 | 1 | 1 | 0 | 1 | 1 | 0 | 1 | 0 | 0 |
| B豌豆基因 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 |
表中给出了两个豌豆的基因,它们均有10个等位基因(即编号相同的基因)。
遗传算法的交叉过程会在两个个体中随机选择1位或者n位基因进行交叉,即这两个个体交换等位基因。
如,A豌豆和B豌豆在第6位基因上进行交叉,则其结果如下
| 编号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
|---|---|---|---|---|---|---|---|---|---|---|
| A豌豆基因 | 0 | 1 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 |
| B豌豆基因 | 1 | 1 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 0 |
当两个个体交叉的等位基因相同时,交叉过程也有可能没有产生新的个体,如交叉A豌豆和B豌豆的第2位基因时,交叉操作并没有产生新的基因。
| 编号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
|---|---|---|---|---|---|---|---|---|---|---|
| A豌豆基因 | 0 | 1 | 1 | 0 | 1 | 1 | 0 | 1 | 0 | 0 |
| B豌豆基因 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 |
一般的会给群体设定一个交叉率,crossRate,表示会在群体中选取一定比例的个体进行交叉,交叉率相对较大,一般取值为0.8。
2.2变异
基因的变异是生物进化的一个主要因素。
遗传算法中变异操作相对简单,只需要将一个随机位基因的值修改就行了,因为其值只为0或1,那么当基因为0时,变异操作会将其值设为1,当基因值为1时,变异操作会将其值设为0。
| 编号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
|---|---|---|---|---|---|---|---|---|---|---|
| A豌豆基因 | 0 | 1 | 1 | 0 | 1 | 1 | 0 | 1 | 0 | 0 |
| A变异后基因 | 0 | 1 | 0 | 0 | 1 | 1 | 0 | 1 | 0 | 0 |
上图表示了A豌豆第3位基因变异后的基因编码。
与交叉率相似,变异操作也有变异率,alterRate,但是变异率会远低于交叉率,否则会产生大量的随机基因。一般变异率为0.05。
2.3选择
选择操作是遗传算法中的一个关键操作,它的主要作用就是根据一定的策略随机选择个体保留至下一代。适应度越优的个体被保留至下一代的概率越大。
实现上,我们经常使用“轮盘赌”来随机选择保留下哪个个体。
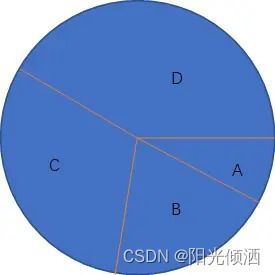
假设有4个豌豆A、B、C、D,它们的适应度值如下:
| 个体 | 适应度值 |
|---|---|
| A | 1 |
| B | 2 |
| C | 3 |
| D | 4 |
适应度值越大越好,则它们组成的轮盘如下图:
但由于轮盘赌选择是一个随机选择过程,A、B、C、D进行轮盘赌选择后产生的下一代也有可能出现A、A、A、A的情况,即虽然有些个体的适应度值不好,但是运气不错,也被选择留到了下一代。
遗产算法的三个主要操作介绍完了,下面我们来看看遗传算法的总体流程:
3.基因编码及操作细节
前面我们说了遗传算法的流程及各个操作,那么对于实际的问题我们应该如何将其编码为基因呢?
3.1二进制编码
对于计算机来所所有的数据都使用二进制数据进行存放,如float类型和double类型的数据。
float类型的数据将保存为32位的二进制数据:1bit(符号位) 8bits(指数位) 23bits(尾数位)
如-1.234567f,表示为二进制位10111111100111100000011001001011
| 类型 | 符号位1 | 指数位8 | 小数位23 |
|---|---|---|---|
| 值 | 1 | 01111111 | 00111100000011001001011 |
Double类型的数据将保存为64位的二进制数据:1bit(符号位) 11bits(指数位) 53bits(尾数位)
如-1.234567d,表示为二进制为1011111111110011110000001100100101010011100110111000100010000111
| 类型 | 符号位1 | 指数位11 | 小数位52 |
|---|---|---|---|
| 值 | 1 | 01111111111 | 001111000000110010010101001100110111000100010000111 |
可以看出同样的数值不同的精度在计算机中存储的内容也不相同。之前的适应度函数![]() ,由于有两个double类型的参数,故其进行遗传算法基因编码时,将有128位基因。
,由于有两个double类型的参数,故其进行遗传算法基因编码时,将有128位基因。
虽然基因数较多,但好在每个基因都是0或者1,交叉及变异操作非常简单。
3.2十进制编码
相比二进制编码,十进制编码的基因长度更短,适应度函数![]() 有两个输入参数,那么一个个体就有2个基因,但其交叉、变异操作相对复杂。
有两个输入参数,那么一个个体就有2个基因,但其交叉、变异操作相对复杂。
交叉操作
方案1:将一个基因作为一个整体,交换两个个体的等位基因。
交换前
| 编号 | 1 | 2 |
|---|---|---|
| A豌豆基因 | 10 | 20 |
| B豌豆基因 | 30 | 40 |
交换第1位基因后
| 编号 | 1 | 2 |
|---|---|---|
| A豌豆基因 | 30 | 20 |
| B豌豆基因 | 10 | 40 |
方案2:将两个个体的等位基因作为一个整体,使其和不变,但是值随机
交换前
| 编号 | 1 | 2 |
|---|---|---|
| A豌豆基因 | 10 | 20 |
| B豌豆基因 | 30 | 40 |
交换第1位基因后
| 编号 | 1 | 2 |
|---|---|---|
| A豌豆基因 | 21 | 20 |
| B豌豆基因 | 19 | 40 |
假设A、B豌豆的第一位基因的和为40,即![]() ,第一位基因的取值范围为0-30,那么A、B豌豆的第一位基因的取值范围为[10,30],即为[0,30]的随机数,。
,第一位基因的取值范围为0-30,那么A、B豌豆的第一位基因的取值范围为[10,30],即为[0,30]的随机数,。
变异操作,将随机的一位基因设置为该基因取值范围内的随机数即可。
3.3轮盘赌
这个过程说起来简单但其实现并不容易。
| 个体 | 适应度值 |
|---|---|
| A | 1 |
| B | 2 |
| C | 3 |
| D | 4 |
我们要将它们的值映射到一个轴上才能进行随机选择,毕竟我们无法去绘制一个轮盘来模拟这个过程
如图,将ABCD根据其值按顺序排列,取[0,10]内的随机数r,若r在[0,1]内则选择A,在(1,3]内则选择B,在(3,6]内则选择C,在(6,10]则选择D。
当然这仍然会有问题,即当D>>A、B、C时,假如它们的值分布如下
| 个体 | 适应度值 |
|---|---|
| A | 1 |
| B | 2 |
| C | 3 |
| D | 100 |
那么显然,选D的概率明显大于其他,根据轮盘赌的选择,下一代极有可能全是D的后代有没有办法均衡一下呢?
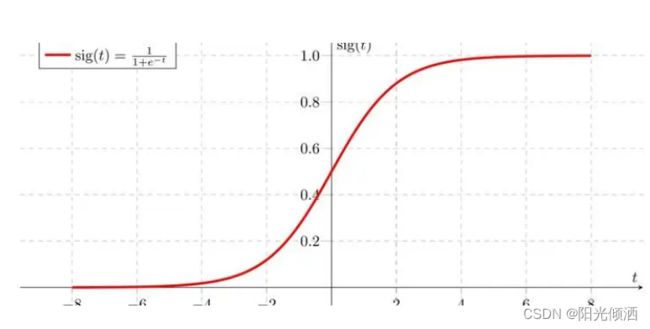
首先我想到了一个函数,
不要问我为什么我不知道什么是神经什么网络的,什么softmax、cnn统统没听说过。
取值为sigmod((fitness-mean)/(max-min)),现在再来计算一下ABCD的值,
| 个体 | 适应度值 |
|---|---|
| A | 0.435 |
| B | 0.438 |
| C | 0.441 |
| D | 0.677 |
这样一来,它们之间的差距没有之前那么大了,只要个体适应度值在均值以上那么它被保留至下一代的概率会相对较大,当然这样缩小了个体之间的差距,对真正优秀的个体来说不太公平,相对应,我们可以在每次选择过程中保留当前的最优个体到下一代,不用参与轮盘赌这个残酷的淘汰过程。
4.实验
由于遗传算法的收敛速度实在是太慢,区区50代,几乎得不到好的结果,so我们把它的最大迭代次数放宽到200代。
方案1.
使用二进制编码来进行求解
参数如下:
| 参数 | 值 |
|---|---|
| 问题维度(维度) | 2 |
| 豌豆的数量(种群数) | 20 |
| 繁殖次数(最大迭代次数) | 200 |
| crossRate(交叉率) | 0.8 |
| alterRate(变异率) | 0.05 |
| 取值范围 | (-100,100) |
| 编码方式 | 二进制 |
| 实验次数 | 10 |
求解过程如上图,可以看出基因收敛的很快,在接近20代时就图中就只剩一个点了,之后的点大概是根据变异操作产生。看一下最后的结果。
| 值 | |
|---|---|
| 最优值 | 0.0 |
| 最差值 | 1374.1828414659183 |
| 平均值 | 214.78194828105424 |
可以看出最好的结果已经得到了最优解,但是10次实验的最差值和平均值都差的令人发指。为什么会这样呢?
问题出在二进制编码上,由于double类型的编码有11位指数位和52位小数位,这会导致交叉、变异操作选到指数位和小数位的概率不均衡,在小数位上的修改对结果的影响太小而对指数为的修改对结果的影响太大,
如-1.234567d,表示为二进制为1011111111110011110000001100100101010011100110111000100010000111
| 类型 | 符号位1 | 指数位11 | 小数位52 |
|---|---|---|---|
| 值 | 1 | 01111111111 | 0011110000001100100101010011100110111000100010000111 |
对指数为第5位进行变异操作后的结果为-2.8744502924382686E-10,而对小数位第5为进行变异操作后的结果为-1.218942。可以看出这两部分对数值结果的影响太不均衡,得出较好的结果时大概率是指数位与解非常相近,否则很难得出好的结果,就像上面的最差值和均值一样。
所以使用上面的二进制编码不是一个好的基因编码方式,因此在下面的实验中,将使用十进制来进行试验。
方案2:
使用:十进制编码来进行求解
参数如下:
| 参数 | 值 |
|---|---|
| 问题维度(维度) | 2 |
| 豌豆的数量(种群数) | 20 |
| 繁殖次数(最大迭代次数) | 200 |
| crossRate(交叉率) | 0.8 |
| alterRate(变异率) | 0.05 |
| 取值范围 | (-100,100) |
| 编码方式 | 十进制 |
| 交叉方式 | 交换基因 |
| 实验次数 | 10 |
我们可以看到直到40代时,所有的个体才收束到一点,但随后仍不断的新的个体出现。我们发现再后面的新粒子总是在同一水平线或者竖直线上,因为交叉操作直接交换了两个个体的基因,那么他们会相互交换x坐标或者y坐标,导致新个体看起来像在一条直线上。
我们来看看这次的结果。
| 值 | |
|---|---|
| 最优值 | 0.4998897017064181 |
| 最差值 | 101.50731384880558 |
| 平均值 | 26.879257652054093 |
这次最优值没有得到最优解,但是最差值没有二进制那么差,虽然也不容乐观。使用交换基因的方式来进行交叉操作的搜索能力不足,加之轮盘赌的选择会有很大概率选择最优个体,个体总出现在矩形的边上。
下面我们先改变轮盘赌的选择策略,使用上面的sigmod函数方案,并且保留最优个体至下一代。
方案3:
使用:十进制编码来进行求解
参数如下:
| 参数 | 值 |
|---|---|
| 问题维度(维度) | 2 |
| 豌豆的数量(种群数) | 20 |
| 繁殖次数(最大迭代次数) | 200 |
| crossRate(交叉率) | 0.8 |
| alterRate(变异率) | 0.05 |
| 取值范围 | (-100,100) |
| 编码方式 | 十进制 |
| 交叉方式 | 交换基因 |
| 轮盘赌方式 | sigmod函数方案,保留最优至下一代 |
| 实验次数 | 10 |
看图好像跟之前的没什么区别,让我们们看看最终的结果:
| 值 | |
|---|---|
| 最优值 | 0.4504701892811481 |
| 最差值 | 18.879980044723276 |
| 平均值 | 6.865234201226049 |
可以看出,最优值没有什么变化,但是最差值和平均值有了较大的提升,说明该轮盘赌方案使算法的鲁棒性有了较大的提升。在每次保留最优个体的情况下,对于其他的个体的选择概率相对平均,sigmod函数使得即使适应度函数值相差不太大的个体被选到的概率相近,增加了基因的多样性。
方案4:
使用:十进制编码来进行求解,改变交叉方案,保持两个个体等位基因和不变的情况下随机赋值。
参数如下:
| 参数 | 值 |
|---|---|
| 问题维度(维度) | 2 |
| 豌豆的数量(种群数) | 20 |
| 繁殖次数(最大迭代次数) | 200 |
| crossRate(交叉率) | 0.8 |
| alterRate(变异率) | 0.05 |
| 取值范围 | (-100,100) |
| 编码方式 | 十进制 |
| 交叉方式 | 修改基因使其和不变 |
| 轮盘赌方式 | sigmod函数方案,保留最优至下一代 |
| 实验次数 | 10 |
上图可以看出该方案与之前有明显的不同,在整个过程中,个体始终遍布整个搜索空间,虽然新产生的个体大多还是集中在一个十字架型的位置上,但其他位置的个体比之前的方案要多。
看看结果,
| 值 | |
|---|---|
| 最优值 | 0.011758796459006677 |
| 最差值 | 2.2756330462780725 |
| 平均值 | 0.8849388632252244 |
这次的结果明显好于之前的所有方案,但仍可以看出,十进制的遗传算法的精度不高,只能找到最优解的附近,也有可能是算法的收敛速度实在太慢,还没有收敛到最优解。
5.总结
遗传算法的探究到此也告一段落,在研究遗传算法时总有一种力不从心的感觉,问题可能在于遗传算法只提出了一个大致的核心思想,其他的实现细节都需要自己去思考,而每个人的思维都不一样,一万个人能写出一万种遗传算法,其实不仅是遗传算法,后面的很多算法都是如此。
为什么没有对遗传算法的参数进行调优,因为遗传算法的参数过于简单,对结果的影响的可解释性较强,意义明显,实验的意义不大。
遗传算法由于是模仿了生物的进化过程,因此我感觉它的求解速度非常的慢,而且进化出来的结果不一定是最适应环境的,就像人的阑尾、视网膜结构等,虽然不是最佳的选择但是也被保留到了今天。生物的进化的随机性较大,要不是恐龙的灭绝,也不会有人类的统治,要不是人类有两只手,每只手有5根手指,也不会产生10进制。
以下指标纯属个人yy,仅供参考
| 指标 | 星数 |
|---|---|
| 复杂度 | ★★★☆☆☆☆☆☆☆ |
| 收敛速度 | ★☆☆☆☆☆☆☆☆☆ |
| 全局搜索 | ★★★★★★★☆☆☆ |
| 局部搜索 | ★☆☆☆☆☆☆☆☆☆ |
| 优化性能 | ★★★★☆☆☆☆☆☆ |
| 跳出局部最优 | ★★★★★★☆☆☆☆ |
| 改进点 | ★★★★★★★☆☆☆ |