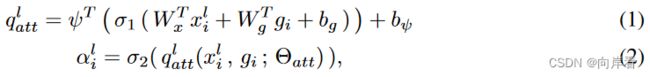Attention U-Net:Learning Where to Look for the Pancreas论文总结和代码实现
论文: https://arxiv.org/abs/1804.03999
中文版:https://blog.csdn.net/hhw999/article/details/110134398
源码: https://github.com/ozan-oktay/Attention-Gated-Networks
目录
一、论文背景和出发点
二、创新点
三、Attention U-Net的具体实现
四、实验
五、结论
一、论文背景和出发点
本文提出了一种用于医学成像的新型注意门(AG)模型,该模型可以自动学习聚焦于不同形状和大小的目标结构。
使用AG训练的模型可以不显著地学习输入图像中需要抑制的不相关区域,同时突出对特定任务有用的显著特征。这样就无需再使用级联卷积神经网络(CNNs)的显式外部组织/器官定位模块。
AGs可以很容易地集成到标准的CNN架构中,如U-Net模型,只需最少的计算开销,同时提高了模型的灵敏度和预测精度。
二、创新点
1. 提出了基于网格的门控,使注意系数更聚焦于局部区域。该方法可以用于密集预测。
2. 提出的soft-attention技术是第一个用于医学成像任务的前馈CNN模型。提出的注意力门可以替代图像分类和图像分割框架中的外部器官定位模型中使用的hard-attention方法。
3. 提出了一种对标准U-Net模型的扩展,在不需要复杂启发式的情况下提高模型对前景像素的灵敏度。
AGs中的特征选择性是通过使用在较粗尺度上提取的上下文信息(门控)来实现的。 注意力门(AG)通过跳跃连接的方式过滤被传播的特征。
三、Attention U-Net的具体实现
1. AG
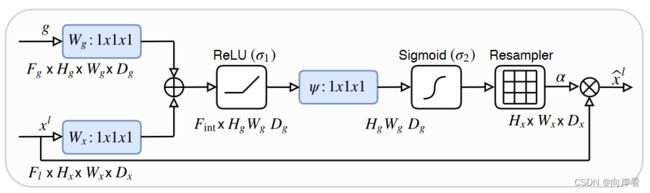
所提出的加性注意门(AG)示意图,如下:
目的:抑制不相关背景区域的特征响应,突出IOU区域。
方法:输入特征(![]() )使用在AG中计算的注意系数(α)进行缩放。
)使用在AG中计算的注意系数(α)进行缩放。
步骤:首先,对输入特征![]() 做1x1的卷积操作,同时也对与
做1x1的卷积操作,同时也对与![]() 同一层的下采样特征
同一层的下采样特征![]() 也做1x1的卷积操作,然后,将卷积后的两个输出特征相加,将相加结果做relu激活,再然后,对激活结果做1x1x1的卷积操作,将特征图通道数变换为1(原文中说这里是线性变换??),其次,对线性变换结果做sigmoid激活,再进行resample(整形,源码中没有这一步),得到一个与原特征大小一致1维的权重矩阵
也做1x1的卷积操作,然后,将卷积后的两个输出特征相加,将相加结果做relu激活,再然后,对激活结果做1x1x1的卷积操作,将特征图通道数变换为1(原文中说这里是线性变换??),其次,对线性变换结果做sigmoid激活,再进行resample(整形,源码中没有这一步),得到一个与原特征大小一致1维的权重矩阵![]() ,最后,权重矩阵
,最后,权重矩阵![]() 与输入特征
与输入特征![]() 相乘,返回一个新的特征图
相乘,返回一个新的特征图![]() 。对应算子公式如下:
。对应算子公式如下:
![]()
其中,![]() 为与
为与![]() 同一层下采样得到的特征,
同一层下采样得到的特征,![]() ,
,![]() 是1x1的卷积操作,
是1x1的卷积操作,![]() ,
,![]() 是偏置项,
是偏置项,![]() 是1x1x1的卷积操作,
是1x1x1的卷积操作,![]() 是softmax激活函数,
是softmax激活函数,![]() 是sigmoid激活函数,
是sigmoid激活函数,![]() 为权重矩阵。
为权重矩阵。
详情可见作者给出的2D unet源码:
import numpy as np
import torch
import torch.nn as nn
from torchsummary import summary
class Attention_block(nn.Module):
def __init__(self, F_g, F_l, F_int):
super(Attention_block, self).__init__()
self.W_g = nn.Sequential(
nn.Conv2d(F_g, F_int, kernel_size=1, stride=1, padding=0, bias=True),
nn.BatchNorm2d(F_int)
)
self.W_x = nn.Sequential(
nn.Conv2d(F_l, F_int, kernel_size=1, stride=1, padding=0, bias=True),
nn.BatchNorm2d(F_int)
)
self.psi = nn.Sequential(
nn.Conv2d(F_int, 1, kernel_size=1, stride=1, padding=0, bias=True),
nn.BatchNorm2d(1),
nn.Sigmoid()
)
self.relu = nn.ReLU(inplace=True)
def forward(self, g, x):
g1 = self.W_g(g)
x1 = self.W_x(x)
psi = self.relu(g1 + x1)
psi = self.psi(psi)
return x * psi
class conv_block(nn.Module):
def __init__(self, ch_in, ch_out):
super(conv_block, self).__init__()
self.conv = nn.Sequential(
nn.Conv2d(ch_in, ch_out, kernel_size=3, stride=1, padding=1, bias=True),
nn.BatchNorm2d(ch_out),
nn.ReLU(inplace=True),
nn.Conv2d(ch_out, ch_out, kernel_size=3, stride=1, padding=1, bias=True),
nn.BatchNorm2d(ch_out),
nn.ReLU(inplace=True)
)
def forward(self, x):
return self.conv(x)
class up_conv(nn.Module):
def __init__(self, ch_in, ch_out):
super(up_conv, self).__init__()
self.up = nn.Sequential(
nn.Upsample(scale_factor=2),
nn.Conv2d(ch_in, ch_out, kernel_size=3, stride=1, padding=1, bias=True),
nn.BatchNorm2d(ch_out),
nn.ReLU(inplace=True)
)
def forward(self, x):
return self.up(x)
class AttU_Net(nn.Module):
def __init__(self, n_channels=3, n_classes=1, scale_factor=1):
super(AttU_Net, self).__init__()
filters = np.array([64, 128, 256, 512, 1024])
filters = filters // scale_factor
self.n_channels = n_channels
self.n_classes = n_classes
self.scale_factor = scale_factor
self.Maxpool = nn.MaxPool2d(kernel_size=2, stride=2)
self.Conv1 = conv_block(ch_in=n_channels, ch_out=filters[0])
self.Conv2 = conv_block(ch_in=filters[0], ch_out=filters[1])
self.Conv3 = conv_block(ch_in=filters[1], ch_out=filters[2])
self.Conv4 = conv_block(ch_in=filters[2], ch_out=filters[3])
self.Conv5 = conv_block(ch_in=filters[3], ch_out=filters[4])
self.Up5 = up_conv(ch_in=filters[4], ch_out=filters[3])
self.Att5 = Attention_block(F_g=filters[3], F_l=filters[3], F_int=filters[2])
self.Up_conv5 = conv_block(ch_in=filters[4], ch_out=filters[3])
self.Up4 = up_conv(ch_in=filters[3], ch_out=filters[2])
self.Att4 = Attention_block(F_g=filters[2], F_l=filters[2], F_int=filters[1])
self.Up_conv4 = conv_block(ch_in=filters[3], ch_out=filters[2])
self.Up3 = up_conv(ch_in=filters[2], ch_out=filters[1])
self.Att3 = Attention_block(F_g=filters[1], F_l=filters[1], F_int=filters[0])
self.Up_conv3 = conv_block(ch_in=filters[2], ch_out=filters[1])
self.Up2 = up_conv(ch_in=filters[1], ch_out=filters[0])
self.Att2 = Attention_block(F_g=filters[0], F_l=filters[0], F_int=filters[0] // 2)
self.Up_conv2 = conv_block(ch_in=filters[1], ch_out=filters[0])
self.Conv_1x1 = nn.Conv2d(filters[0], n_classes, kernel_size=1, stride=1, padding=0)
def forward(self, x):
# encoding path
x1 = self.Conv1(x)
x2 = self.Maxpool(x1)
x2 = self.Conv2(x2)
x3 = self.Maxpool(x2)
x3 = self.Conv3(x3)
x4 = self.Maxpool(x3)
x4 = self.Conv4(x4)
x5 = self.Maxpool(x4)
x5 = self.Conv5(x5)
# decoding + concat path
d5 = self.Up5(x5)
x4 = self.Att5(g=d5, x=x4)
d5 = torch.cat((x4, d5), dim=1)
d5 = self.Up_conv5(d5)
d4 = self.Up4(d5)
x3 = self.Att4(g=d4, x=x3)
d4 = torch.cat((x3, d4), dim=1)
d4 = self.Up_conv4(d4)
d3 = self.Up3(d4)
x2 = self.Att3(g=d3, x=x2)
d3 = torch.cat((x2, d3), dim=1)
d3 = self.Up_conv3(d3)
d2 = self.Up2(d3)
x1 = self.Att2(g=d2, x=x1)
d2 = torch.cat((x1, d2), dim=1)
d2 = self.Up_conv2(d2)
d1 = self.Conv_1x1(d2)
return d1
if __name__ == '__main__':
device = torch.device("cuda:0" if torch.cuda.is_available() else "cpu")
net = AttU_Net().to(device)
# 打印网络结构和参数
summary(net, (3, 224, 224))这里增加了主函数,方便观察模型结构。
2. 将AG整合到UNet
目的:消除跳跃连接产生的不相关和嘈杂的响应(加强特征和去噪)。
方法:在每次上采样拼接操作之前,添加AG模块。AGs在前向传播和反向传播时通过对神经元的激活操作,减少背景区域产生的权重,加强对IOU区域的提取。
作者对![]() 层卷积参数的更新方式的过程进行了推导,对应推导公式如下:
层卷积参数的更新方式的过程进行了推导,对应推导公式如下:
四、实验
数据集:已标注的150例胃癌患者腹部3D CT扫描(CT-150)图像、NIH-TCIA(胰脏数据集)。
训练分配比例:进行训练(120)和测试(30);训练(30)和测试(120)。
评估指标:dice score、surface to surface distance(s2s)。
实验1:
由上图可见,胰腺预测的结果表明,注意力门(AGs)通过提高模型的表达能力(AGs提高了前景区域的提取率)来提高recall值。
实验2:
各种当前较为先进的CT胰腺分割模型的结果,与att u-net相比,att u-net有显著提升。
预测效果:
五、结论
提出了一种新的用于医学图像分割的注意力门控模型。该方法消除了使用额外目标定位模型的必要性。所提出的方法是通用的和模块化的,因此它可以很容易地应用于图像分类和回归问题,如在自然图像分析和机器翻译的例子。实验结果表明,所提出的AGs对组织/器官的识别和定位非常有利。对于可变的小尺寸器官,如胰腺,这一点尤其正确,而对于全局的分类任务,预期也会有类似的行为。
参考博文:图像分割UNet系列------Attention Unet详解