杂谈 | 人类微生物组研究:解析挑战与前景
谷禾健康
人类微生物组研究正在从描述关联发展到了解整个微生态对人类的影响。虽然存在挑战,但在应用数据驱动的微生物组诊断和干预方面正在取得进展,这可能会在未来十年内带来精准医学的突破。
本文我们来探讨关于微生物组的研究进展及其对人类健康的影响及应用。
01
对微生物组认识、理解的历程:
简单 ➞ 复杂,潜力 ➞ 挑战
随着对微生物组的理解深入,发现巨大的潜力
过去 20 年,我们对微生物组在人类宿主生理学中作用的理解发生了重大转变。
开创性研究表明,与人类细胞和基因相比,原核生物为人类“全生物”贡献了相同数量的细胞和多倍的基因,同时影响哺乳动物从消化和新陈代谢过程到免疫甚至大脑功能。
微生物组的变化与越来越多的人类疾病有关,包括癌症。这些发现使人们普遍希望,积累的关于宿主-微生物组相互作用的知识,将迅速转化为人类疾病的治疗方法。
对微生物组的认识开始客观,同时意识到复杂性
随着该领域的稳步成熟,科学家们开始更好地了解整个微生物组。他们意识到这并不像他们最初想的那么简单。随着他们开始了解微生物之间的相互作用是多么复杂,以及将微生物组用于人类治疗有多么大的挑战,最初围绕它的兴奋和炒作开始平息。
面临挑战,需要综合多方面的努力
要克服这些挑战,许多研究者需要进行长期、数据驱动的研究。他们要综合各种看起来不相干的科学和医学学科,包括进化论、人类学和营养学等等,还要结合新技术的开发和优化,以及注意事项和混杂因素的识别和最小化,共同推动了这一探索。他们的目标是要弄清楚宿主和微生物组之间的互动关系和分子机制,以期找到因果关系。虽然充满了挑战,但只要大家协作一致,一定可以获得成果。这些成果将在未来十年内实现对可利用的治疗性微生物组目标进行更广泛和可重复的识别和调节。
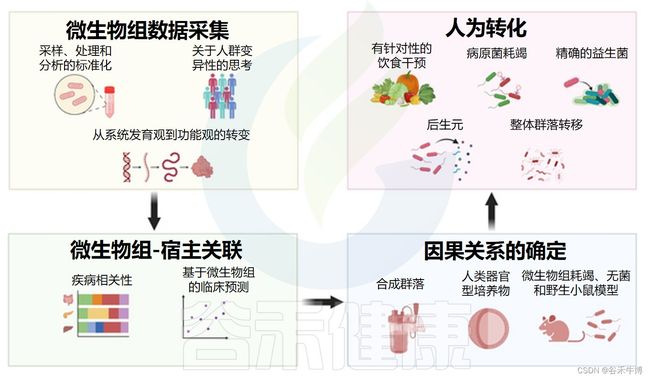
微生物组研究中的挑战和新兴方法
doi: 10.1371/journal.pbio.3002053
02
目前微生物研究进展:
面临的障碍 & 解决方案
本世纪第一个十年,研究微生物组结构表征、对生理功能的重要性,由此衍生到各个领域
事实上,本世纪微生物组研究的第一个十年,集中于证明肠道微生物组对生理功能的重要性,以及在各种生理和疾病背景下基于下一代测序的微生物组群落结构表征。
这带来了微生物组与各种健康结果之间的关联、相关性和预测的识别,报告规模和细节的不断提升。与此同时,该领域广泛描述了微生物特征响应多种因素的动态调节,例如饮食、宿主免疫 、基因组学和昼夜振荡。
▼
障碍一
不同研究之间的差异
随着这些重大进展,人们也认识到了主要的技术和概念障碍,这些障碍对微生物组发现的解释、概括和转化到临床具有挑战性。
使微生物组相关观察结果的解释复杂化的技术挑战之一是:不同研究之间样本收集、处理和分析方法的广泛差异。
此外,在微生物组处理和分析级联的每个步骤中发生的污染和注释错误可能会引入伪影和偏差,这些伪影和偏差难以解决并使真实和虚假生物信号之间的区别复杂化。
解决方案:将真实信号与混杂因素分开
了解与这些步骤中的每一个相关的范围、限制和混杂因素,对于将真实信号与混杂因素分开至关重要,特别是在微生物丰度低或无微生物丰度的样本。更好地协调这些方法,加上多种技术和生物控制,可以更准确地解释可以在人群、地域、性别和种族之间推广和复制。
然而,如果不冒技术可变性和多样性减少的风险,微生物组处理和分析标准化就不能过度执行,而技术可变性和多样性构成了推动研究创新的引擎。
▼
障碍二
微生物群结构内在的个体间变异性
大多数人类微生物组研究面临的一个概念挑战是,微生物群落结构中内在的和生理上重要的个体间变异性。
如何在大样本人群数据上去识别和消除这些差异,应该是目前微生物组特征分析面临的巨大分析挑战,因为它们代表了一种固有的、难以解决的“噪音”。
解决方案:基于深度人工智能的方法等创新方法
可以识别和利用不同的微生物组配置来探索微生物组对个体人类表型的贡献,范围从血糖反应到补充剂和食物携带相同疾病易感位点的个体之间的不同疾病表现。
总而言之,使用基于深度人工智能的方法等创新方法应对技术和生物变异性的挑战,将加深微生物组知识与精准医学的合理整合。
▼
障碍三
微生物组-人类特征的因果证明
一个同样艰巨的挑战涉及从微生物组-人类特征关联到因果关系证明的进展。事实上,在建议与临床结果相关、相互关联和预测的许多微生物群中,只有少数基因产物和代谢物可以证明对生理和疾病状态有因果影响,而我们预计一部分微生物特征是先发于疾病病理改变的特征,而有一部分特征是继发于以疾病状态为特征的环境变化。
越来越多的工具正在开发中,通过揭示因果关系和机制洞察力,将前者“驱动”微生物组改变与后者“乘客”改变区分开来。
解决方案:无菌或使用抗生素的动物模型或其他改进的技术,测试单个细菌菌株或群落的影响
一种经常使用的方法依赖于微生物群耗尽(通过抗生素治疗)或微生物群缺乏(无菌)小鼠模型,这些模型允许引入单个细菌菌株或群落以测试它们对宿主生理学的影响。
抗生素杀菌模型局限性:多因素混杂,难以预测
抗生素是现代医学的基石,因为它们能够在感染期间抑制病原体的生长。抗生素的广泛特征是它们的抑制机制是抑菌(生长停止)或杀菌(致死),并且它们的活性范围是宽的或窄的。大多数抗生素的作用主要在物种单一培养中进行了研究,尽管临床环境中的治疗不可避免地会对人类肠道上的多物种群落产生意想不到的后果。抗生素会对肠道细菌造成附带损害,并降低肠道微生物群的多样性,由此可能出现艰难梭菌感染倾向增加和癌症治疗后死亡率增加。
抗生素扰动期间的群落动态很难预测,因为环境可以通过种间相互作用、pH 调节和代谢转化改变抗生素的作用。使用多种治疗方案,例如可变剂量、顺序安排多种化合物和同时使用鸡尾酒治疗使预测进一步复杂化。
同时多种药物可产生协同或拮抗作用, 由此杀死的程度分别大于或小于单独药物的总和,但是他们对肠道菌群的扰动和影响研究还甚少,检测中面临这些用药信息准确跟踪记录也较困难。而且抗生素导致的多重耐药细菌的定植也会导致抗生素耐药基因转移到微生物群中,最终会导致对各种有效抗生素的耐药性增加,并增加致命感染的风险。
其他新技术:改进的体内模型、类器官和片上器官平台
随着更多具有人类代表性的实验平台的出现,野鼠体内的微生物群与圈养外的家鼠相似,正迅速受到关注。这些改进的体内模型得到了合成人类共生群落的发展、以前“无法培养”微生物在菌株水平分辨率下的改进培养和表征,以及通过非靶向代谢组学和蛋白质组学对生物活性微生物效应因子或代谢物的无偏见阐明的补充。
类器官和片上器官平台能够研究人体组织环境中的单个细菌、细菌群落及其生物活性产物。
注:
类器官(organoid)是指由人体组织或干细胞等体外培养出的三维结构,能够模拟某些人体器官的结构和功能。类器官的制备通常是通过将干细胞或组织细胞在特定的培养条件下进行分化和自组装,形成类似于人体器官的结构。类器官的制备可以为疾病研究和药物筛选提供更真实、更可靠的模型。例如,肝脏类器官可以用于测试药物代谢和毒性,肠道类器官可以用于研究肠道微生物和肠道疾病,而大脑类器官可以用于研究神经退行性疾病等。
片上器官平台(organ-on-a-chip platform)是一种仿生学技术,它是通过微流控技术、微加工技术和3D打印技术等手段,将人体组织和器官的细胞培养在微型芯片上,模拟人体器官的生理、病理过程,以实现高通量、高精度的药物筛选、毒性测试和疾病研究。这种技术可以模拟人体器官的微环境,如细胞间相互作用、细胞信号传递、细胞分化、细胞迁移、细胞凋亡等生理和病理过程,从而更好地研究疾病的发生和发展,以及评价药物的安全性和有效性。
凭借这些新技术获得的能力,该领域正在揭示基因组测序之外的功能读数,解码难以培养和低生物量微生物组的贡献,并评估重要但研究不足的非细菌共生王国及其错综复杂的相互作用组网络。
事实上,对共生和机会病毒(包括噬菌体)、真菌和寄生虫的潜在功能及其对细菌共生生态系统和人类宿主的影响的不断探索,构成了正在推进的研究领域,当然肠道细菌还是最主要的。对这些特征不明确的共生微生物的研究需要进一步开发各种工具,包括改进的计算参考数据集、分子开发工具和体内定植模型。
03
展望微生物组研究的下一个时代:
从分子机制到诊断与治疗的广泛应用
通过对微生物分子和功能见解的不断扩展,可能会在微生物组研究的下一个时代开发广泛的诊断、预后和治疗人类应用。
治疗性微生物组使用的一个有前景的例子:
粪便微生物组移植 (FMT)
除了治疗常见的艰难梭菌感染之外,显示出对肝病、炎症和免疫相关疾病的积极改善效果
促进微生物组研究领域的蓬勃发展
失败的试验和风险强调了对机制的了解的重要性
为了避免人体微生物研究领域中早期技术失误:
- 需深入了解宿主-微生物组相互作用的复杂性和模块化
- 修改当前研究范式
为了辅助人类疾病的诊断和治疗需要:
- 持续、详尽地探索微生物组的分子机制和调控
- 从总体描述性群落关联到离散的生物活性治疗靶点的微观功能
- 关注共生微生物及其与人类结合对应物的影响,研究下游对人类宿主产生、调节和降解的化合物的生化和结构阐明。
作为向前迈出的积极一步,微生物组研究能够做到以下:
早期检测
识别和利用微生物组粘膜,免疫和全身信号,以便通过离散的疾病特征进行早期检测和患者分层。
治疗层面
“一刀切”的传统观念正在发生改变,人们不再简单地认为所有人的微生物组都可以通过相同的方式进行调节,而是转向将微生物组视为“信号中心”的观点,也就是说更加注重研究和借鉴微生物群落的信号传递机制,以更好地治疗相关疾病。
微生物组被视为特定环境的中继或缓冲器,整合环境和内源性信号,影响携带健康或疾病易感遗传特征的个体的生理和疾病表现。
一个备受关注的话题是“益生菌干预法”。证据表明益生菌可能有助于治疗和预防传染病,改善过敏,降低血糖,联合免疫治疗提高治疗效果。益生菌治疗会改变腔内微生物群,这种改变进一步会显着影响全身代谢,包括胰岛素抵抗,炎症,代谢等。
将微生物组作为一个高度模块化的“指纹”,有助于开发合理的、针对具体情况的和数据驱动的干预措施,例如个性化营养、精准益生菌、代谢物补充(“后生元”疗法)和定向病原体抑制模式。
这些方法的验证和整合,加上对大量微生物群及其相关生物活性产品对人类宿主的影响的持续探索,将有助于实现该领域改变人类健康的巨大前景。
参考文献
Puschhof J, Elinav E. Human microbiome research: Growing pains and future promises. PLoS Biol. 2023 Mar 17;21(3):e3002053.