GeSciLiVis | 想知道你感兴趣的基因有多少人在研究吗!?用这个包来解决吧!!!~
1写在前面
天气好热啊,我这里还下着大暴雨!~
不知道各位小伙伴那里的温度怎么样,端午临近,各位有假期吗!?
换组后工作轻松了不少,也有时间做点自己的事情了。
欢迎各位小伙伴后台提问,推荐想看的教程等等!~
今天分享一个极大提高效率的R包,GeSciLiVis包。☺️
毕竟工欲善其事,必先利其器,好的工具可以极大地提高效率,降低你的疲劳感,哈哈哈哈哈哈哈。
2用到的包
rm(list = ls())
library(tidyverse)
library(GeSciLiVis)
3函数展示
GeSciLiVis基于基因列表和用户定义的关键词,利用Pubmed检索来可视化每个基因的发文情况。
主要提供了三个函数:
1️⃣ human_official_gene_names, 可以查看目前截止2023年2月5日HGNC中收录的人的官方基因名。
2️⃣ mouse_official_gene_names, 可以查看目前截止2023年2月5日HGNC中收录的小鼠的官方基因名。
3️⃣ pub_activity_plot,可视化目的基因的发文情况。
pub_activity_plot(
g_list,
k_list,
species = "mouse",
output_dir = "output",
use_preloaded_gene_names=T,
show_progressbar = F
)
4函数一:human_official_gene_names
这个函数可以查看目前的人类官方基因名,建议大家从这里面用$提取去检索哦。
DT::datatable(human_official_gene_names)
5函数二:mouse_official_gene_name
同样的,如果你研究的是小鼠可以看这个数据集。
DT::datatable(mouse_official_gene_names)
6函数三:pub_activity_plot
6.1 示例一
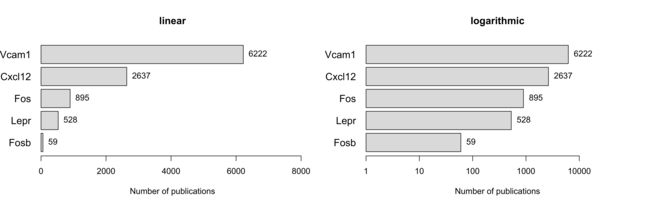
我们先建几个关键词,就三种细胞吧。
keyword_list <- c("mesenchymal stem cell", "Endothelial cell", "Adipocytes")
接着是一些我们感兴趣的基因。
gene_list_mouse <- c("Cxcl12", "Fos", "Lect1", "Lepr", "Vcam1", "Fosb")
pub_activity_plot(g_list = gene_list_mouse, k_list = keyword_list,
species = "mouse", output_dir="output",
use_preloaded_gene_names=T)
6.2 示例二
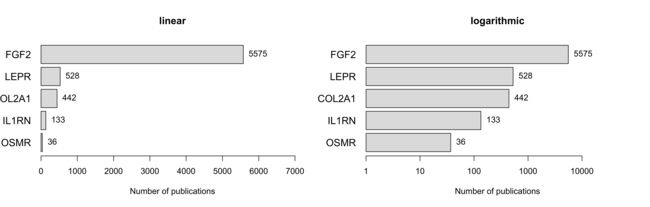
再观察一下这些基因在人类mesenchymal stem cell, Endothelial cell和Adipocytes中的发文情况吧。
gene_list_human = c("LEPR", "KITL", "OSMR", "IL1RN", "FGF2", "COL2A1")
pub_activity_plot(gene_list_human, keyword_list,
"human", output_dir="output",
use_preloaded_gene_names=T)
接着在你的工作目录下,会直接输出结果,连PMID都准备好了哦。
复制好PMID就用魔法棒来解决批量下载的问题吧。
冲冲冲,用起来吧,各位!~

点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰
chatPDF | 别再自己读文献了!让chatGPT来帮你读吧!~
WGCNA | 值得你深入学习的生信分析方法!~
ComplexHeatmap | 颜狗写的高颜值热图代码!
ComplexHeatmap | 你的热图注释还挤在一起看不清吗!?
Google | 谷歌翻译崩了我们怎么办!?(附完美解决方案)
scRNA-seq | 吐血整理的单细胞入门教程
NetworkD3 | 让我们一起画个动态的桑基图吧~
RColorBrewer | 再多的配色也能轻松搞定!~
rms | 批量完成你的线性回归
CMplot | 完美复刻Nature上的曼哈顿图
Network | 高颜值动态网络可视化工具
boxjitter | 完美复刻Nature上的高颜值统计图
linkET | 完美解决ggcor安装失败方案(附教程)
......
本文由 mdnice 多平台发布