Brian2学习笔记_Introduction to Brian part2:Synapses
本文根据Brian2官方英文教程进行翻译总结。
Brian2官方安装教程链接:
https://brian2.readthedocs.io/en/stable/introduction/install.html
使用教程Tutorial:
Introduction to Brian part 1 : Neurons 原文
Introduction to Brian part 2 : Synapses 原文
Introduction to Brian part 3 : Simulation 原文
运行的三种环境:Brian提供的基于网页的交互式模拟仿真器“Binder”;本地的Jupyter Notebook;Ipython或标准Python终端。
与之前一样,本文所有代码均在Jupyter上运行。从导入Brian包开始:
from brian2 import *
%matplotlib inline
prefs.codegen.target = "numpy"
第三行代码是由于我们未安装Microsoft Visual C++ 14.0,所以不能运行Cython。可以通过加入上述代码来避免这种情况,代价是运行速度变慢。
文章目录
- Introduction to Brian part2:Synapses
-
- 2.1 最简单的突触
- 2.2 加入权重
- 2.3 引入时滞
- 2.4 更复杂的连接
- 2.5 更复杂的突触模型:STDP
Introduction to Brian part2:Synapses
上一篇文章介绍了Brian的神经元(Neurons)。在生物体内,神经元是通过突触连接的。突触前神经元通过突触将含有信息的脉冲传递给突触后神经元。并且,不同类型的突触可以改变神经元的状态,这也使神经网络的理论基础。
2.1 最简单的突触
一旦你有了一些神经元,下一步就是通过突触将它们连接起来。我们将从最简单的突触类型开始,它会在峰值后引起变量的瞬时变化。
start_scope()
eqs = '''
dv/dt = (I-v)/tau : 1
I : 1
tau : second
'''
G = NeuronGroup(2, eqs, threshold='v>1', reset='v = 0', method='exact')
G.I = [2, 0]
G.tau = [10, 100]*ms
# Comment these two lines out to see what happens without Synapses
S = Synapses(G, G, on_pre='v_post += 0.2')
S.connect(i=0, j=1)
M = StateMonitor(G, 'v', record=True)
run(100*ms)
plot(M.t/ms, M.v[0], label='Neuron 0')
plot(M.t/ms, M.v[1], label='Neuron 1')
xlabel('Time (ms)')
ylabel('v')
legend()
运行结果如下:

这里需要说明以下几点。
首先,让我们回顾一下NeuronGroup的情况。我们创建了两个神经元,每个神经元都有相同的微分方程但变量 I I I 和 t a u tau tau 的值不同。Neuron 0的变量 I = 2 , t a u = 10 ∗ m s I=2,tau=10*ms I=2,tau=10∗ms,这意味着它会以较高的速率反复发射脉冲。Neuron 1的变量 I = 0 , t a u = 100 ∗ m s I=0,tau=100*ms I=0,tau=100∗ms,这意味着它如果没有突触,就根本不会发射脉冲(刺激电流 I I I 为 0 0 0)。你可以通过注释掉定义突触的两行代码来证明这一点。
接下来我们定义突触:Synapses(source, target, …) 意味着我们要定义一个从源(source)到目标(target)的突触模型。在本例中,源和目标是相同的,都是G。语法 o n _ p r e = ′ v _ p o s t + = 0. 2 ′ on\_pre='v\_post \,+\! = 0.2' on_pre=′v_post+=0.2′ 意味着当突触前神经元出现脉冲时,它会立刻使突触后神经元发生瞬时变化 v _ p o s t + = 0.2 v\_post \,+\! = 0.2 v_post+=0.2。_post表示引用的 v v v 值是突触后神经元膜电位的值,增加0.2。
所以总的来说,这个模型的作用是: G G G 中的两个神经元通过突触连接时,源神经元发出一个脉冲,目标神经元的 v v v 值就会增加 0.2 0.2 0.2。
然而,我们只定义了突触模型,还没有真正创建任何突触。下一行 S . c o n n e c t ( i = 0 , j = 1 ) S.connect(i=0, j=1) S.connect(i=0,j=1) 创建了一个从Neuron 0到Neuron 1的突触。
2.2 加入权重
前文中,我们将权重固定为0.2,但通常我们希望不同的突触有不同的权重,可以通过引入突触方程来实现:
start_scope()
eqs = '''
dv/dt = (I-v)/tau : 1
I : 1
tau : second
'''
G = NeuronGroup(3, eqs, threshold='v>1', reset='v = 0', method='exact')
G.I = [2, 0, 0]
G.tau = [10, 100, 100]*ms
# Comment these two lines out to see what happens without Synapses
S = Synapses(G, G, 'w : 1', on_pre='v_post += w')
S.connect(i=0, j=[1, 2])
S.w = 'j*0.2'
M = StateMonitor(G, 'v', record=True)
run(50*ms)
plot(M.t/ms, M.v[0], label='Neuron 0')
plot(M.t/ms, M.v[1], label='Neuron 1')
plot(M.t/ms, M.v[2], label='Neuron 2')
xlabel('Time (ms)')
ylabel('v')
legend()
运行结果如下:

这个例子与上一个非常相似,但多了一个突触权重变量 w w w。字符串 ′ w : 1 ′ 'w:1' ′w:1′ 也是一个方程,定义了一个无量纲参数 w w w。我们现在将脉冲行为改变为 o n _ p r e = ′ v _ p o s t + = w ′ on\_pre='v\_post \,+\!= w' on_pre=′v_post+=w′,这样每个突触的行为就会根据 w w w 的值而不同。为了说明这一点,我们创建了第三个神经元,它的行为与第二个神经元完全相同,并将Neuron 0分别连接到Neuron 1和2。我们还通过 S . w = ′ j ∗ 0. 2 ′ S.w = 'j*0.2' S.w=′j∗0.2′ 设置了权重。其中, i i i表示源神经元索引, j j j表示目标神经元索引。所以这将产生一个从0到1的突触连接,其权重为 0.2 = 0.2 ∗ 1 0.2=0.2*1 0.2=0.2∗1,从0到2的突触连接,其权重为 0.4 = 0.2 ∗ 2 0.4=0.2*2 0.4=0.2∗2。
2.3 引入时滞
到目前为止,突触都是即时发生,可以加入一些时滞的延迟:
start_scope()
eqs = '''
dv/dt = (I-v)/tau : 1
I : 1
tau : second
'''
G = NeuronGroup(3, eqs, threshold='v>1', reset='v = 0', method='exact')
G.I = [2, 0, 0]
G.tau = [10, 100, 100]*ms
S = Synapses(G, G, 'w : 1', on_pre='v_post += w')
S.connect(i=0, j=[1, 2])
S.w = 'j*0.2'
S.delay = 'j*2*ms'
M = StateMonitor(G, 'v', record=True)
run(50*ms)
plot(M.t/ms, M.v[0], label='Neuron 0')
plot(M.t/ms, M.v[1], label='Neuron 1')
plot(M.t/ms, M.v[2], label='Neuron 2')
xlabel('Time (ms)')
ylabel('v')
legend()
运行结果如下:

观察上图,我们通过添加一行代码 S . d e l a y = ′ j ∗ 2 ∗ m s ′ S.delay='j*2*ms' S.delay=′j∗2∗ms′ 使得突触从0到1(j=1)有2ms的延迟,从0到2(j=2)有4ms的延迟。
2.4 更复杂的连接
到目前为止,我们都明确地指定了突的触连接。但对于较大的网络,这通常不可能。为此,我们通常需要指定一些条件。
start_scope()
N = 10
G = NeuronGroup(N, 'v:1')
S = Synapses(G, G)
S.connect(condition='i!=j', p=0.2)
在这里,我们创建了一个含有 N N N 个仿真神经元和一个仿真突触的模型。实际上它什么也不做,只是用来展示这种连接。
代码 S . c o n n e c t ( c o n d i t i o n = ′ i ! = j ′ , p = 0.2 ) S.connect(condition='i!=j', p=0.2) S.connect(condition=′i!=j′,p=0.2) 表示只有当 i ! = j i!=j i!=j 满足时,将有0.2的概率连接神经元 i i i 和 j j j。那么,我们如何才能看到这种连接关系呢?这里有一个小函数可以让我们把它可视化。
def visualise_connectivity(S):
Ns = len(S.source)
Nt = len(S.target)
figure(figsize=(10, 4))
subplot(121)
plot(zeros(Ns), arange(Ns), 'ok', ms=10)
plot(ones(Nt), arange(Nt), 'ok', ms=10)
for i, j in zip(S.i, S.j):
plot([0, 1], [i, j], '-k')
xticks([0, 1], ['Source', 'Target'])
ylabel('Neuron index')
xlim(-0.1, 1.1)
ylim(-1, max(Ns, Nt))
subplot(122)
plot(S.i, S.j, 'ok')
xlim(-1, Ns)
ylim(-1, Nt)
xlabel('Source neuron index')
ylabel('Target neuron index')
visualise_connectivity(S)
结果如下:
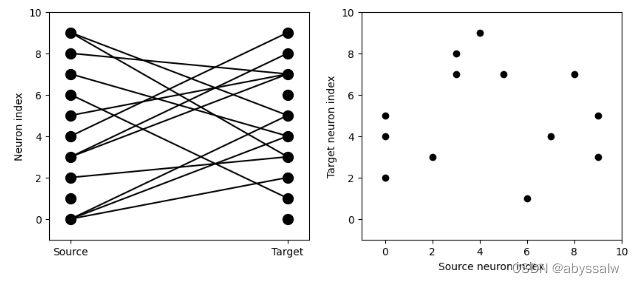
左图左边垂直排列着10个源神经元,右边垂直排列着10个目标神经元,两个神经元之间的线代表它们之间存在一个突触。右图是另一种形象化的表示方法。图中的每个黑点都是一个突触,x值是源神经元索引,y值是目标神经元索引。让我们看看当我们改变连接的概率时,这些数字是如何变化的:
start_scope()
N = 10
G = NeuronGroup(N, 'v:1')
for p in [0.1, 0.5, 1.0]:
S = Synapses(G, G)
S.connect(condition='i!=j', p=p)
visualise_connectivity(S)
suptitle('p = '+str(p))
结果如下:
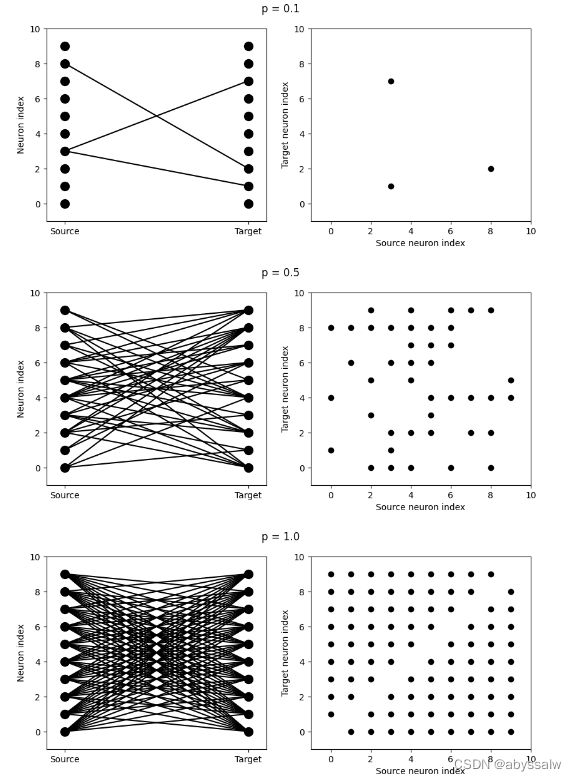
接下来,我们来观察改变连接条件会发生什么?如让它们只与相邻的神经元相连:
start_scope()
N = 10
G = NeuronGroup(N, 'v:1')
S = Synapses(G, G)
S.connect(condition='abs(i-j)<4 and i!=j')
visualise_connectivity(S)
结果如下:

尝试使用该单元格来查看其他连接条件的情况。您还可以使用生成器语法更有效地创建这样的连接。在像上面那样的小例子中,这无关紧要,但对于大量的神经元构成的网络,直接指定哪些神经元应该连接比仅仅指定一个条件要有效得多。注意,下面的例子使用skip_if_invalid来避免边界错误(例如,不要尝试将索引为1的神经元连接到索引为-2的神经元)。
start_scope()
N = 10
G = NeuronGroup(N, 'v:1')
S = Synapses(G, G)
S.connect(j='k for k in range(i-3, i+4) if i!=k', skip_if_invalid=True)
visualise_connectivity(S)
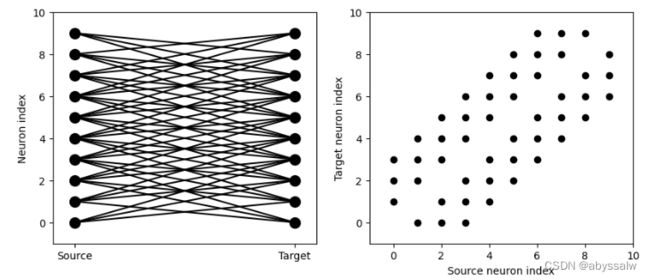
如果每个源神经元精确地连接到一个目标神经元(通常用于两个大小相同的独立组,而不是像本例中完全相同的源和目标组),则存在一种非常有效的特殊语法。例如,1对1连接是这样的:
start_scope()
N = 10
G = NeuronGroup(N, 'v:1')
S = Synapses(G, G)
S.connect(j='i')
visualise_connectivity(S)
结果如下:
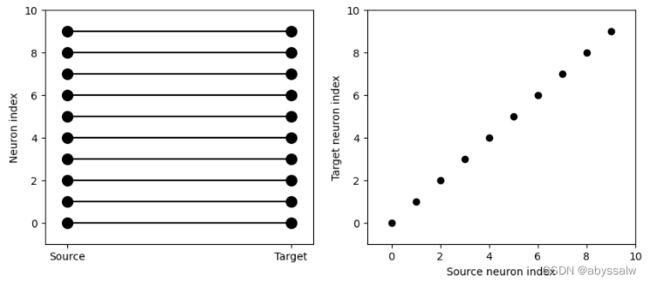
你还可以使用字符串指定权重值。让我们看一个例子,我们给每个神经元分配一个空间位置,并有一个依赖于距离的连接函数。我们通过标记的大小来可视化突触的权重:
start_scope()
N = 30
neuron_spacing = 50*umetre
width = N/4.0*neuron_spacing
# Neuron has one variable x, its position
G = NeuronGroup(N, 'x : metre')
G.x = 'i*neuron_spacing'
# All synapses are connected (excluding self-connections)
S = Synapses(G, G, 'w : 1')
S.connect(condition='i!=j')
# Weight varies with distance
S.w = 'exp(-(x_pre-x_post)**2/(2*width**2))'
scatter(S.x_pre/um, S.x_post/um, S.w*20)
xlabel('Source neuron position (um)')
ylabel('Target neuron position (um)')
2.5 更复杂的突触模型:STDP
Brian的突触框架非常通用,可以实现短期可塑性(STP)或脉冲时间依赖可塑性(spike-timing dependent plasticity, STDP)。STDP通常由如下等式定义: Δ w = ∑ t p r e ∑ t p o s t W ( t p o s t − t p r e ) \Delta w=\sum_{tpre} \sum_{tpost} W(t_{post}-t_{pre}) Δw=tpre∑tpost∑W(tpost−tpre)
也就是说,突触权重 w w w 的变化是所有突触前脉冲发射时间 t p r e t_{pre} tpre 和突触后脉冲发射时间 t p o s t t_{post} tpost 之差的某个 w w w 函数的总和。一个常用的 w w w 函数是:KaTeX parse error: Expected & or \\ or \cr or \end at end of input: …ases} A_{pre} e
W ( Δ t ) = { A p r e e − Δ t / τ p r e , Δ t > 0 A p o s t e Δ t / τ p o s t , Δ t < 0 W(\Delta t)=\begin{cases} {A_{pre}e^{-\Delta t/ \tau_{pre}}}, & \Delta t>0\\ {A_{post}e^{\Delta t/ \tau_{post}}}, & \Delta t<0 \end{cases} W(Δt)={Apree−Δt/τpre,AposteΔt/τpost,Δt>0Δt<0
代码实现如下:
tau_pre = tau_post = 20*ms
A_pre = 0.01
A_post = -A_pre*1.05
delta_t = linspace(-50, 50, 100)*ms
W = where(delta_t>0, A_pre*exp(-delta_t/tau_pre), A_post*exp(delta_t/tau_post))
plot(delta_t/ms, W)
xlabel(r'$\Delta t$ (ms)')
ylabel('W')
axhline(0, ls='-', c='k')
运行结果:

直接用上述方程来模拟是非常低效的,因为我们必须对所有成对的脉冲求和。这在生理上也是不现实的,因为神经元无法记住之前所有的脉冲发射时间。事实证明,有一种更有效、生理上更合理的方法可以达到同样的效果。
我们定义了两个新的变量,它们是突触前和突触后活动的“痕迹”(trace),由微分方程控制: τ p r e d d t a p r e = − a p r e \tau_{pre} \frac {d}{dt}a_{pre}=-a_{pre} τpredtdapre=−apre τ p o s t d d t a p o s t = − a p o s t \tau_{post} \frac {d}{dt}a_{post}=-a_{post} τpostdtdapost=−apost 当一个突触前脉冲出现时,突触前痕迹与权重将会被更新。 a p r e → a p r e + A p r e a_{pre} \rightarrow a_{pre} + A_{pre} apre→apre+Apre w → w + a p r e w \rightarrow w+a_{pre} w→w+apre 当一个突触后脉冲出现时: a p o s t → a p o s t + A p o s t a_{post} \rightarrow a_{post} + A_{post} apost→apost+Apost w → w + a p r e w \rightarrow w+a_{pre} w→w+apre 为了证明这个公式是等价的,你只需要检查方程的和是否是线性的,并考虑两种情况:如果突触前脉冲发生在突触后脉冲之前发射会发生什么?反之亦然。试着把它画出来。
现在我们有了一个只依赖于微分方程和脉冲事件的公式,我们可以把它变成Brian代码。
start_scope()
taupre = taupost = 20*ms
wmax = 0.01
Apre = 0.01
Apost = -Apre*taupre/taupost*1.05
G = NeuronGroup(1, 'v:1', threshold='v>1', reset='')
S = Synapses(G, G,
'''
w : 1
dapre/dt = -apre/taupre : 1 (event-driven)
dapost/dt = -apost/taupost : 1 (event-driven)
''',
on_pre='''
v_post += w
apre += Apre
w = clip(w+apost, 0, wmax)
''',
on_post='''
apost += Apost
w = clip(w+apre, 0, wmax)
''')
这里有一些注意事项:首先,在定义突触时,我们使用复杂的多行字符串,定义了三个突触变量(w, apre和apost)。在apre和apost的定义之后,我们还增加了一个新的语法(事件驱动)。这意味着,尽管这两个变量随着时间不断变化,但Brian应该只在事件(脉冲)发生时更新它们。这是因为我们不需要脉冲生成时刻之外的apre和apost的值,并且只在需要时更新它们,这样效率才会更高。
接下来是参数 on_pre=…。
第一行代码 v _ p o s t + = w v\_post \,+\!= w v_post+=w:将突触权重施加给目标神经元。
第二行代码 a p r e + = A p r e apre \,+\!= Apre apre+=Apre,对上面的规则进行编码。
第三行代码,也对上面的规则进行编码,但我们增加了一个额外的特征:我们将突触权重值限制在最小值 0 0 0 和最大值 w m a x wmax wmax 之间,这样权重就不会太大或为负。选择函数clip(x, low, high)实现此功能。
最后,我们有一个参数 on_post=…。它提供了计算突触后神经元放电的语句。注意,在这种情况下,我们不修改 v v v,只修改突触变量。现在让我们来看看当突触前脉冲在突触后脉冲之前到达时所有的变量是如何表现的。
start_scope()
taupre = taupost = 20*ms
wmax = 0.01
Apre = 0.01
Apost = -Apre*taupre/taupost*1.05
G = NeuronGroup(2, 'v:1', threshold='t>(1+i)*10*ms', refractory=100*ms)
S = Synapses(G, G,
'''
w : 1
dapre/dt = -apre/taupre : 1 (clock-driven)
dapost/dt = -apost/taupost : 1 (clock-driven)
''',
on_pre='''
v_post += w
apre += Apre
w = clip(w+apost, 0, wmax)
''',
on_post='''
apost += Apost
w = clip(w+apre, 0, wmax)
''', method='linear')
S.connect(i=0, j=1)
M = StateMonitor(S, ['w', 'apre', 'apost'], record=True)
run(30*ms)
figure(figsize=(4, 8))
subplot(121)
plot(M.t/ms, M.apre[0], label='apre')
plot(M.t/ms, M.apost[0], label='apost')
legend()
subplot(122)
plot(M.t/ms, M.w[0], label='w')
legend(loc='best')
xlabel('Time (ms)')

有几点要注意:
首先,我们使用了一个小技巧,让神经元0在10ms时发放一个脉冲,而让神经元1在20ms时发放一个脉冲。你能看出它们是如何工作的吗?
其次,我们已经用clock_driven替换了event_driven,这样你就可以看到apre和apost是如何随着时间变化的。试着改变脉冲发放时间,看看会发生什么。
最后,我们验证一下这个公式是否等价于原来的公式。
start_scope()
taupre = taupost = 20*ms
Apre = 0.01
Apost = -Apre*taupre/taupost*1.05
tmax = 50*ms
N = 100
# Presynaptic neurons G spike at times from 0 to tmax
# Postsynaptic neurons G spike at times from tmax to 0
# So difference in spike times will vary from -tmax to +tmax
G = NeuronGroup(N, 'tspike:second', threshold='t>tspike', refractory=100*ms)
H = NeuronGroup(N, 'tspike:second', threshold='t>tspike', refractory=100*ms)
G.tspike = 'i*tmax/(N-1)'
H.tspike = '(N-1-i)*tmax/(N-1)'
S = Synapses(G, H,
'''
w : 1
dapre/dt = -apre/taupre : 1 (event-driven)
dapost/dt = -apost/taupost : 1 (event-driven)
''',
on_pre='''
apre += Apre
w = w+apost
''',
on_post='''
apost += Apost
w = w+apre
''')
S.connect(j='i')
run(tmax+1*ms)
plot((H.tspike-G.tspike)/ms, S.w)
xlabel(r'$\Delta t$ (ms)')
ylabel(r'$\Delta w$')
axhline(0, ls='-', c='k')

