全文链接:https://tecdat.cn/?p=33449
原文出处:拓端数据部落公众号
当面对多个模型时,我们有多种选择。模型选择因其简单性而具有吸引力,但我们正在丢弃有关模型中不确定性的信息。
print(f"Runing 模型平均
一种替代方法是执行模型选择,但讨论所有不同的模型以及给定信息准则的计算值。重要的是要将所有这些数字和测试放在我们问题的背景下,以便我们和我们的客户能够更好地了解我们方法可能存在的局限性和缺点。如果你在学术界,你可以使用这种方法向论文、演示文稿、论文等的讨论部分添加元素。
另一种方法是执行模型平均。现在的想法是使用模型的加权平均值生成元模型(和元预测)。有几种方法可以做到这一点,PyMC3 包括其中的 3 种,我们将简要讨论,您将在 Yuling Yao 等人的工作中找到更彻底的解释。
伪贝叶斯模型平均
贝叶斯模型可以通过其边缘概率进行加权,这被称为贝叶斯模型平均。我们可以使用以下公式来做到这一点:
这种方法称为伪贝叶斯模型平均或类似赤池的加权,是一种启发式方法,用于根据信息标准值计算每个模型(给定一组固定的模型)的相对概率。看看分母只是一个归一化项,以确保权重总和为 1。
使用贝叶斯自举进行伪贝叶斯模型平均
上述计算权重的公式是一种非常好且简单的方法,但有一个主要警告,它没有考虑 IC 计算中的不确定性。
堆叠
在PyMC3中实现的第三种方法被称为预测分布的堆叠,并且最近被提出。我们希望在一个元模型中组合多个模型,以最小化元模型和真实生成模型之间的分歧,当使用对数评分规则时,这相当于:
加权后验预测样本
一旦我们计算了权重,使用上述 3 种方法中的任何一种,我们就可以使用它们来获得加权后验预测样本。PyMC3 提供了以简单方式执行这些步骤的函数,因此让我们通过示例查看它们的实际效果。
简而言之,我们的问题如下:我们想探索几种灵长类动物的乳汁成分,假设来自大脑较大的灵长类动物的雌性产生更有营养的牛奶(这样做是为了*支持这种大大脑的发育)。对于进化生物学家来说,这是一个重要的问题,为了给出和回答,我们将使用3个变量,两个预测变量:新皮层的比例与总质量的比较 大脑和母亲体重的对数。对于预测变量,每克牛奶的千卡。使用这些变量,我们将构建 3 个不同的线性模型:
- 仅使用新皮层变量的模型
- 仅使用质量变量对数的模型
- 使用两个变量的模型
d.iloc[:, 1:] = d.iloc[:, 1:] - d.iloc[:, 1:].mean()
d.head()现在我们有了数据,我们将仅使用 neocortex。
with pm.Model() as model_0:
trace_0 = pm.sample(2000, return_inferencedata=True)第二个模型与第一个模型完全相同,只是我们现在使用质量的对数
with pm.Model() as model_1:
trace_1 = pm.sample(2000, return_inferencedata=True)最后是第三个模型使用 neocortex和 变量log_mass
with pm.Model() as model_2:
trace_2 = pm.sample(2000, return_inferencedata=True)现在我们已经对 3 个模型的后验进行了采样,我们将对它们进行视觉比较。一种选择是使用forestplot支持绘制多个迹线的函数。
az.plot_fo
另一种选择是在同一图中绘制多条迹线是使用densityplot 。
az.plot_d
现在我们已经对 3 个模型的后验进行了采样,我们将使用 WAIC(广泛适用的信息标准)来比较 3 个模型。我们可以使用 PyMC3 附带的compare功能来做到这一点。
comp = az.compare(model_dict)
comp我们可以看到最好的模型是,具有两个预测变量的模型。请注意,数据帧按从最低到最高 WAIC 的顺序(即从好到最差的模型)。
现在,我们将使用copmuted来生成预测,而不是基于单个模型,而是基于加权模型集。
ppc_w = pm.sample_posterior_predictive_w(
请注意,我们正在传递按其索引排序的权重。
我们还将计算最低 WAIC 模型的 PPC
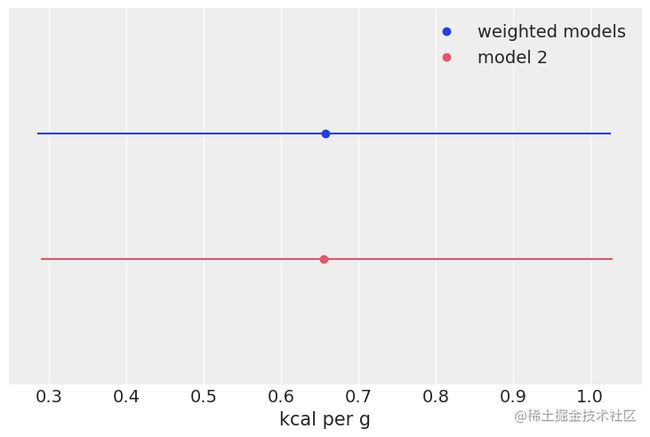
ppc_2 = pm.sample_posterior_predi比较这两种预测的一种简单方法是绘制它们的平均值和 hpd 区间
plt.yticks([])
plt.ylim(-1, 2)
plt.legend();正如我们所看到的,两个预测的平均值几乎相同,但加权模型中的不确定性更大。我们已经有效地将我们应该选择哪个模型的不确定性传递到后验预测样本中。
结语:
还有其他方法可以平均模型,例如,显式构建一个包含我们拥有的所有模型的元模型。然后,我们在模型之间跳转时执行参数推理。这种方法的一个问题是,在模型之间跳跃可能会妨碍后验的正确采样。
版本信息
%load_ext watermark
%watermark -n -u -v -iv -w最受欢迎的见解
4.R语言中的block Gibbs吉布斯采样贝叶斯多元线性回归
6.R语言贝叶斯Poisson泊松-正态分布模型分析职业足球比赛进球数