易基因:m6A-seq等揭示RBM33参与调控m6A去甲基化酶ALKBH5活性及其底物选择性|科研进展
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
RNA结合蛋白(RNA-binding protein,RBP)是一类结构和功能多样化的蛋白质,参与多种生物过程。越来越多的证据表明,RBP通过调控编码或非编码RNA的可变剪接、转运、稳定性、降解和翻译,在基因表达的转录后调控中发挥着关键作用,但RBP中RNA结合motif蛋白33(RNA-binding motif protein 33, RBM33)的生物学功能在很大程度上未知。N6-甲基腺苷(m6A)甲基化是真核生物mRNA中最丰富的内部修饰,是一个动态可逆的过程, m6A由m6A甲基转移酶复合体(MTC)沉积。尽管MTC可以被各种细胞组分招募到靶转录本,但目前还不清楚如何实现去甲基化选择性,是否存在类似的接头蛋白来促进所选转录本的m6A去甲基化呢?
2023年5月30日,美国芝加哥大学何川教授团队和美国佛罗里达大学钱志坚教授团队合作在《Molecular Cell》杂志上发表题为“RBM33 is a unique m6A RNA binding protein that regulates ALKBH5 demethylase activity and substrate selectivity”的研究论文,该研究通过MeRIP-seq、RIP-seq、RNA-seq等实验分析方法揭示了在头颈鳞状细胞癌 (head-neck squamous cell carcinoma,HNSCC) 中,RNA 结合蛋白RBM33参与调控m6A去甲基化酶ALKBH5活性及其底物选择性,并进一步验证了靶向ALKBH5/RBM33复合体m6A 去甲基化酶活性作为HNSCC 治疗策略的潜在可能。
标题:RBM33 is a unique m6A RNA binding protein that regulates ALKBH5 demethylase activity and substrate selectivity(RBM33是一种特异性m6A RNA结合蛋白,可以调控ALKBH5去甲基化酶活性和底物选择性)
时间:2023-05-30
期刊:Molecular Cell
影响因子:IF 16
技术平台:PAR-CLIP-seq 、m6A-seq(MeRIP-seq)、RNA-seq、qRT-PCR、WB等
研究摘要:
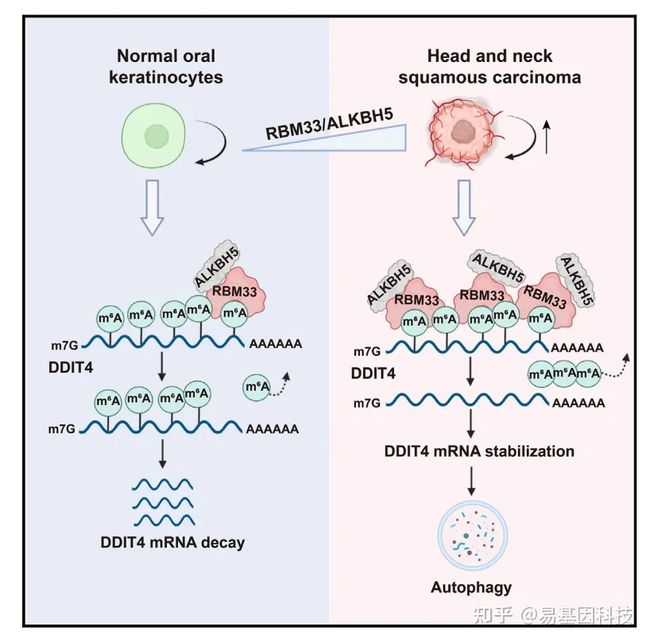
本研究鉴定出一种以前未被识别的m6A结合蛋白——RBM33,RBM33通过与ALKBH5结合形成复合体,在ALKBH5介导的mRNA m6A去甲基化转录本亚群中发挥关键作用。RBM33将ALKBH5招募到其m6A标记的底物上,并通过去除其SUMO化来激活ALKBH5去甲基化酶活性。研究进一步证明RBM33对头颈部鳞状细胞癌(HNSCC)的肿瘤发生至关重要。RBM33通过招募ALKBH5去甲基化和稳定DDIT4 mRNA来促进自噬,进而促进RBM33在HNSCC细胞中的致癌功能。总之,本研究揭示了在肿瘤发生过程中m6A转录本亚群选择性去甲基化的分子机制,这可能解释了其他细胞过程中去甲基化的选择性,并表明了它在维持HNSCC肿瘤发生中的重要性。
图形摘要
研究设计
研究结果:
(1)RBM33与ALKBH5形成复合体,调控依赖ALKBH5的mRNA m6A甲基化
- LC-MS分析发现ALKBH5是UM-SCC-1细胞中与RBM33相互作用的蛋白质之一;
- 在UM-SCC-1细胞中,内源性ALKBH5可以和内源性RBM33发生相互作用;
- 免疫染色分析显示,RBM33在细胞中与ALKBH5共定位。
- RBM33不与strep标记的METTL3、METTL14和FTO相互作用;
- 与ALKBH5过表达诱导的m6A去甲基化类似,RBM33过表达导致mRNA m6 A甲基化显著降低;
- ALKBH5过表达引起的细胞mRNA m6A下调可以被敲低RBM33所逆转;
- 同样敲除ALKBH5 可以阻止RBM33过表达所介导的m6A下调。
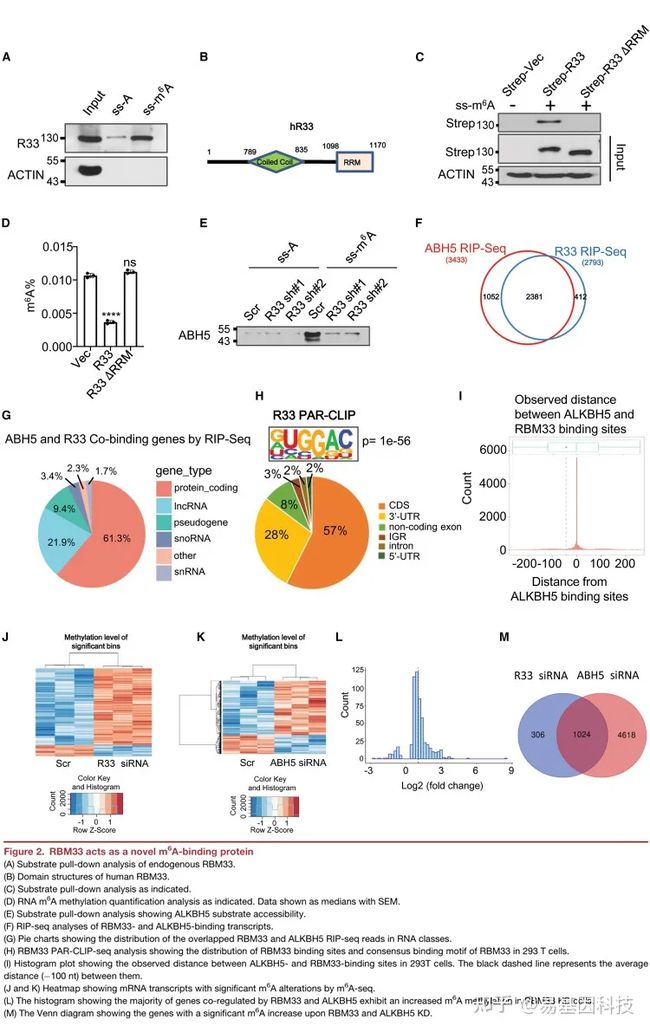
(2)RBM33是新的m6A结合蛋白,通过RRM结合m6A RNA转录本,并招募ALKBH5将其去甲基化
- 内源性RBM33与m6A修饰的RNA底物结合亲和力比与未修饰的RNA寡核苷酸的结合亲和力高;
- 去除RBM33的RRM结构域去除后丧失其与m6A修饰的RNA寡核苷酸结合的能力;
- RIP-Seq显示近70%的ALKBH5靶基因与RBM33结合。
- 绝大多数RBM33和ALKBH5的共同结合位点位于蛋白质编码转录本和LncRNAs中;
- PAR-CLIP揭示m6A修饰经典序列“RACH”出现在RBM33结合位点;
- 超过50%的RBM33结合位点位于ALKBH5结合位点上游100 nt处;
- RBM33 KD导致1330个RNA转录本的m6A修饰上调,其中65%的转录本的m6A修饰同样可以被ALKBH5 敲低所诱导。
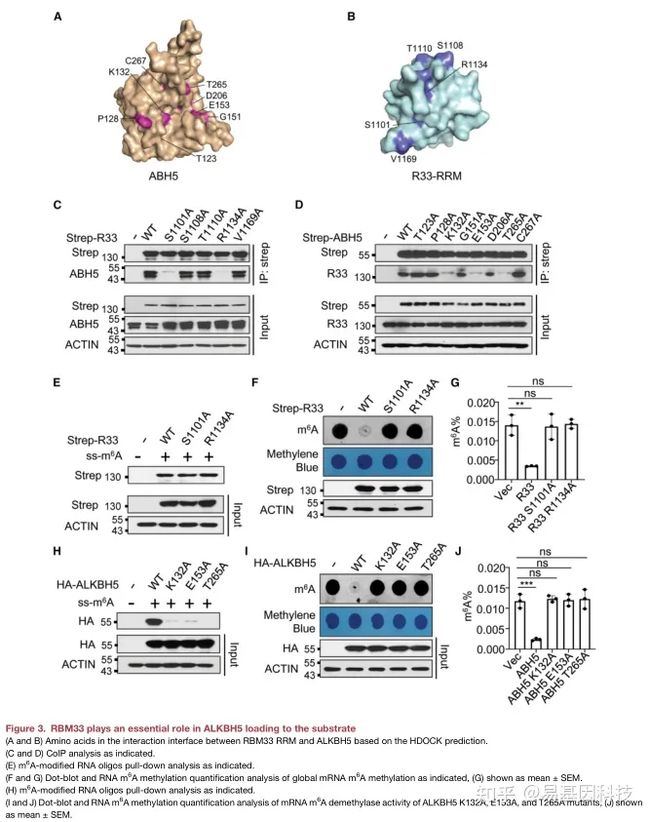
(3)ALKBH5通过RBM33结合m6A修饰底物以调节HNSCC细胞中m6A修饰
- RBM33蛋白中的S1101A或 R1134A显著破坏了ALKBH5与RBM33之间的相互作用
- ALKBH5中的K132A、E153A或T265A,显著破坏了ALKBH5与RBM33之间的相互作用。
- RBM33的S1101A和R1134A突变体与野生型RBM33对m6A修饰底物的结合亲和力相似;
- RBM33S1101A和RBM33R1134A突变体都失去了调控mRNA m6A甲基化的能力;
- ALKBH5的K132A、E153A和T265A突变体丧失与m6A修饰底物的结合能力;
- ALKBH5的K132A、E153A和T265A突变体丧失m6A去甲基化酶活性。
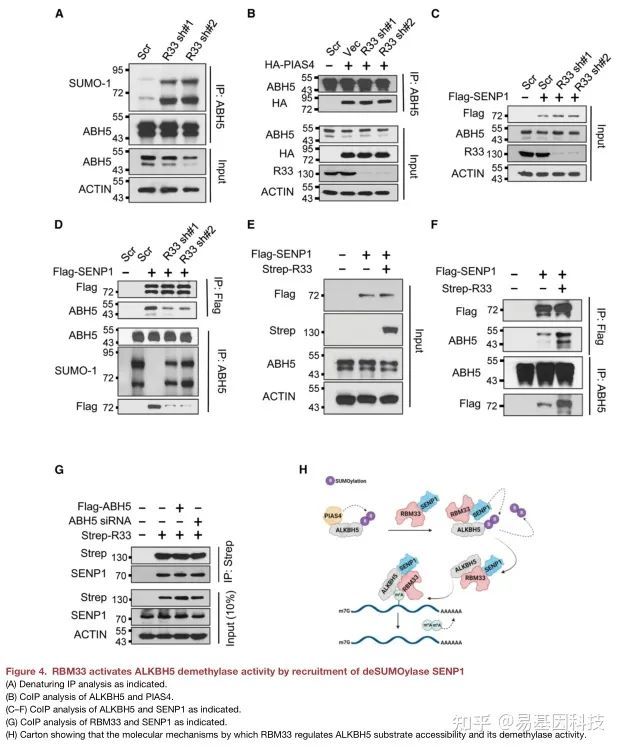
(4)RBM33通过募集deSUMOylase SENP1激活ALKBH5去甲基化酶活性
- RBM33 KD显著诱导ALKBH5 SUMOylation;
- RBM33 KD不影响ALKBH5和PIAS4相互作用,但破坏了ALKBH5和SENP1的相互作用;
- RBM33过表达增强了ALKBH5与SENP1的相互作用;
- RBM33与SENP1互作,而SENP1不依赖于ALKBH5。
(5)RBM33通常在HNSCC中上调,并且是肿瘤发生所必需的
- 免疫组化显示,RBM33在不同阶段HNSCC患者原发肿瘤中的表达高表达;
- RBM33 KD特异性诱导HNSCC细胞凋亡并抑制其克隆形成;
- RBM33 KD抑制HNSCC细胞克隆形成;
- RBM33 KD显著抑制异种移植小鼠体内HNSCC的生长。
(6)RBM33和ALKBH5相互依赖以维持HNSCC的致瘤性
- ALKBH5在HNSCC原发肿瘤中的表达远高于正常邻近组织;
- ALKBH5 KD可选择性诱导HNSCC细胞凋亡;
- ALKBH5 KD显著抑制HNSCC细胞的集落形成能力;
- 在UMSCC-1细胞中,ALKBH5过表达诱导的细胞增殖增加被RBM33 KD阻断,反之亦然;
- ALKBH5 KD对异种移植小鼠HNSCC体内细胞生长的抑制作用略高于RBM33 KD;
- ALKBH5小分子抑制剂2,4- PDCA显著抑制了小鼠中人HNSCC患者来源的异种移植物(PDX)的生长
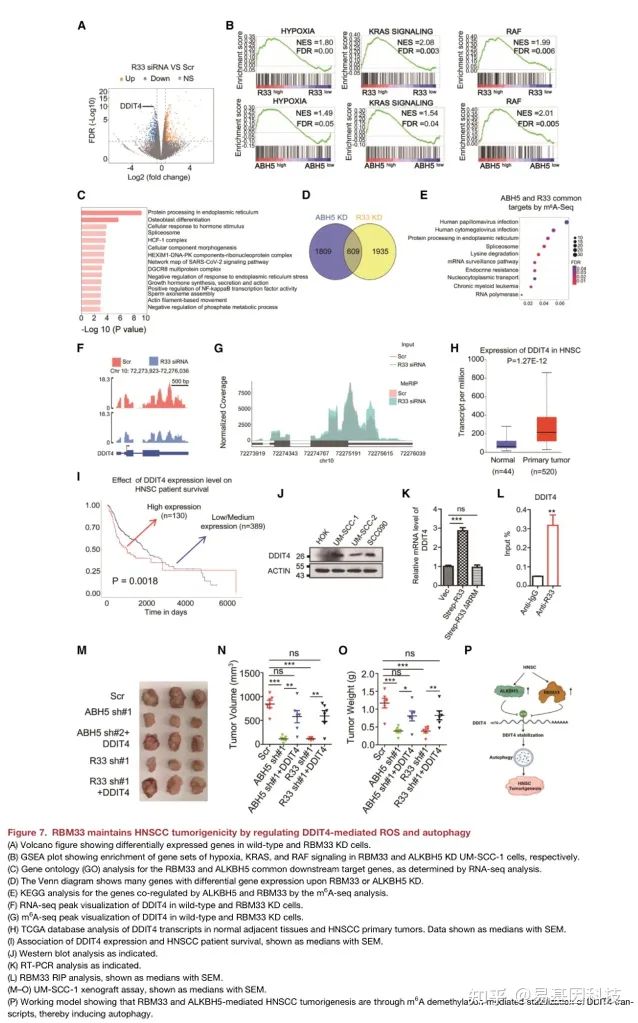
(7)DDIT4在HNSCC中是RBM33关键的下游靶点
- 基因集富集分析(GSEA)分析显示,RBM33或ALKBH5 KD上的下调基因在缺氧、RAS和RAF通路中富集;
- GO分析表明,RBM33和ALKBH5共同调控的基因在内质网中蛋白质剪切途径中富集;
- RBM33或ALKBH5 KD上m6A甲基化上调的转录本也参与内质网中蛋白质加工和剪接体途径。
- DDIT4在RBM33或ALKBH5 KD上m6A甲基化修饰显著增加;
- DDIT4的高表达与HNSCC患者的不良预后相关
- DDIT4的转录水平被野生型而不是△RRM的RBM33或ALKBH5突变体促进;
- DDIT4促进自噬,RBM33或ALKBH5 KD均能显著抑制自噬。
- 过表达DDIT4解除RBM33或ALKBH5 KD介导的异种移植小鼠HNSCC生长抑制。
研究结论:
RBM33是一种新的m6A结合蛋白,可与ALKBH5形成复合物。ALKBH5/RBM33 通过特异性去除DDIT4 转录本上的m6A修饰、促进DDIT4 RNA 稳定性,促进细胞自噬,进而促进HNSCC 的生长。
关于易基因RNA m6A甲基化测序(MeRIP-seq)技术
易基因MeRIP-seq技术利用m6A特异性抗体富集发生m6A修饰的RNA片段(包括mRNA、lncRNA等rRNA去除所有RNA),结合高通量测序,可以对RNA上的m6A修饰进行定位与定量,总RNA起始量可降低至10μg,最低仅需1μg总RNA。广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症发生与发展、药物应答等研究领域;可应用于动物、植物、细胞及组织的m6A检测。
大样本量m6A-QTL性状关联分析,传统MeRIP单个样品价格高,通常难以承担。易基因开发建立MeRIP-seq2技术,显著提成IP平行性,实现不同样本间相对定量,降低检测成本。
易基因提供适用于不同科研需求的MeRIP技术:
- m6A甲基化-常量mRNA 甲基化测序(MeRIP-seq)
- m6A甲基化-常量mRNA +lncRNA甲基化测序(lnc-MeRIP-seq)
- m6A甲基化-微量mRNA +lncRNA甲基化测序(Micro-lnc-MeRIP-seq)
- 高通量m6A甲基化-常量mRNA甲基化测序(MeRIP-seq2)
技术优势:
- 起始量低:样本起始量可降低至10-20μg,最低仅需1μg总RNA;
- 转录组范围内:可以同时检测mRNA和lncRNA;
- 样本要求:可用于动物、植物、细胞及组织的m6A检测;
- 重复性高:IP富集重复性高,最大化降低抗体富集偏差;
- 应用范围广:广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症的发生与发展、药物应答等研究领域。
研究方向:
m6A甲基化目前主要运用在分子机制的理论性研究
- 疾病发生发展:肿瘤、代谢疾病(如肥胖/糖尿病)、神经和精神疾病(如阿尔兹海默症/抑郁症)、炎症…
- 发育和分化:早期胚胎发育、个体/组织/器官生长发育、干细胞分化与命运决定、衰老
- 环境暴露与响应:污染、抗逆、生活方式
关于m6A甲基化研究思路
(1)整体把握m6A甲基化图谱特征:m6A peak数量变化、m6A修饰基因数量变化、单个基因m6A peak数量分析、m6A peak在基因元件上的分布、m6A peak的motif分析、m6A peak修饰基因的功能分析
(2)筛选具体差异m6A peak和基因:差异m6A peak鉴定、非时序数据的分析策略、时序数据的分析策略、差异m6A修饰基因的功能分析、差异m6A修饰基因的PPI分析、候选基因的m6A修饰可视化展示
(3)m6A甲基化组学&转录组学关联分析:Meta genes整体关联、DMG-DEG对应关联、m6A修饰目标基因的筛选策略
(4)进一步验证或后期试验
易基因科技提供全面的RNA甲基化研究整体解决方案。
参考文献:
Yu F, Zhu AC, Liu S, Gao B, Wang Y, Khudaverdyan N, Yu C, Wu Q, Jiang Y, Song J, Jin L, He C, Qian Z. RBM33 is a unique m6A RNA-binding protein that regulates ALKBH5 demethylase activity and substrate selectivity. Mol Cell. 2023 Jun 15;83(12):2003-2019.e6.
相关阅读:
干货:m6A RNA甲基化MeRIP-seq测序分析实验全流程解析
项目集锦 | 易基因近期m6A甲基化(MeRIP-seq)研究成果
干货系列:m6A RNA甲基化研究的前期探索性实验思路
项目文章 | 90天见刊,易基因m6A RNA甲基化(MeRIP)+转录组组学研究