[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,
Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network
- 论文信息
- 问题描述
- 方法
-
- 模型
- 前处理
- 后处理
-
- Post-processing
- Verification module
- 数据增广
- 实验结果
- 总结
- References
论文信息
题目: Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network
发表: IEEE TBME, 2022
作者信息: Muhammad Uzair Zahid, Serkan Kiranyaz, Turker Ince, Ozer Can Devecioglu, Muhammad E. H.
Chowdhury, Amith Khandakar, Anas Tahir and Moncef Gabbouj
问题描述
虽然当前已经有很多方法(比如知名的PT算法)被提出来用于ECG R-peak的检测, 然后这些方法在低质量的ECG信号上的识别效果并不是很好 .
下图展示的就是PT在CPSC 数据库上测试出现漏报以及误报的例子,
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第1张图片](http://img.e-com-net.com/image/info8/9bb2a9d5eb0b4191892d9827c8c29b2d.jpg)
这篇论文提出一种新的深度学习方法,并在MIT-BIH和CPSC两个数据库上进行了测试并取得了不错的效果.
方法
方法主要由数据增广, 前处理, 模型, 后处理三部分组成.
模型
模型如下图所示
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第2张图片](http://img.e-com-net.com/image/info8/bab1842f28a24f4a85fea87937f0f67c.jpg) 可以看到模型采用了UNet这样一种Encoder-Decoder架构, 实际上就是UNet在一维信号上的应用, 那怎么用呢? 实际上就是把R-peak 的检测看作密集的分类任务, 最终每个位置的输出为该位置为R-peak的概率.
可以看到模型采用了UNet这样一种Encoder-Decoder架构, 实际上就是UNet在一维信号上的应用, 那怎么用呢? 实际上就是把R-peak 的检测看作密集的分类任务, 最终每个位置的输出为该位置为R-peak的概率.
模型的输入为(N, 1, L), L=8000,对应20s的ECG segment, 实际上也是滑窗的长度.
模型的输出尺寸也为(N, 1, L).
模型定义如下:
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第3张图片](http://img.e-com-net.com/image/info8/b5337d268cfb4b948ef9b6b17b40d9fa.jpg)
可以看到: 6个down sampling layer将输出的尺寸降为原来的1/64, 然后6个up sampling layers将输出的尺寸升为encoder out的64倍, 输入尺寸保持不变, 最后再经过sigmoid激活, 将每个position的输出转化为概率.
采用BCE loss, 最终损失计算如下:
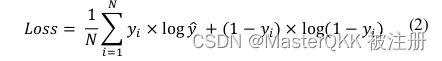 可以看到,每个position上实际上就是Binary cross entropy,
可以看到,每个position上实际上就是Binary cross entropy,
前处理
前处理比较简单, 由于实验采用了LOSO,
以CPSC数据为例, 该数据库的信息如下:
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第4张图片](http://img.e-com-net.com/image/info8/8e51d591b83b45cd906c17d9c832d878.jpg)
处理大致分为两步:
(1)通过滑动窗口将数据划分为segments,
这里需要注意的是:在构造训练集时,采用的是Non-overlaped window, 而在构造测试集时采用的是Overlaped window.
具体差异见下图,
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第5张图片](http://img.e-com-net.com/image/info8/bbe41de3fd0649b3af8d3df7cc42372f.jpg)
(2) range scaling
就是将每个segment的值缩放到[-1, 1], 并且该scaling是逐segment进行的,
作者是通过调用wfdb.processing.normalize_bound来实现的.
后处理
后处理部分较为复杂, 作者将其称之为post-processing和Verification module.
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第6张图片](http://img.e-com-net.com/image/info8/6036a8658f594e62be121029003aa31f.jpg)
简单来说后处理部分就是利用thresholding, 以及原始的ECG信号等先验信息来设计规则进一步筛选,最终得出有效的R-peak位置.
Post-processing
如代码所示,分为三步:
(1) 通过设置threshold筛选peaks;
(2) 利用极值的定义通过所谓的correct_peaks进一步修正peaks的位置, 这里调用的实际上是wfdb.processing.correct_peaks;
(3) 设置规则进一步筛选peaks;
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第7张图片](http://img.e-com-net.com/image/info8/387f0800738c4006b1178d832be49f85.jpg)
Verification module
代码部分逻辑比较复杂, 效果如上图所示,
def verifier(ecg, R_peaks, R_probs, ver_wind = 60):
#
del_indx = []
wind = 30
check_ind = np.squeeze(np.array(np.where(R_probs < 0.95)))
if R_peaks[check_ind[-1]] == R_peaks[-1]:
check_ind = check_ind[:-1]
for ind in check_ind:
two_ind = [ind, ind+1]
diff = R_peaks[two_ind[1]] - R_peaks[two_ind[0]]
# 60 for chinese data
if diff < ver_wind:
try:
two_probs = [ R_probs[two_ind[0]] , R_probs[two_ind[1]] ]
two_peaks = [ R_peaks[two_ind[0]] , R_peaks[two_ind[1]] ]
beat1 = ecg[ two_peaks[0] - wind : two_peaks[0] + wind ]
beat2 = ecg[ two_peaks[1] - wind : two_peaks[1] + wind ]
for i in range(two_ind[0]-1,two_ind[0]-1-30,-1):
for thr_p in [0.6,0.5,0.4,0.3,0.2,0.1]:
if(R_probs[i] > thr_p):
#print(i)
prv_beat = ecg[ R_peaks[i] - wind : R_peaks[i] + wind ]
break
for i in range(two_ind[1]+1,two_ind[1]+1+30):
for thr_p in [0.6,0.5,0.4,0.3,0.2,0.1]:
if i == len(R_probs):
nxt_beat = ecg[ R_peaks[i-1] - wind : R_peaks[i-1] + wind]
break
if(R_probs[i] > thr_p):
#print(i)
nxt_beat = ecg[ R_peaks[i] - wind : R_peaks[i] + wind]
break
else:
# Continue if the inner loop wasn't broken.
continue
# Inner loop was broken, break the outer.
break
if len(nxt_beat) != 60:
nxt_beat = prv_beat
if len(prv_beat) != 60:
prv_beat = nxt_beat
if len(beat1) != 60:
beat1 = beat2
if len(beat2) != 60:
beat2 = beat1
X1 = np.corrcoef(np.squeeze(beat1),np.squeeze(prv_beat))[0,1]
X2 = np.corrcoef(np.squeeze(beat1),np.squeeze(nxt_beat))[0,1]
Y1 = np.corrcoef(np.squeeze(beat2),np.squeeze(prv_beat))[0,1]
Y2 = np.corrcoef(np.squeeze(beat2),np.squeeze(nxt_beat))[0,1]
si = np.argmin([X1*X2, Y1*Y2])
del_indx.append(two_ind[si])
except:
pass
R_peaks_ver = np.delete(R_peaks, del_indx)
R_probs_ver = np.delete(R_probs, del_indx)
return R_peaks_ver, R_probs_ver
数据增广
由于心律失常搏动明显少于正常搏动, 为了使S-(室上性异位搏动)和V-(心室异位搏动)搏动检测更准确和鲁棒, 我们通过添加来自噪声应力测试数据库(NST-DB)的基线漂移和运动伪影,从包含一个或多个心律失常搏动的20秒ECG segment来生成增强的心律失常搏动。
这里的Data augmentation实际上起到了使模型更加鲁棒以及可以segment Imbalance的作用.
实验结果
下表为分别在CPSC和MITDB上的实验结果,
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第9张图片](http://img.e-com-net.com/image/info8/7271eb8162d14e7eb3d29b5b77828c63.jpg)
作者还分别对Data augmentation和Verification module进行了ablations, 如下:
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第10张图片](http://img.e-com-net.com/image/info8/25797fdfed724ec09abe198547b3638e.jpg)
总结
效果能这么好,主要还是深度学习模型与规则/先验信息的结合,
References
1.Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network
![[论文评析]Robust R-Peak Detection in Low-Quality Holter ECGs using 1D Convolutional Neural Network,_第8张图片](http://img.e-com-net.com/image/info8/9756a13d83bb44f8ac4919435000d986.jpg)