DEAP库文档教程五----计算统计
本小结将重点围绕模型在计算统计方面的问题,进行详细的论述
1、Computing Statistics
通常情况下,我们想要在优化过程中编辑数据。Statistic模块可以在任何设计好的目标上改变一些本不可改变的数据。为了达到这个目的,需要使用与工具箱中完全相同的语法在静态数据中注册统计函数。
states = tools.Statistics(key = lambda ind : ind.fitness.values)
使用key的第一个参数作为统计对象。这个key必须支持一个可以在之后被应用到数据上的函数从而得到统计结果。之前的例子使用了fitness.values()中每一个元素的属性。
states.register('avg', numpy.mean)
states.register('std', numpy.std)
states.register('min', numpy.min)
states.register('max', numpy.max)
2、Predefined Algorithms -预定义算法
当使用一个预定义的算法时,例如esSimple()/eaMuPlusLambada()/eaMuCommaLambda()/eaGenerateUpdata(),之前创建的统计目标可以作为算法的属性。
pop, logbook = algorithms.esSimple(pop, toolbox, cxpb = 0.5, mutpb = 0.2,
ngen = 0, stats = stats, verbose = True)
统计将会在每一次迭代中自动的进行计算。详细参数在优化过程中会打印在屏幕上。一旦算法返回,最终的种群和一个logbook将会返回。在下一节可以看到更详细的信息。
3、Writing Your Own Algorithm
这种情况主要是解决不是直接调用的模块函数,就是函数式自己编写的情况下,我们如何记录结果数据
当编写自己的算法时,包含统计时十分简单的。只需要去在需要的目标上编写统计。例如,在一个给定种群上编写统计需要调用compile()方法完成。
record = stats.compile(pop)
compile--编译
这些用于编辑函数的属性必须在一个迭代元素中,这样这些key才会被调用。这里,我们的种群(pop)包含了许多个体。统计目标将会在每一个个体上调用key函数获取fitness.values属性的值。这个结果数组的值最终会给到每一个统计函数并且将结果输入到record字典中,每一个key都会与相应的函数相关联。
4、Multi-objective Statistics
正如统计可以通过numpy函数直接进行计算,所有的目标将会通过默认numpy的属性联合在一起。接下来,一个需要明确的事情是每一个axis的操作。这会通过给予axis一个额外的属性作为注册函数达成。
stats = tools.Statistics(key=lambda ind: ind.fitness.values)
stats.register("avg", numpy.mean, axis=0)
stats.register("std", numpy.std, axis=0)
stats.register("min", numpy.min, axis=0)
stats.register("max", numpy.max, axis=0)
5、Logging Data
一旦数据通过统计产生,我们可以使用Logbook对它进行存储。Logbook是用来
按时间顺序排列的条目(如字典)。它会直接兼容数据类型并且返回统计目标,但是不会被数据限制。实际上,任何东西都可以包含在日志的条目中。
logbook = tools.Logbook()
logbook.record(gen=0, evals=30, **record)
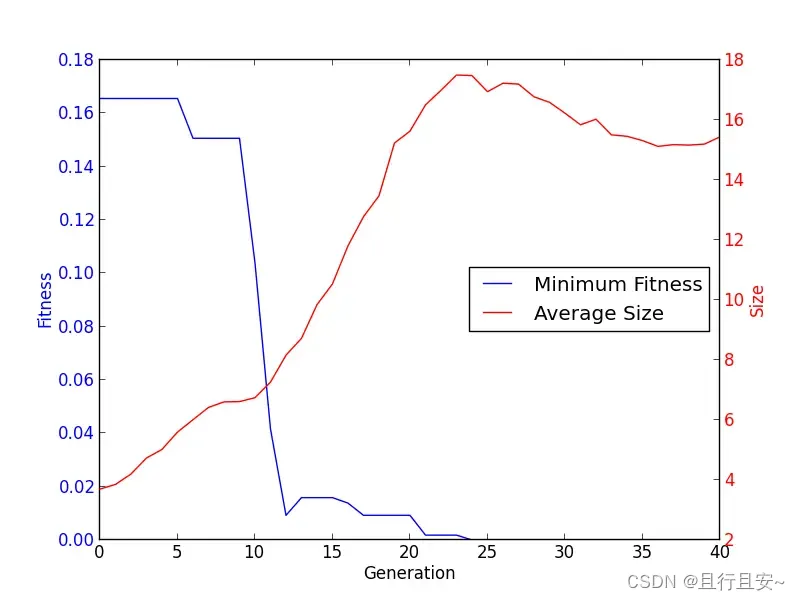
6、Some Plotting Sugar
在优化过程中最常用的操作就是在图中显示进化过程。Logbook可以有效地执行这一操作。使用选择方法,我们可以调用需要的数据并且使用matplotlib去绘制图形。
gen = logbook.select("gen")
fit_mins = logbook.chapters["fitness"].select("min")
size_avgs = logbook.chapters["size"].select("avg")
import matplotlib.pyplot as plt
fig, ax1 = plt.subplots()
line1 = ax1.plot(gen, fit_mins, "b-", label="Minimum Fitness")
ax1.set_xlabel("Generation")
ax1.set_ylabel("Fitness", color="b")
for tl in ax1.get_yticklabels():
tl.set_color("b")
ax2 = ax1.twinx()
line2 = ax2.plot(gen, size_avgs, "r-", label="Average Size")
ax2.set_ylabel("Size", color="r")
for tl in ax2.get_yticklabels():
tl.set_color("r")
lns = line1 + line2
labs = [l.get_label() for l in lns]
ax1.legend(lns, labs, loc="center right")
plt.show()