SCS【34】单细胞/T细胞/抗体免疫库数据分析(immunarch)
简 介
immunarch是专为分析T细胞受体(TCR)和B细胞受体(BCR)而设计的R包,主要为医学科学家和生物信息学家量身定制。immunarch的使命是使免疫测序数据分析尽可能轻松,帮助您专注于研究而不是编码。
关键特性
1. Data agnostic
快速轻松地操作免疫库数据:
A. 软件包自动检测文件的格式-不再猜测文件的格式(可分析任何类型的数据:single-cell, bulk, data tables, databases ),只需将它们传递给软件包;
B. 支持所有流行的TCR和BCR分析格式,包括单细胞数据:ImmunoSEQ, IMGT, MiTCR, MiXCR, MiGEC, MigMap, VDJtools, TCR, AIRR, 10XGenomics, ArcherDX,未来会有更多;
C. 适用于熟悉的任何数据源: R data frames, data tables from data.table, databases like MonetDB, Apache Spark data frames via sparklyr;
2. Beginner-friendly
免疫库分析变得简单:
A. 大多数方法都包含在几个具有明确名称的主要函数中——不再需要记住几十个具有模糊名称的函数。
B. 曲目重叠分析(常用指标包括重叠系数、Jaccard指数和Morisita 's重叠指数)。
C. 基因使用估计(相关性,Jensen-Shannon散度,聚类)。
D. 多样性评价(生态多样性指数、基尼指数、逆辛普森指数、方差分析)。
E. 跨越时间点的克隆型跟踪,广泛应用于疫苗接种和癌症免疫学领域;
F. K-mer分布测量和统计。
G. 即将发布的下一个版本:CDR3氨基酸理化性质评估,突变网络。
3. Seamless publication-ready plots with a built-in tool for visualisation manipulation
A. 丰富的可视化程序与ggplot2;
B. 内置工具FixVis使您的情节出版准备就绪:轻松更改字体大小,文本角度,标题,图例和更多明确的GUI;
软件包安装
软件包安装有几个方式,每个都是常用的方法,非常方便,这里我就用第一种方式进行安装:
Latest release on CRAN
install.packages("immunarch")Latest release on GitHub
install.packages(c("devtools", "pkgload")) # skip this if you already installed these packages
devtools::install_github("immunomind/immunarch")
devtools::reload(pkgload::inst("immunarch"))Latest pre-release on GitHub
install.packages(c("devtools", "pkgload")) # skip this if you already installed these packages
devtools::install_github("immunomind/immunarch", ref="dev")
devtools::reload(pkgload::inst("immunarch"))数据读取
数据需要我们准备两个文件,一个是meta文件,这个也是做单细胞分析必须有的文件,另一个就是单细胞表达矩阵,这个大家也非常熟悉,因此格式就不多说,我们看下immdata例子即可:
library(immunarch) # Load the package into R
library(knitr)
data(immdata) # Load the test dataset
kable(immdata$meta)| Sample | ID | Sex | Age | Status | Lane |
|---|---|---|---|---|---|
| A2-i129 | C1 | M | 11 | C | A |
| A2-i131 | C2 | M | 9 | C | A |
| A2-i133 | C4 | M | 16 | C | A |
| A2-i132 | C3 | F | 6 | C | A |
| A4-i191 | C8 | F | 22 | C | B |
| A4-i192 | C9 | F | 24 | C | B |
| MS1 | MS1 | M | 12 | MS | C |
| MS2 | MS2 | M | 30 | MS | C |
| MS3 | MS3 | M | 8 | MS | C |
| MS4 | MS4 | F | 14 | MS | C |
| MS5 | MS5 | F | 15 | MS | C |
| MS6 | MS6 | F | 15 | MS | C |
kable(immdata$data$`A2-i129`[1:5, ])| Clones | Proportion | CDR3.nt | CDR3.aa | V.name | D.name | J.name | V.end | D.start | D.end | J.start | VJ.ins | VD.ins | DJ.ins | Sequence |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 173 | 0.0203529 | TGCGCCAGCAGCCAAGAAGGGACAGGGTATTCCGGGGAGCTGTTTTTT | CASSQEGTGYSGELFF | TRBV4-1 | TRBD1 | TRBJ2-2 | 16 | 18 | 26 | 31 | -1 | 1 | 4 | NA |
| 163 | 0.0191765 | TGCGCCAGCAGCTACAGGGTTGGCACAGATACGCAGTATTTT | CASSYRVGTDTQYF | TRBV4-1 | TRBD1 | TRBJ2-3 | 11 | 13 | 18 | 22 | -1 | 1 | 3 | NA |
| 66 | 0.0077647 | TGTGCCACCAGCACCAACAGGGGCGGAACCCCAGCAGATACGCAGTATTTT | CATSTNRGGTPADTQYF | TRBV15 | TRBD1 | TRBJ2-3 | 11 | 16 | 22 | 34 | -1 | 4 | 11 | NA |
| 54 | 0.0063529 | TGTGCCACCAGCATCGGAGGCGGGAGCTACGAGCAGTACTTC | CATSIGGGSYEQYF | TRBV15 | TRBD2 | TRBJ2-7 | 11 | 19 | 25 | 26 | -1 | 7 | 0 | NA |
| 48 | 0.0056471 | TGTGCCAGCAGTCCTTGGACAGGGAGTATGGCCCTCCACTTT | CASSPWTGSMALHF | TRBV27 | TRBD1 | TRBJ1-6 | 11 | 16 | 23 | 31 | -1 | 4 | 7 | NA |
colnames(immdata$data$`A2-i129`[1:5, ])
## [1] "Clones" "Proportion" "CDR3.nt" "CDR3.aa" "V.name"
## [6] "D.name" "J.name" "V.end" "D.start" "D.end"
## [11] "J.start" "VJ.ins" "VD.ins" "DJ.ins" "Sequence"实例操作
计算基本统计数据并可视化
统计数据主要包括
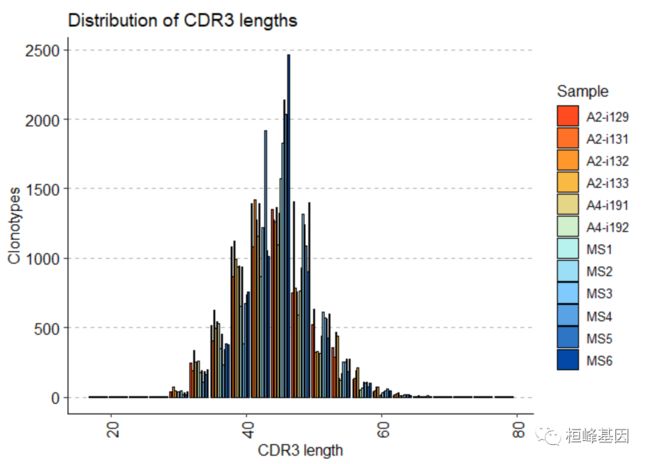
可视化 CDR3 长度分布
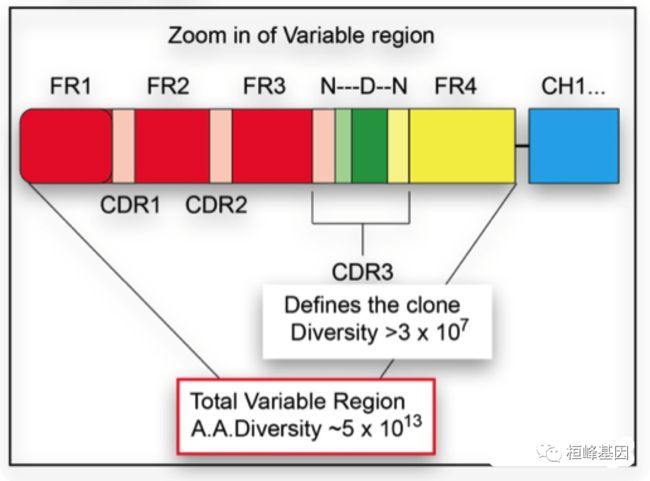
T/B 细胞表面有能特异性结合某种抗原的受体,叫做T/B 细胞表面受体 TCR/BCR (T/B cell recepter)。TCR/BCR 上存在一块区域叫互补决定区(Complementary Determining Region, CDR),包含CDR1、CDR2、CDR3,其中CDR3最高变,在抗原识别中起关键作用。
repExplore(immdata$data, "lens") %>%
vis() # Visualise the length distribution of CDR3可视化克隆型的相对丰度
repClonality(immdata$data, "homeo") %>%
vis() # Visualise the relative abundance of clonotypes探索和比较T-cell and B-cell 的功能
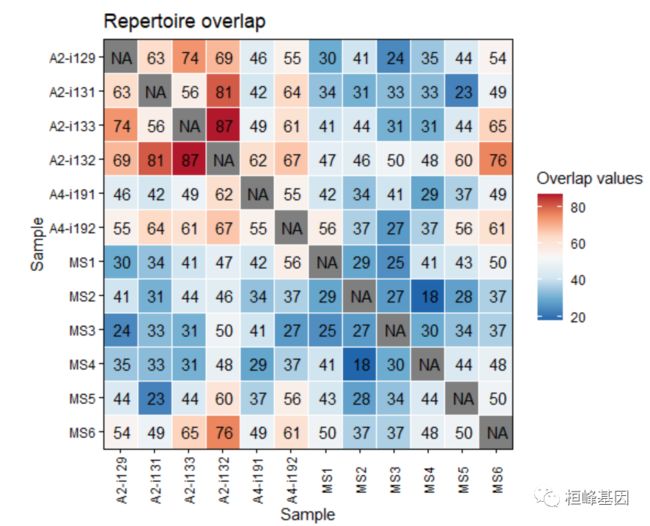
建立库之间共享的公共克隆型热图
repOverlap(immdata$data) %>%
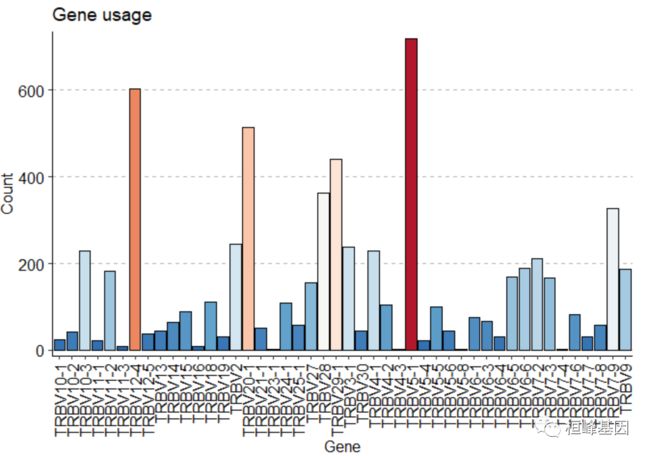
vis()可视化 V-gene 的分布
geneUsage(immdata$data[[1]]) %>%
vis()基于按病人的状态分组可视化多样性
这时可参考method里面的多样性估计的方法:
method Picks a method used for estimation out of a following list: chao1, hill, div, gini.simp, inv.simp, gini, raref, d50, dxx.
每个方法具体详细可参考如下:
chao1
repDiversity(immdata$data) %>%
vis(.by = "Status", .meta = immdata$meta)Hill numbers
repDiversity(.data = immdata$data, .method = "hill", .max.q = 6, .min.q = 1, .do.norm = NA,
.laplace = 0) %>%
vis()diversity
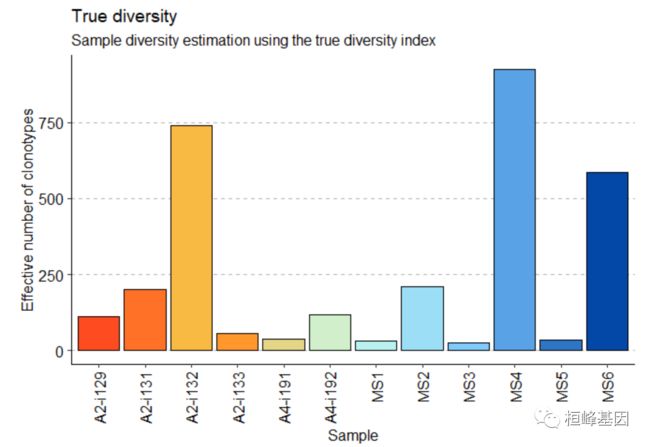
repDiversity(.data = immdata$data, .method = "div", .q = 5, .do.norm = NA, .laplace = 0) %>%
vis()Gini-Simpson
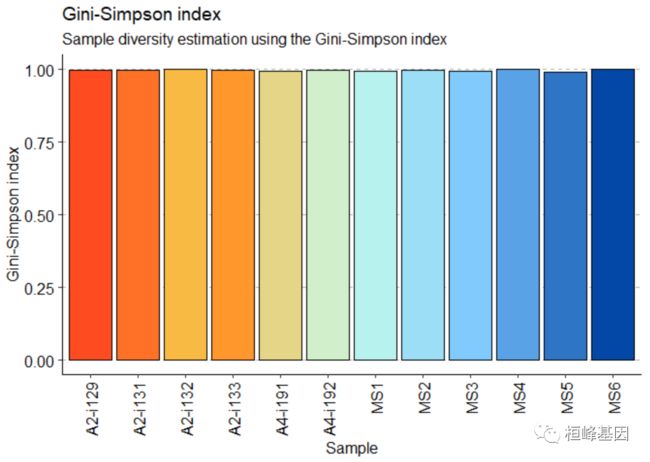
repDiversity(.data = immdata$data, .method = "gini.simp", .q = 5, .do.norm = NA,
.laplace = 0) %>%
vis()inverse Simpson
repDiversity(.data = immdata$data, .method = "inv.simp", .do.norm = NA, .laplace = 0) %>%
vis()Gini coefficient
repDiversity(.data = immdata$data, .method = "gini", .do.norm = NA, .laplace = 0)
## [,1]
## A2-i129 0.2297097
## A2-i131 0.2252784
## A2-i133 0.2513861
## A2-i132 0.2017009
## A4-i191 0.3863010
## A4-i192 0.3064599
## MS1 0.3610387
## MS2 0.1561629
## MS3 0.2396675
## MS4 0.1224806
## MS5 0.3320779
## MS6 0.1278508
## attr(,"class")
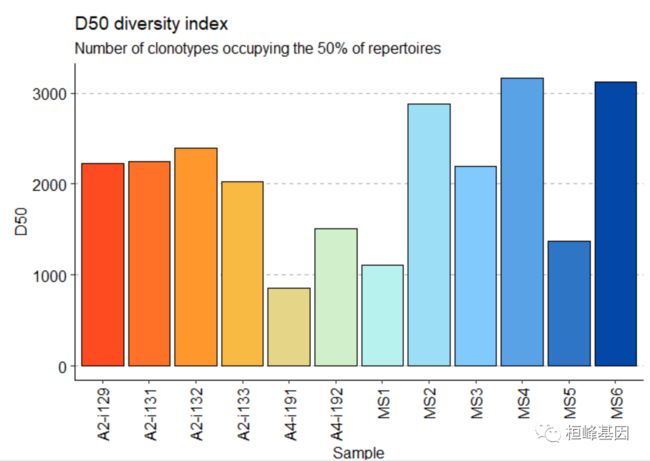
## [1] "immunr_gini" "matrix" "array"d50
repDiversity(.data = immdata$data, .method = "d50") %>%
vis()Reference
Nazarov V, Tsvetkov V, Fiadziushchanka S, Rumynskiy E, Popov A, Balashov I, Samokhina M (2023). immunarch: Bioinformatics Analysis of T-Cell and B-Cell Immune Repertoires.
单细胞生信分析教程往期回顾
桓峰基因公众号推出单细胞生信分析教程并配有视频在线教程,目前整理出来的相关教程目录如下:
Topic 6. 克隆进化之 Canopy
Topic 7. 克隆进化之 Cardelino
Topic 8. 克隆进化之 RobustClone
SCS【1】今天开启单细胞之旅,述说单细胞测序的前世今生
SCS【2】单细胞转录组 之 cellranger
SCS【3】单细胞转录组数据 GEO 下载及读取
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【5】单细胞转录组数据可视化分析 (scater)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【14】单细胞调节网络推理和聚类 (SCENIC)
SCS【15】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (CellPhoneDB)
SCS【16】从肿瘤单细胞RNA-Seq数据中推断拷贝数变化 (inferCNV)
SCS【17】从单细胞转录组推断肿瘤的CNV和亚克隆 (copyKAT)
SCS【18】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (iTALK)
SCS【19】单细胞自动注释细胞类型 (Symphony)
SCS【20】单细胞数据估计组织中细胞类型(Music)
SCS【21】单细胞空间转录组可视化 (Seurat V5)
SCS【22】单细胞转录组之 RNA 速度估计 (Velocyto.R)
SCS【23】单细胞转录组之数据整合 (Harmony)
SCS【24】单细胞数据量化代谢的计算方法 (scMetabolism)
SCS【25】单细胞细胞间通信第一部分细胞通讯可视化(CellChat)
SCS【26】单细胞细胞间通信第二部分通信网络的系统分析(CellChat)
SCS【27】单细胞转录组之识别标记基因 (scran)
SCS【28】单细胞转录组加权基因共表达网络分析(hdWGCNA)
SCS【29】单细胞基因富集分析 (singleseqgset)
SCS【30】单细胞空间转录组学数据库(STOmics DB)
SCS【31】减少障碍,加速单细胞研究数据库(Single Cell PORTAL)
SCS【32】基于scRNA-seq数据中推断单细胞的eQTLs (eQTLsingle)
SCS【33】单细胞转录之全自动超快速的细胞类型鉴定 (ScType)
利用这个软件包实现了全自动化快速免疫细胞分析,还是要求有一定生信分析基础的,有需求的老师可以联系桓峰基因,关注桓峰基因公众号,轻松学生信,高效发文章!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!
http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/