基于随机森林+小型智能健康推荐助手(心脏病+慢性肾病健康预测+药物推荐)——机器学习算法应用(含Python工程源码)+数据集(一)
目录
- 前言
- 总体设计
-
- 系统整体结构图
- 系统流程图
- 运行环境
-
- Python环境
- 依赖库
- 模块实现
-
- 1. 疾病预测
-
- 1)数据预处理
-
- 心脏病数据集预处理
- 慢性肾病数据预处理
- 2)模型训练及保存
-
- 定义模型结构
-
- 心脏病数据集定义模型
- 慢性肾病数据集定义模型
- 保存模型
-
- 心脏病模型保存
- 慢性肾病数据集定义模型
- 3)模型应用
-
- 慢性肾病模型应用创新
- 慢性肾病模型应用创新
- 其它相关博客
- 工程源代码下载
- 其它资料下载
前言
本项目基于Kaggle上公开的数据集,旨在对心脏病和慢性肾病进行深入的特征筛选和提取。它利用了随机森林机器学习模型,通过对这些特征进行训练,能够预测是否患有这些疾病。不仅如此,该项目还会根据患者的症状或需求,提供相关的药物推荐,从而实现了一款实用性强的智能医疗助手。
首先,项目收集了来自Kaggle的公开数据集,这些数据包含了与心脏病和慢性肾病相关的丰富信息。然后,通过数据预处理和特征工程,从这些数据中提取出最相关的特征,以用于机器学习模型的训练。
接下来,项目采用了随机森林机器学习模型,这是一种强大的分类算法。通过使用训练数据,模型能够学习不同特征与心脏病和慢性肾病之间的关联。一旦模型经过训练,它可以对新的患者数据进行预测,判断患者是否有这些疾病。
除了疾病预测,该项目还具备一个药物推荐系统。基于患者的症状、需求和疾病诊断,系统会推荐适合的药物和治疗方案,以提供更全面的医疗支持。
综合来看,这个项目不仅可以预测心脏病和慢性肾病,还可以提供个性化的治疗建议。这种智能医疗助手有望提高医疗决策的准确性,为患者提供更好的医疗体验,并对医疗资源的合理分配起到积极作用。
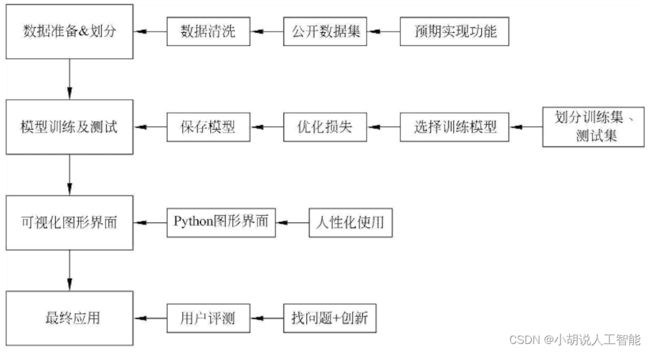
总体设计
本部分包括系统整体结构图和系统流程图。
系统整体结构图
系统整体结构如图所示。
系统流程图
系统流程如图所示。
运行环境
Python环境
需要Python 3.6及以上配置,在Windows环境下推荐下载Anaconda完成Python所需环境的配置,下载地址为https://www.anaconda.com/,也可下载虚拟机在Linux环境下运行代码。
依赖库
使用下面命令安装:
pip install pandas
模块实现
本项目包括2个功能,每个功能有3个模块:疾病预测、药物推荐、模块应用,下面分别给出各模块的功能介绍及相关代码。
1. 疾病预测
本模块是一个小型健康预测系统,预测两种疾病心脏病和慢性肾病。
1)数据预处理
心脏病数据集来源地址为https://archive.ics.uci.edu/dataset/45/heart+disease;慢性肾病数据集来源地址为https://www.kaggle.com/mansoordaku/ckdisease。两个数据集均包括300多名测试者的年龄、性别、静息血压、胆固醇含量等数据。
心脏病数据集预处理
加载数据集和数据预处理,大部分是通过Pandas库实现,相关代码如下:
#导入相应库函数
import pandas as pd
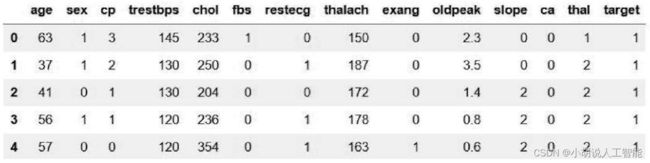
#读取心脏病数据集
df = pd.read_csv("../Thursday9 10 11/heart.csv")
df.head()
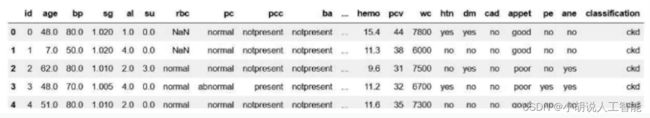
自动从csv中读取相应的数据,如图所示。
检查数据是否有缺省值,如果有数据会显示为NaN,且当数据有缺省值时不能对数据绘图可视化。
#检查是否有缺省值
df.loc[(df['age'].isnull()) |
(df['sex'].isnull()) |
(df['cp'].isnull()) |
(df['trestbps'].isnull()) |
(df['chol'].isnull()) |
(df['fbs'].isnull()) |
(df['restecg'].isnull()) |
(df['thalach'].isnull()) |
(df['exang'].isnull()) |
(df['oldpeak'].isnull()) |
(df['slope'].isnull()) |
(df['ca'].isnull()) |
(df['target'].isnull())]
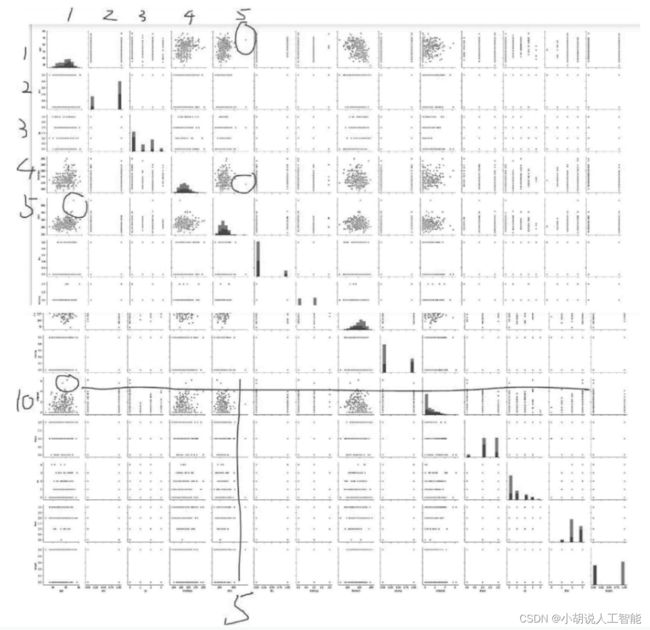
数据集没有缺省值,数据的尺度比较大,通过绘图方式观察可以检查出错误数据,如图所示。
#通过seaborn绘图,观察数据
sns.pairplot(df.dropna(), hue='target')
通过观察,第五列(血液中胆固醇含量)和第十行(静息血压)有部分点和其它点有较大距离,绘制数据分布图进一步分析。
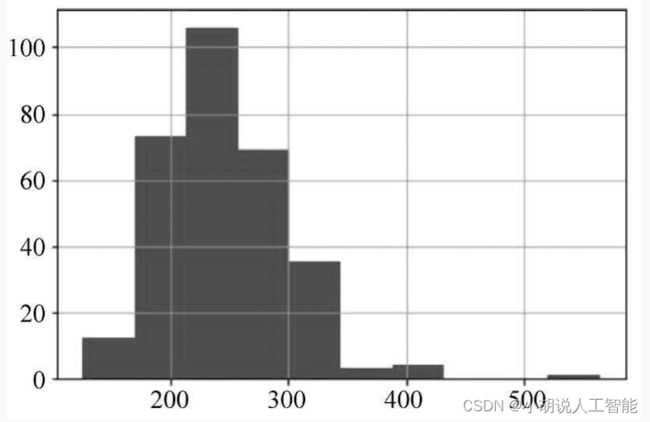
#绘制血液中胆固醇数据分布
df['chol'].hist()
#绘制静息血压分布图
df[‘treatbps’].hist()
数据分布如图1和图2所示。血液中胆固醇含量达到500,静息血压最大值达到200。经过查阅资料,静息血压正常值应该在120~140,但是接近200的患者数据,是符合实际的。取得胆固醇含量最大值的同样是患者,没有不符合实际情况的数据。
下面是改变数据类型,例如,胸痛类型,1~4是类别变量,它的大小并不具备比较性,但是训练时数值大小会影响权重。所以要把类别变量转化为伪变量,把4个类别拆成4件,分别用0,1表示有或没有。
#将类别变量转换为伪变量
a = pd.get_dummies(df['cp'], prefix = "cp")
b = pd.get_dummies(df['thal'], prefix = "thal")
c = pd.get_dummies(df['slope'], prefix = "slope")
frames = [df, a, b, c]
df = pd.concat(frames, axis = 1)
#保留转换后的变量即可,删除原来的类别变量
df = df.drop(columns = ['cp', 'thal', 'slope'])
转换成功后如图所示。
最后使用Scikit-learn的train_test_split()自动划分训练集和测试集。
#标签是target,是否患病
y = df.target.values
x_data = df.drop(['target'], axis = 1)
#丢弃标签,也就是最后一行target
#按4:1划分训练集测试集
x_train, x_test, y_train, y_test = train_test_split(x_data,y,test_size = 0.2,random_state=0)
x_train = x_train.T
y_train = y_train.T
x_test = x_test.T
y_test = y_test.T
#心脏病数据集预处理完成
慢性肾病数据预处理
通过Pandas读取慢性肾病数据集,读取成功效果如图所示。
#读取肾病数据集
df = pd.read_csv("../Thursday9 10 11/kidney_disease.csv")
df.head()
对数据类型进行处理,例如食欲(appet)数据为good和poor,脓细胞团(pcc)为notpresent和present,将类别变量转换为伪变量0和1。
#yes/no; abnormal/normal;present/notpresent;good/poor都转换为0/1
df[['htn','dm','cad','pe','ane']] = df[['htn','dm','cad','pe','ane']].replace(to_replace={'yes':1,'no':0})
df[['rbc','pc']] = df[['rbc','pc']].replace(to_replace={'abnormal':1,'normal':0})
df[['pcc','ba']] = df[['pcc','ba']].replace(to_replace={'present':1,'notpresent':0})
df[['appet']] = df[['appet']].replace(to_replace={'good':1,'poor':0,'no':np.nan})
df['classification'] = df['classification'].replace(to_replace={'ckd':1.0,'ckd\t':1.0,'notckd':0.0,'no':0.0})
df.rename(columns={'classification':'class'},inplace=True)
#将对患病有积极作用的变量设为0
df['pe'] = df['pe'].replace(to_replace='good',value=0)
df['appet'] = df['appet'].replace(to_replace='no',value=0)
df['cad'] = df['cad'].replace(to_replace='\tno',value=0)
df['dm'] = df['dm'].replace(to_replace={'\tno':0,'\tyes':1,' yes':1, '':np.nan})
#ID列去掉,为了表格中数据条理清晰而建立的变量
df.drop('id',axis=1,inplace=True)
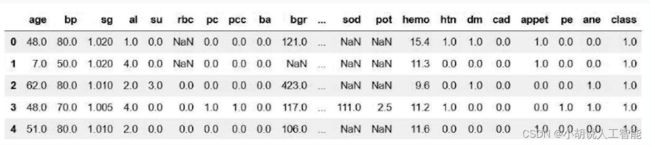
转换成功后如图所示。
检查默认值的变量如下图所示。从图中可以看出缺省值数量不小,由于数据集不大,需要采用均值归一法,对病人和正常人分别取所有测量值的平均值来填补缺省值。
#对病人所有测量值取均值
average0_age = df.loc[df['class'] ==True, 'age'].mean()
average0_bp = df.loc[df['class'] == True, 'bp'].mean()
average0_sg = df.loc[df['class'] == True, 'sg'].mean()
average0_al = df.loc[df['class'] == True, 'al'].mean()
average0_su = df.loc[df['class'] == True, 'su'].mean()
average0_rbc = df.loc[df['class'] == True, 'rbc'].mean()
average0_pc = df.loc[df['class'] == True, 'pc'].mean()
average0_pcc = df.loc[df['class'] == True, 'pcc'].mean()
average0_ba = df.loc[df['class'] == True, 'ba'].mean()
average0_bgr = df.loc[df['class'] == True, 'bgr'].mean()
average0_bu = df.loc[df['class'] == True, 'bu'].mean()
average0_sc = df.loc[df['class'] == True, 'sc'].mean()
average0_sod = df.loc[df['class'] == True, 'sod'].mean()
average0_pot = df.loc[df['class'] == True, 'pot'].mean()
average0_hemo = df.loc[df['class'] == True, 'hemo'].mean()
average0_htn = df.loc[df['class'] == True, 'htn'].mean()
average0_dm = df.loc[df['class'] == True, 'dm'].mean()
average0_cad = df.loc[df['class'] == True, 'cad'].mean()
average0_appet = df.loc[df['class'] ==True, 'appet'].mean()
average0_pe = df.loc[df['class'] == True, 'pe'].mean()
average0_ane = df.loc[df['class'] == True, 'ane'].mean()
#对正常人所有测量值取均值
average1_age = df.loc[df['class'] == False, 'age'].mean()
average1_bp = df.loc[df['class'] == False, 'bp'].mean()
average1_sg = df.loc[df['class'] == False, 'sg'].mean()
average1_al = df.loc[df['class'] == False, 'al'].mean()
average1_su = df.loc[df['class'] == False, 'su'].mean()
average1_rbc = df.loc[df['class'] == False, 'rbc'].mean()
average1_pc = df.loc[df['class'] == False, 'pc'].mean()
average1_pcc = df.loc[df['class'] == False, 'pcc'].mean()
average1_ba = df.loc[df['class'] == False, 'ba'].mean()
average1_bgr = df.loc[df['class'] == False, 'bgr'].mean()
average1_bu = df.loc[df['class'] == False, 'bu'].mean()
average1_sc = df.loc[df['class'] == False, 'sc'].mean()
average1_sod = df.loc[df['class'] == False, 'sod'].mean()
average1_pot = df.loc[df['class'] == False, 'pot'].mean()
average1_hemo = df.loc[df['class'] == False, 'hemo'].mean()
average1_htn = df.loc[df['class'] == False, 'htn'].mean()
average1_dm = df.loc[df['class'] == False, 'dm'].mean()
average1_cad = df.loc[df['class'] == False, 'cad'].mean()
average1_appet = df.loc[df['class'] == False, 'appet'].mean()
average1_pe = df.loc[df['class'] == False, 'pe'].mean()
average1_ane = df.loc[df['class'] == False, 'ane'].mean()
#根据是患者还是正常人,求出的均值赋给所有缺省值。如果为null,则取均值
df.loc[(df['class']==True)&(df['age'].isnull()),'age']= average0_age
df.loc[(df['class']==True)&(df['bp'].isnull()),'bp']= average0_bp
df.loc[(df['class']==True)&(df['sg'].isnull()),'sg']= average0_sg
df.loc[(df['class']==True)&(df['al'].isnull()),'al']= average0_al
df.loc[(df['class']==True)&(df['su'].isnull()),'su']= average0_su
df.loc[(df['class']==True)&(df['rbc'].isnull()),'rbc']= average0_rbc
df.loc[(df['class']==True)&(df['pc'].isnull()),'pc']= average0_pc
df.loc[(df['class']==True)&(df['pcc'].isnull()),'pcc']= average0_pcc
df.loc[(df['class']==True)&(df['ba'].isnull()),'ba']= average0_ba
df.loc[(df['class']==True)&(df['bgr'].isnull()),'bgr']= average0_bgr
df.loc[(df['class']==True)&(df['bu'].isnull()),'bu']= average0_bu
df.loc[(df['class']==True) &(df['sc'].isnull()),'sc']= average0_sc
df.loc[(df['class']==True)&(df['sod'].isnull()),'sod']= average0_sod
df.loc[(df['class']==True)&(df['pot'].isnull()),'pot']= average0_pot
df.loc[(df['class']==True) &(df['hemo'].isnull()),'hemo'] =average0_hemo
df.loc[(df['class']==True) &(df['htn'].isnull()),'htn'] = average0_htn
df.loc[(df['class']==True) &(df['dm'].isnull()),'dm'] = average0_dm
df.loc[(df['class']==True) &(df['cad'].isnull()),'cad'] = average0_cad
df.loc[(df['class']==True)&(df['appet'].isnull()),'appet']=average0_appet
df.loc[(df['class']==True)&(df['pe'].isnull()),'pe'] = average0_pe
df.loc[(df['class']==True) &(df['ane'].isnull()),'ane'] = average0_ane
#正常人
df.loc[(df['class']==False)&(df['age'].isnull()),'age']= average1_age
df.loc[(df['class'] ==False) &(df['bp'].isnull()),'bp'] = average1_bp
df.loc[(df['class'] ==False) &(df['sg'].isnull()),'sg'] = average1_sg
df.loc[(df['class'] ==False) &(df['al'].isnull()),'al'] = average1_al
df.loc[(df['class'] ==False) &(df['su'].isnull()),'su'] = average1_su
df.loc[(df['class'] ==False) &(df['rbc'].isnull()),'rbc'] = average1_rbc
df.loc[(df['class'] ==False) &(df['pc'].isnull()),'pc'] = average1_pc
df.loc[(df['class'] ==False)&(df['pcc'].isnull()),'pcc'] = average1_pcc
df.loc[(df['class'] ==False)&(df['ba'].isnull()),'ba'] = average1_ba
df.loc[(df['class'] ==False&(df['bgr'].isnull()),'bgr'] = average1_bgr
df.loc[(df['class'] ==False)&(df['bu'].isnull()),'bu'] = average1_bu
df.loc[(df['class'] ==False)&(df['sc'].isnull()),'sc'] = average1_sc
df.loc[(df['class'] ==False)&(df['sod'].isnull()),'sod'] = average1_sod
df.loc[(df['class'] ==False)&(df['pot'].isnull()),'pot'] = average1_pot
df.loc[(df['class'] ==False)&(df['hemo'].isnull()),'hemo']= average1_hemo
df.loc[(df['class'] ==False)&(df['htn'].isnull()),'htn'] = average1_htn
df.loc[(df['class'] ==False)&(df['dm'].isnull()),'dm'] = average1_dm
df.loc[(df['class'] ==False) &(df['cad'].isnull()),'cad'] = average1_cad
df.loc[(df['class']==False)&(df['appet'].isnull()),'appet']=average1_appet
df.loc[(df['class'] ==False) &(df['pe'].isnull()),'pe'] = average1_pe
df.loc[(df['class'] ==False) &(df['ane'].isnull()),'ane'] = average1_ane
#再次检查是否有缺省值
df.loc[(df['age'].isnull()) |
(df['bp'].isnull()) |
(df['sg'].isnull()) |
(df['al'].isnull()) |
(df['su'].isnull()) |
(df['rbc'].isnull()) |
(df['pc'].isnull()) |
(df['pcc'].isnull()) |
(df['ba'].isnull()) |
(df['bgr'].isnull()) |
(df['bu'].isnull()) |
(df['sc'].isnull()) |
(df['sod'].isnull()) |
(df['pot'].isnull()) |
(df['hemo'].isnull()) |
(df['htn'].isnull()) |
(df['dm'].isnull()) |
(df['cad'].isnull()) |
(df['appet'].isnull()) |
(df['pe'].isnull()) |
(df['ane'].isnull()) |
(df['class'].isnull())]
#使用Scikit-learn的train_test_split()函数自动划分训练集和测试集
X_train, X_test, y_train, y_test = train_test_split(df.iloc[:,:-1], df['class'],test_size = 0.33, random_state=44,stratify= df['class'] )
#慢性肾病数据预处理完成
2)模型训练及保存
数据加载进模型之后,需要定义模型结构,寻找优化参数并保存模型。
定义模型结构
本部分包括心脏病数据集定义模型和慢性肾病数据集定义模型。
心脏病数据集定义模型
相关代码如下:
#由于一次尝试过多参数会导致内存不足,所以分段寻找最大值
num=np.zeros(20,int)
for i in range(0,20):
num[i]=i
"""
每次将num扩大20,迭代改变随机森林中树的数量以及权重分配等参数。使用GridSearchCV自动寻找最优参数。
"""
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split, GridSearchCV
tuned_parameters = [{'n_estimators':num,'class_weight':[None,{0: 0.33,1:0.67},'balanced'],'random_state':[1]}]
rf = GridSearchCV(RandomForestClassifier(), tuned_parameters, cv=10,scoring='f1')
rf.fit(x_train.T, y_train.T)
print('Best parameters:')
print(rf.best_params_)
#训练完成后,使用print()函数输出最佳参数,森林中有1005棵树的时候准确率最高
#保存最优参数,将最佳参数带入模型进行训练
rf_best = rf.best_estimator_
rf_best.fit(x_train.T, y_train.T)
acc = rf.score(x_test.T,y_test.T)*100
accuracies['Random Forest'] = acc
print("Random Forest Algorithm Accuracy Score : {:.2f}%".format(acc))
#最佳参数的模型训练完成后,在测试集上计算模型准确率,达89.86%
慢性肾病数据集定义模型
相关代码如下:
#寻找随机森林最优参数
tuned_parameters= [{'n_estimators':[7,8,9,10,11,12,13,14,15,16],'max_depth':[2,3,4,5,6,None],'class_weight':[None,{0: 0.33,1:0.67},'balanced'],'random_state':[42]}]
clf = GridSearchCV(RandomForestClassifier(), tuned_parameters, cv=10,scoring='f1')
clf.fit(X_train, y_train)
print('Best parameters:')
print(clf.best_params_)
#训练完成后,使用print()函数输出最佳参数,随机森林中有7棵树时准确率最高
#将最优参数带入模型进行训练
accuracies = {}
rf = RandomForestClassifier(class_weight=None, max_depth= 6,n_estimators = 7, random_state = 42)
rf.fit(X_train, y_train)
acc = rf.score(X_test, y_test)*100
accuracies['Random Forest'] = acc
print("Random Forest Algorithm Accuracy Score : {:.2f}%".format(acc))
#最佳参数的模型训练完成后,在测试集上计算模型准确率,达100%
保存模型
为了能够被Python程序读取,需要将模型保存为.pkl格式的文件,利用pickle库中的模块进行模型的保存。
心脏病模型保存
相关代码如下:
import pickle
with open("model.pkl", "wb") as f:
pickle.dump(rf, f)
慢性肾病数据集定义模型
相关代码如下:
import pickle
with open("model_kidney.pkl", "wb") as f:
pickle.dump(rf, f)
3)模型应用
对于人的疾病来说,误诊带来的风险是极大的,正确率不是100%对患者就是损失。然而以往仅仅预测是否患上疾病,是一个二分类问题。原理上,机器对疾病的预测基于各项指标的权重。本项目充分运用机器学习的优点,将各特征重要性与各指标的数值相乘求和,得到一个加权值。在程序内部,预先对患者和正常人的各指标平均值与对应特征重要性加权求和,得到患者和正常人的平均水平。在定性判断为病人后,将用户的数值与病人均值做对比,定量给出用户的病情相对于大多数患者严重程度;如果用户被判断为正常人,则将他的各项数值与正常人均值做对比,给出用户相对于大多数正常人的亚健康程度。经过处理,不仅做到定性判断,还对用户情况进行量化。一方面防止偶然的误诊带来风险;另一方面给予患者与疾病抗争的希望,给体检正常的人敲响亚健康的警钟。
慢性肾病模型应用创新
#通过eli5得到各特征重要性
import eli5
from eli5.sklearn import PermutationImportance
perm = PermutationImportance(rf, random_state=1).fit(x_test.T, y_test.T)
eli5.show_weights(perm, feature_names = x_test.T.columns.tolist())
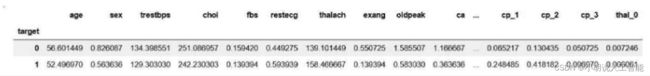
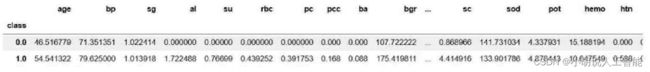
输出的各特征重要性如图3所示。从图中可以看出,年龄因素,对患病是否有影响,并不重要,符合常识。然后分别获得患者和正常人的平均值,如图4所示。
#求均值
df.groupby('target').mean()
#正常人平均水平
average0_count=np.multiply(average0,w)
average0_sum=sum(average0_count)
#病人平均水平
average1_count=np.multiply(average1,w)
average1_sum=sum(average1_count)
#输出得到的数值
print(average1_sum) #患者
print(average0_sum) #正常人
患者和正常人加权求和后的值如图所示。
将这个值保存,当用户使用时,判断出是患者还是正常人之后,根据比值大小定量判断具体情况。
慢性肾病模型应用创新
通过eli5得到各特征重要性,输出的各特征重要性如图所示。
import eli5
from eli5.sklearn import PermutationImportance
perm = PermutationImportance(rf, random_state=1).fit(x_test.T, y_test.T)
eli5.show_weights(perm, feature_names = x_test.T.columns.tolist())
import eli5 #for purmutation importance
from eli5.sklearn import PermutationImportance
perm = PermutationImportance(rf, random_state=1).fit(X_test, y_test)
eli5.show_weights(perm, feature_names = X_test.columns.tolist())
分别获取患者和正常人平均值,如图所示。
#求均值
#正常人平均水平
average0_count=np.multiply(average0,w)
average0_sum=sum(average0_count)
#病人平均水平
average1_count=np.multiply(average1,w)
average1_sum=sum(average1_count)
print(average1_sum)#患者
print(average0_sum)#正常人
#输出得到的数值
print(average1_sum)#病人
print(average0_sum)#正常人
患者和正常人加权求和后的值如图所示。
将这个值保存,当用户使用时,判断是患者还是正常人之后,根据比值大小定量出具体情况。
其它相关博客
-
基于随机森林+小型智能健康推荐助手(心脏病+慢性肾病健康预测+药物推荐)——机器学习算法应用(含Python工程源码)+数据集(二)
-
基于随机森林+小型智能健康推荐助手(心脏病+慢性肾病健康预测+药物推荐)——机器学习算法应用(含Python工程源码)+数据集(三)
工程源代码下载
详见本人博客资源下载页
其它资料下载
如果大家想继续了解人工智能相关学习路线和知识体系,欢迎大家翻阅我的另外一篇博客《重磅 | 完备的人工智能AI 学习——基础知识学习路线,所有资料免关注免套路直接网盘下载》
这篇博客参考了Github知名开源平台,AI技术平台以及相关领域专家:Datawhale,ApacheCN,AI有道和黄海广博士等约有近100G相关资料,希望能帮助到所有小伙伴们。