PSP - 配置 AlphaFold2 Multimer 的模型训练参数
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/133377559
整理配置 AlphaFold2 Multimer 的模型训练参数,参考相关论文、 AlphaFold v2.3.0 Technical Note、OpenFold、UniFold 等开源框架。AlphaFold2 的单体 (Monomer) 结构预测,在论文中,比较详细的提供了模型训练参数与细节,但是,在复合物 (Multimer) 中,并没有明确的提交全部训练参数配置。
参考论文与源码:
- Highly accurate protein structure prediction with AlphaFold
- Protein complex prediction with AlphaFold-Multimer
- GitHub - AlphaFold v2.3.0 Technical Note
- GitHub - OpenFold
- GitHub - Uni-Fold
训练参数核心分为6个模块:
- 数据
data:包括通用(common)、数据处理(data_module)、评估(eval)、预测(predict)、训练(train)、监督(supervised)等。- 其中
监督(supervised),主要是一些开关,与导入与训练中评估的相关特征all_atom_mask、all_atom_positions、resolution、is_distillation,以及use_clamped_fape与clamp_prob配合使用。具体参考 OpenFold 与 AF2 的源码。
- 其中
ema模型参数动态更新,每个 step 进行一次 EMA 滑动更新,设置成0.999相当于1000次更新完成,。globals全局参数,模型网络结构的一些参数,与 OpenFold 一致,注意训练时,chunk_size需要设置成null。loss参数:包括chain_centre_mass(Diff)、distogram(Same)、experimentally_resolved(Same)、fape(Same with Unifold)、lddt(Same)、masked_msa(Same)、supervised_chi(Same)、tm(Unused)、violation(Same)、rmsd_ca(自研)。- 计算 loss 的
openfold/utils/loss.py函数multi_chain_perm_align(),来自于Unifold - loss 部分需要调节的超参数较多。
- 计算 loss 的
model参数,网络设计的相关参数,需要与 AF2 匹配。relax参数,模型 relax 的相关参数,与OpenFold一致。
即核心修改就是 data 部分,与 loss 部分,其余不需修改。
其中 chunk_size 参数描述:
Note that chunking (as defined in section 1.11.8 of the AlphaFold 2 supplement) is enabled by default in inference mode. To disable it, set
globals.chunk_sizetoNonein the config. If a value is specified, OpenFold will attempt to dynamically tune it, considering the chunk size specified in the config as a minimum. This tuning process automatically ensures consistently fast runtimes regardless of input sequence length, but it also introduces some runtime variability, which may be undesirable for certain users. It is also recommended to disable this feature for very long chains (see below). To do so, set thetune_chunk_sizeoption in the config toFalse.
其中 chunk_size在训练时,设置成 None:
if train:
c.globals.blocks_per_ckpt = 1
c.globals.chunk_size = None
1. 数据模块
data 数据参数包括:common 通用、data_module 数据模块、eval 评估、predict 预测、train 训练、supervised 监督 等
- 暂时不需要修改
data_module数据处理、predict预测、supervised监督 - 通用
common参数:可修改参数是max_extra_msa,AF2 与 OpenFold Multimer (最新) 都是 2048,当前是1024,继承至 Monomer 逻辑。 train、eval训练:max_msa_clusters建议修改成 508;crop_size修改成 640,注意这个与显存占用相关。- 全部
template模版参数,暂时不需要修改,当前是 4。 distillation_prob:蒸馏数据配比由 0.75 降低至 0.5。- 主要修改点,即 MSA 部分,当前工程来源于 OpenFold Monomer 第1版,部分参数没有与 Multimer 对齐。
注意:在训练参数中,参数
interface_threshold即ca_ca_threshold,参考get_spatial_crop_idx()函数,OpenFold 与 UniFold 实现不同。
2. Loss 模块
loss参数包括:
chain_centre_mass(Diff):权重 1.0;UniFold,1.0;OpenFold 0.05,差距较大,OpenFold 多处显示是 0.05,更为可信,而且一般原子层面loss都是权重较低,值较大,建议 0.05。distogram(Same):权重 0.3,一致experimentally_resolved(Same):权重0.01,一致fape(Same with Unifold):权重1.0;一致lddt(Same):权重 0.01;一致。masked_msa(Same):权重 2.0;一致supervised_chi(Same):权重1.0;一致。tm(Unused):false 来源于 OpenFold Multimer Debug 版未添加,正式版已经添加;UniFold PAE 启用权重0.1,OpenFold 权重 0.1,建议启用。violation(Same):0.5,OpenFold 0.03 (Initial 不可信),UniFold 0.5 (Finetune),建议 0.5。
其他:eps 1.0e-08,OpenFold 是 1.0e-08,UniFold 与 AF2 一致,从 1.0e-4 ~ 1.0e-10。
即:
loss = chain_centre_mass*0.05 + distogram*0.3 + experimentally_resolved*0.01 + fape*1.0 + lddt*0.01 + masked_msa*2.0 + supervised_chi*1.0 + tm*0.1 + violation*0.5
训练日志如下:
2023-09-26 07:25:17,232 INFO 25675 [loss.py:1995] [CL] loss_name: fape, weight: 1.0, loss: 1.2547515630722046, add: 1.2547515630722046
2023-09-26 07:25:17,233 INFO 25675 [loss.py:1995] [CL] loss_name: supervised_chi, weight: 1.0, loss: 0.15294387936592102, add: 0.15294387936592102
2023-09-26 07:25:17,234 INFO 25675 [loss.py:1995] [CL] loss_name: distogram, weight: 0.3, loss: 0.40942829847335815, add: 0.12282849103212357
2023-09-26 07:25:17,235 INFO 25675 [loss.py:1995] [CL] loss_name: experimentally_resolved, weight: 0.01, loss: 0.002576087834313512, add: 2.57608771789819e-05
2023-09-26 07:25:17,239 INFO 25675 [loss.py:1995] [CL] loss_name: lddt, weight: 0.01, loss: 1.8179388046264648, add: 0.018179386854171753
2023-09-26 07:25:17,240 INFO 25675 [loss.py:1995] [CL] loss_name: chain_centre_mass, weight: 0.05, loss: 0.0, add: 0.0
2023-09-26 07:25:17,240 INFO 25675 [loss.py:1995] [CL] loss_name: violation, weight: 0.5, loss: 0.0052171749994158745, add: 0.0026085874997079372
2023-09-26 07:25:17,241 INFO 25675 [loss.py:1995] [CL] loss_name: masked_msa, weight: 2.0, loss: 0.39116162061691284, add: 0.7823232412338257
2023-09-26 07:25:17,243 INFO 25675 [loss.py:1995] [CL] loss_name: tm, weight: 0.1, loss: 4.158883094787598, add: 0.41588830947875977
3. 相关参考
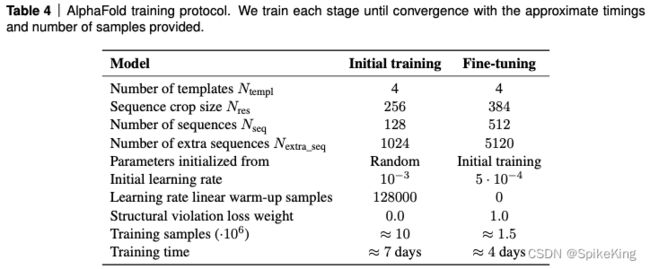
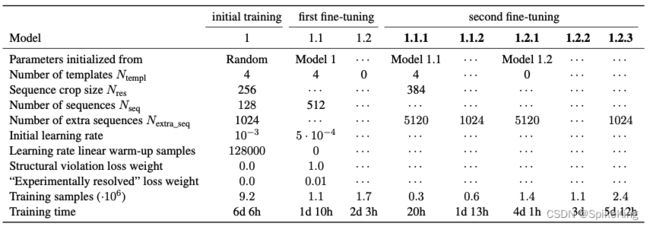
AF2 Monomer 训练协议 (Training Protocol) 的超参数,即:
不同训练阶段, AF2 Monomer 参数配置不同,如 MSA、Template 等,即:
AF2 Monomer Loss 权重,即:
其中, clamp_prob 参数,与 AF2 基本一致:
supervised:
clamp_prob: 0.9
AF2 的 clamp_prob 参数配置如下:
其中,decay 参数,与 AF2 不一致,但是 OpenFold 与 UniFold 都是 0.999,因此建议 0.999,修改:
ema:
decay: 0.999 # AF2 中是0.99,不建议修改
优化器 (Optimization) 参数配置,参考: