DeepMind 利用无监督学习开发 AlphaMissense,预测 7100 万种基因突变
类基因组共有 31.6 亿个碱基对,无时无刻不在经历复制、转录和翻译,也随时有着出错突变的风险。
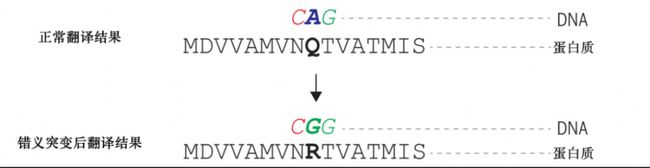
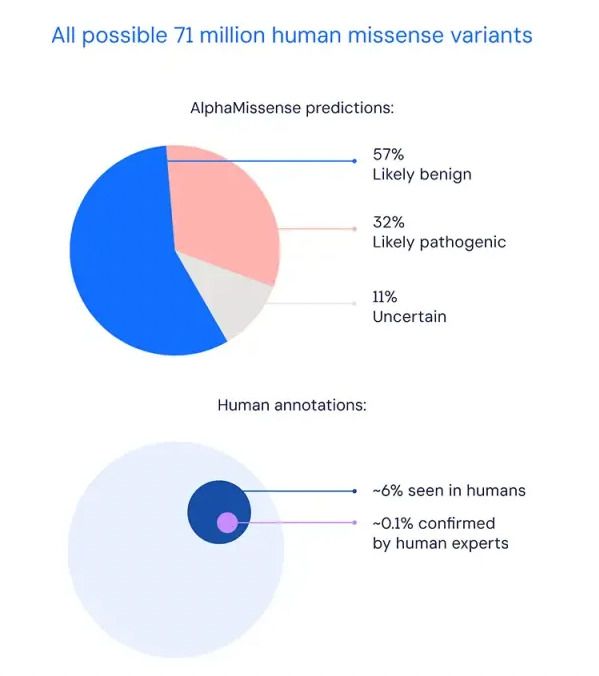
错义突变是基因突变中的一种常见形式,然而人类目前只观察到了其中的一小部分,能够解读的更是只有 0.1%。
准确预测错义突变的作用,对于罕见病、遗传病的研究和防治有着重要作用。这次,DeepMind 又出手了。
作者 | 雪菜
编辑 | 三羊、铁塔
人类基因组共有 31.6 亿个碱基对。这些碱基对每天会经历复制、转录、翻译,最终表达成为蛋白质,调控人类日常生理活动。
在如此庞大的工作量下,即使是精细的人体也很难做到毫无差错。稍有不慎,碱基对就可能配位错误,导致基因突变,日积月累甚至引发癌症。
错义突变 (Missense Mutation) 是一种常见的基因突变形式。 由于 DNA 中碱基突变,翻译得到的氨基酸发生了变化,最终导致整个蛋白质功能被破坏。
目前人类观察到了 400 多万种错义突变,但仅能将 2% 的错义突变归类为致病突变或是良性突变。
准确预测错义突变的作用能够加深人类对于罕见病的理解,并针对潜在的遗传病进行预防和治疗。 虽然变异效应多重分析 (MAVEs) 可以对蛋白质的突变进行系统的分析,并准确预测其临床效果,但这一方法需要大量的人力物力,难以对所有错义突变可能进行全面的分析。
为此,DeepMind 通过 AlphaFold 分析了蛋白质的整体结构,并结合弱标签学习和无监督学习开发了 AlphaMissense,对错义突变的后果进行了系统的分析。AlphaMissense 利用 ClinVar 数据集进行了验证,预测正确率达到 90%。
随后,AlphaMissense 对人类可能出现的 7,100 万种错义突变进行了预测,其中 32% 可能为致病性突变,57% 可能为良性突变。这些结果将极大促进分子生物学、基因组学、临床医学等学科的发展。这一成果已发表于「Science」。
相关成果已发表于「Science」
论文链接:
https://www.science.org/doi/10.1126/science.adg7492
实验过程
AlphaMissense:AlphaFold + 微调
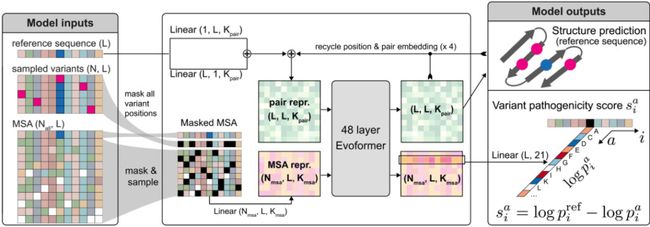
将一串氨基酸序列输入 AlphaMissense 后,它会对序列中任一氨基酸变化的致病性进行预测。 AlphaMissense 的实现和 AlphaFold 非常相似,只在架构上做了细微的调整。
AlphaMissense 的训练集来源广泛,但主要来自于人类和非人灵长类。其中,来源于人类的良性错义突变有 1,248,533 个,致病错义突变则从可能出现但尚未被观察到的 65,314,044 个突变中抽取。
AlphaMissense 的训练包括两步。首先,同 AlphaFold 一样,AlphaMissense 需要预测多序列对比 (Multiple Sequence Alignments) 中被随机掩码的氨基酸,进而预测单链蛋白质的结构,并进行蛋白质语言建模。
随后,研究人员利用人类蛋白质对 AlphaMissense 进行微调 (fine-tuning),并设定了模型的输出目标,即错义突变的致病性。
由于未被观察到的错义突变中存在相当数量的良性突变,但在训练过程中都将其归为了致病突变,因此 AlphaMissense 训练集的噪音很大。为了提升训练集的数量和质量,研究人员使用自蒸馏 (self-distillation) 的方式对数据进行了过滤。
临床数据验证:不同数据集中的表现
训练完成后,利用标注后的临床数据 (ClinVar 数据集)、罕见发育障碍患者中的新发突变 (de novo variants) 和 ProteinGym 中的 MAVE 结果对 AlphaMissense 进行验证。
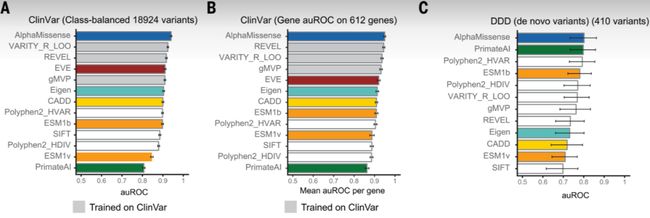
首先,研究人员对 AlphaMissense 在 ClinVar 数据集中的表现进行了评价。在对 18,924 个突变位点进行分析后,AlphaMissense 的 auROC 为 0.940,较之前最先进的进化模型 (EVE) 有所提升 (0.911)。
在对错义突变进行临床评价时,人们一般会关注特定疾病相关的基因。因此,分辨出这些基因中良性和致病的错义突变尤为重要。研究人员利用 AlphaMissense 对 ClinVar 中的 612 个基因进行分析,其 auROC 为 0.950,优于 EVE 的 0.921。
最后,研究人员分析了 AlphaMissense 在解密发育障碍 (DDD, Deciphering Developmental Disorders) 数据集中的预测结果。AlphaMissense 的 auROC 为 0.809,与 PrimateAI 的 0.797 相当。
A:对 ClinVar 中突变位点的分析;
B:对 ClinVar 中基因的分析;
C:对 DDD 数据集的分析。
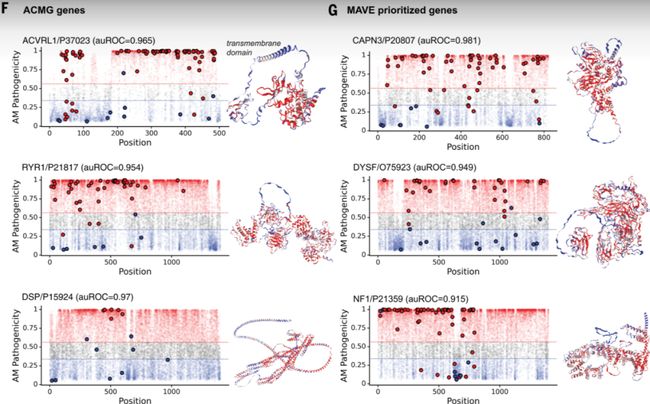
同时,AlphaMissense 对 Cancer Hotspots、ACMG (American College of Medical Genetics) 和其他 MAVE 数据的预测结果也较其他模型更为优异。上述结果说明,AlphaMissense 在多个数据集中表现优于现有模型。
总体预测性能:反映蛋白质突变趋势
用临床数据对 AlphaMissense 进行验证之后,研究人员利用 AlphaMissense 对 2.16 亿个氨基酸在人类常见的 19,233 个蛋白质中可能发生的突变进行了预测,最终得到了 7,100 万种错义突变的预测结果。
AlphaMissense 的致病性预测结果在 0-1 之间,越接近 1 说明致病可能性越高。由于绝大多数预测结果接近 0 和 1,因此 0.2 至 0.8 之间的数据可能不太准确。最终,他们将预测结果分为三类:可能致病、可能良性和无法确定。
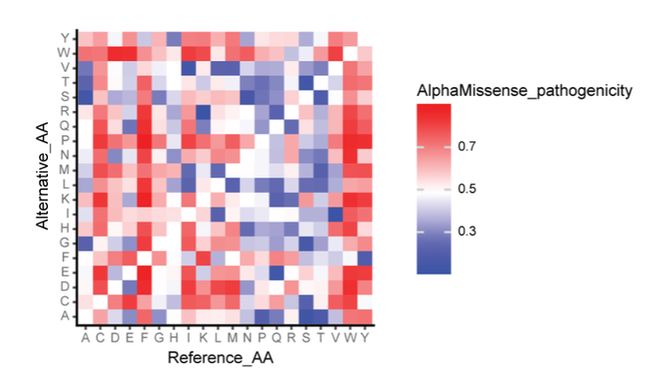
为对 AlphaMissense 的预测性能进行整体评价,研究人员计算了所有蛋白质的单个氨基酸致病性。结果显示,芳香族氨基酸和半胱氨酸的突变更容易引发疾病,与实际结果一致,因为这两种氨基酸起到了维持蛋白质结构的作用。
将 AlphaMissense 的预测结果和 AlphaFold 预测的蛋白质结构可视化之后,我们可以看到这些蛋白质的突变趋势。 比如,蛋白质结构紊乱的区域与良性突变的发生区域相对应,这与蛋白质组学的预测结果也是一致的。
左侧为 AlphaMissense 预测的致病性,可能致病的错义突变为红色,可能良性的错义突变为蓝色,已被收录于 ClinVar 数据集的突变以实心圆标注。右侧为 AlphaFold 预测的蛋白质结构,不同颜色表示这一区域突变致病性,与 AlphaMissense 相对应。
预测准确率:与 MAVE 结果一致性
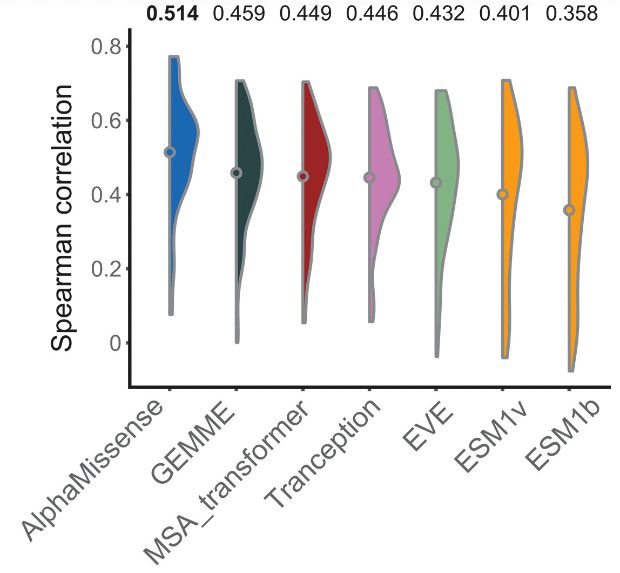
为调查 AlphaMissense 和 MAVE 结果之间的一致性,研究人员利用 AlphaMissense 对两组 MAVE 数据进行了分析。与其他预测方法相比,AlphaMissense 与 MAVE 数据最为接近。
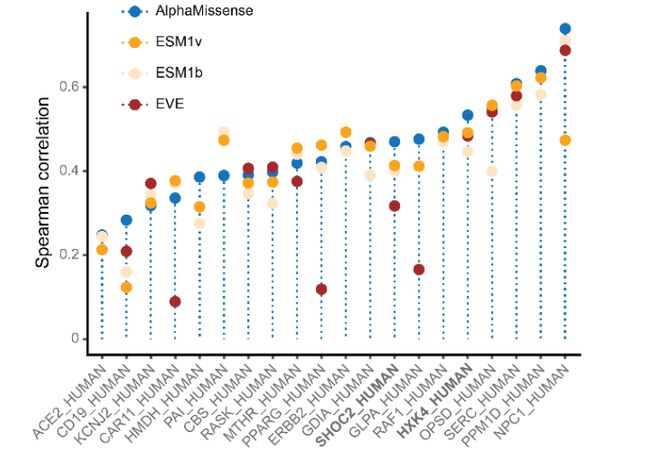
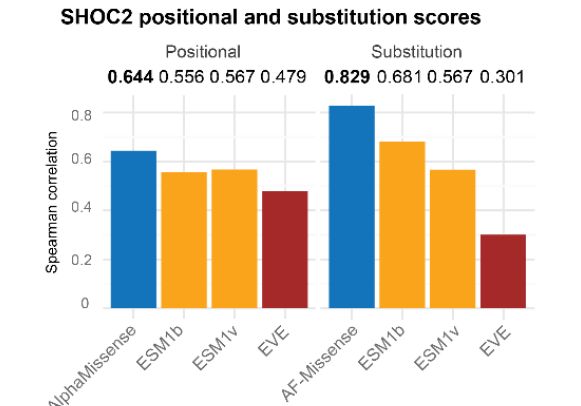
随后,他们又将 AlphaMissense 的预测数据和实验验证过的错义突变致病性进行了对比。SHOC2 蛋白可以与 MRAS 和 PP1C 蛋白形成复合物,激活 Ras-MAPK 癌症通路。AlphaMissense 与 MAVE 对这一突变和 Ras 癌症细胞的相关性进行了预测,得到的斯皮尔曼相关系数为 0.47,优于其他模型 (ESM1v: 0.41, ESM1b: 0.40, EVE: 0.32)。
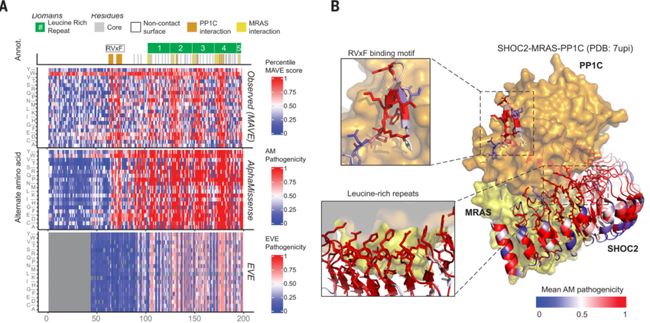
进一步的,研究人员探究了 AlphaMissense 对 SHOC2 蛋白不同区域氨基酸错义突变致病性的预测结果。在 SHOC2 的前 80 个氨基酸中,MAVE 预测第 63-74 号氨基酸突变是致病的,因为这一区域会通过 RVxF 与 PP1C 蛋白结合。AlphaMissense 是唯一一个识别出这一重要区域的模型。
A:不同模型对 SHOC2 蛋白前 200 氨基酸突变致病性的预测结果。自上而下分别为实际情况 (MAVE)、AlphaMissense 和 EVE;
B:SHOC2 蛋白(红色和蓝色)和 MRAS(黄色)、PP1C(金色)蛋白组成的复合体结构图。
而且,AlphaMissense 能够反映出不同种类氨基酸错义突变后的结果。 对于 SHOC2 蛋白而言,AlphaMissense 的预测结果与实际结果最为接近。
上述结果共同说明,AlphaMissense 的预测结果与 MAVE 相当,能够对基因错义突变的结果进行准确的预测。
最后,Deepmind 将这一模型和预测结果都开源公布在了社区中,希望这种结论能够为其他学科的研究提供帮助。
模型链接:
https://github.com/deepmind/alphamissense
基因突变:遥不可及又如影随形
提到基因突变,我们很容易想到 X 光、核辐射、亚硝酸盐等危险元素,或者是电影生化危机、绿巨人之中的桥段,觉得这些离我们过于遥远。诚然,我们在生活中接触到的辐射非常少,但基因突变还是发生在生活中的每时每刻,也切实地改变了我们的生活。
在生活中,我们不可避免地会接触到辐射源,比如太阳光。太阳光中 6% 的辐射来源于紫外线,而紫外线就是致癌因素之一,因此长时间暴晒会增加皮肤癌的危险。
即使不接触辐射源,DNA 在复制、转录、翻译的时候也不可避免地犯一些错误,引起基因突变,只是这些突变可能是良性的,或是被免疫机制及时清除了。
但同时,基因突变也为我们的生活提供了便利,尤其是在农业生产中。农作物突变体能够提高作物的产量,提升作物耐盐碱的能力,甚至帮助防治虫害。对这些突变体进行繁育筛选后,这些优良的特性就能保留下来,提升粮食产量。
然而,人体基因突变的可能性太多,我们目前了解到的不过沧海一粟。借助 AlphaMissense,我们能够对基因突变的结果进行相对可靠的预测,再加以反推,也许就能找到遗传病、罕见病背后的机制,为疾病的防治提供新方法。
同时,AlphaMissense 还为其他领域的研究提供了素材。也许不久之后,我们就能看到 AlphaMissense 对其他物种基因突变的解读,进而合理利用基因突变,让基因工程为我们的生活带来更多福祉。
参考链接:
[1]https://www.science.org/doi/10.1126/science.abj6987
[2]https://www.cshl.edu/discovery-of-new-stem-cell-pathway-indicates-route-to-much-higher-yields-in-maize-staple-crops/