R: fivz_pca_biplot的异常问题
(一) 椭圆置信区间边框颜色显示的问题:
原代码:
fviz_pca_biplot(NR.pca,
win.asp = 0.8,
# Fill individuals by groups
geom.ind = "point",
fill.ind = data_NR$Label, # 个体的填充
col.ind = "black", #habillage = data_NR$Label, 个体的边框及对应名称颜色
palette = c("#00AFBB", "#FC4E07"), # "jco"
pointshape = 21, pointsize = 3, #符号大小
addEllipses = TRUE,#添加浓度椭圆
repel = TRUE,#避免文字重叠
# Color variable by groups
geom.var = "arrow",
alpha.var = "contrib",
#col.var = "contrib",
gradient.cols = c("#00AFBB", "#FC4E07"), #variable的颜色也是按照palette进行修改的
select.var = list(cos2 = 30),#选择cos2响应前30
labelsize = 3,
legend.title = list(fill = "Species", color = "kmeans",alpha = "Contrib"),
title = "NR",
xlab = "PC1 (18.2%)", ylab = "PC2 (11.8%)",
) + font("title", size = 20, color = "black", face = "bold") +
font("subtitle", size = 12, color = "black", face = "bold") +
font("xlab", size = 14, color = "black", face = "bold") +
font("ylab", size = 14, color = "black", face = "bold") +
font("xy.text", size = 12, color = "black", face = "bold") +
border("black", 0.2) + rotate_x_text(0) +
font ("legend.title", size = 12, color = "black", face = "bold") +
font("legend.text", size = 10, color = "black", face = "bold") +
theme(plot.title = element_text(hjust = 0.45, vjust = -8)) +
theme(aspect.ratio = 0.5)
运行以上代码,得到下面的结果,图1中左侧椭圆的边框为蓝色,右侧边框颜色为红色。
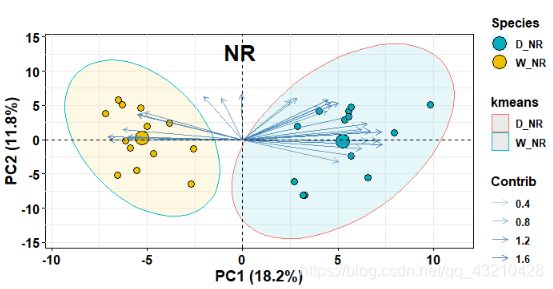
图1 椭圆置信区间边框颜色显示的问题
解决办法:
将上述代码中的< “col.var = “contrib”, >取消注释再次运行就正常,如下图:
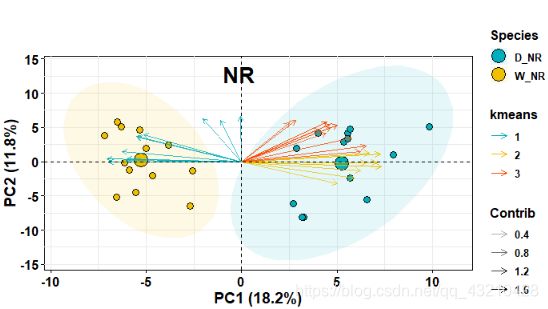
图2 椭圆置信区间边框颜色显示的问题
(二)PCA分析后的变量个数与原数据中变量个数不匹配
原代码:
# 不希望k-means算法依赖于任意变量单位,因此我们首先使用R函数scale()缩放数据
NR_scale <- scale(data_NR[,1:168])
# 估计最佳聚集数目
fviz_nbclust(NR_scale, kmeans, method = "wss")
#利用kmeans创建变量分组
#创建3组变量
set.seed(123)
res.km <- kmeans(var$coord, centers = 3, nstart = 25) #由于k均值聚类结果的最终结果对随机开始分配敏感,因此我们指定nstart = 25。这意味着R将尝试25种不同的随机开始分配,然后选择与簇内变异最小的结果相对应的最佳结果。R中nstart的默认值为1。但是,强烈建议以较大的nstart值(例如25或50)计算k均值聚类,以获得更稳定的结果。
grp <- as.factor(res.km$cluster)
# Color variables by groups
fviz_pca_var(NR.pca, col.var = grp,
palette = c("#0073C2FF", "#EFC000FF", "#868686FF"),
legend.title = "Cluster")
#绘图
NR_biplot <- fviz_pca_biplot(NR.pca,
win.asp = 0.8,
# Fill individuals by groups
geom.ind = "point",
fill.ind = data_NR$Label, # 个体的填充
col.ind = "black", #habillage = data_NR$Label, 个体的边框及对应名称颜色
palette = c("#00AFBB", "#EFC000FF", "#FC4E07"), # "jco"
pointshape = 21, pointsize = 3, #符号大小
addEllipses = TRUE,#添加浓度椭圆
repel = TRUE,#避免文字重叠
# Color variable by groups
geom.var = "arrow",
alpha.var = "contrib",
#col.var = grp,
gradient.cols = c("#00AFBB", "#EFC000FF", "#FC4E07"), #variable的颜色也是按照palette进行修改的
select.var = list(cos2 = 30),#选择cos2响应前30
labelsize = 3,
legend.title = list(fill = "Species", color = "kmeans",alpha = "Contrib"),
title = "NR",
xlab = "PC1 (18.2%)", ylab = "PC2 (11.8%)",
) + font("title", size = 20, color = "black", face = "bold") +
font("subtitle", size = 12, color = "black", face = "bold") +
font("xlab", size = 14, color = "black", face = "bold") +
font("ylab", size = 14, color = "black", face = "bold") +
font("xy.text", size = 12, color = "black", face = "bold") +
border("black", 0.2) + rotate_x_text(0) +
font ("legend.title", size = 12, color = "black", face = "bold") +
font("legend.text", size = 10, color = "black", face = "bold") +
theme(plot.title = element_text(hjust = 0.45, vjust = -8)) +
theme(aspect.ratio = 0.5)
NR_biplot
运行以上代码,一直报错下面内容:
Error in fviz(X, element = "var", axes = axes, geom = geom.var, color = col.var, :
The length of color variableshould be the same as the number of rows in the data.
分析原因:
变量的颜色数量与原数据中行数(代表变量个数)不同。
解决办法:
以上代码并没有问题,解决办法也很简单,重新检查数据发现,在利用kmeans进行分组时,res.km <- kmeans(var$coord, centers = 3, nstart = 25)这句代码里的分类数据集var$coord是另外一组数据的结果,而不是列表NR.pca中的结果,所以将该代码改为调用NR.pca中的数据即可res.km <- kmeans(NR.pca$var$coord, centers = 3, nstart = 25)。
(三) 手动定义颜色数量过少
在利用(二)中代码运行时,当其中“palette, gradient.cols”的颜色设置小于3个时,就会报错,如下所示:
错误: Insufficient values in manual scale. 3 needed but only 2 provided.
原因:
设置分组共3组,颜色数量设置小于3个时,不够分配而报错。
解决办法:
为其提供更多的颜色,大于等于3个颜色均可,会按照从前往后的顺序选择颜色进行绘图。
(四)Biplot只显示变量结果
原代码:
#Biplot
fviz_pca_biplot(XB.pca,
# Individuals
geom.ind = "point",
palette = "jco",
addEllipses = TRUE,
# Variables
gradient.cols = "RdYlBu",
legend.title = list(fill = "Species", color = "Contrib",
alpha = "Contrib")
)
运行以上代码时,结果如图3,由图3可知,只显示了变量(化合物)的结果(loading plot),而未显示出样本的结果(score plot)。
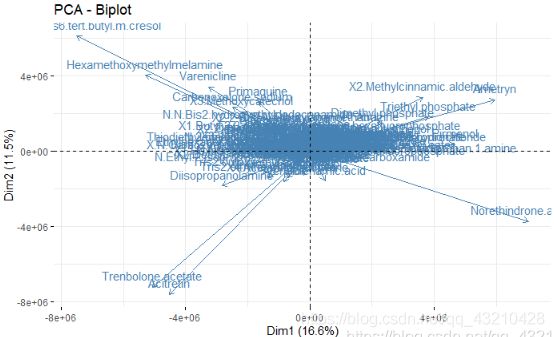
图3 Biplot
原因:
"XB.pca"是用其他R包进行标准化后的结果,所以此代码调用该数据集时未进行标准化。
解决方法:
利用factoextra进行PCA分析可视化时,需要将数据先利用factoMineR中的PCA()进行缩放scale(),才可以绘制图形。所以在运行上述代码前加上以下代码即可:
XB.pca <- PCA(data[,1:168], graph = TRUE)
- PCA() 默认进行scale()。
- PCA() 分析中不能含有字符,当导入原始数据具有名称或分组等非数值型信息时,可利用matrix的特性,调用数据列进行PCA() 分析(如:上述代码为调用data数据集中的第1到第168列数据,第169列为样本分组,故不调用)。
(五)fivz_pca_biplot与fivz_pca_ind中fill()的作用不同
代码1:
fviz_pca_ind(iris.pca,
geom.ind = "point",
fill.ind = iris$Species,
col.ind = "black", #habillage = data_NR$Label,
palette = c("#00AFBB", "#EFC000FF", "#FC4E07"),
pointshape = 20, pointsize = 3, #符号大小
addEllipses = TRUE
)
代码2:
fviz_pca_biplot(iris.pca,
win.asp = 0.8,
# Fill individuals by groups
geom.ind = "point",
fill.ind = iris$Species, # 个体的填充
col.ind = "black", #habillage = data_NR$Label, 个体的边框及对应名称颜色
palette = c("#00AFBB", "#EFC000FF", "#FC4E07"), # "jco"
pointshape = 21, pointsize = 3, #符号大小
addEllipses = TRUE
)
运行代码1时,报错内容如下:
Error in if (fill %in% names(data) & is.null(add.params$fill)) add.params$fill <- fill :
参数长度为零
提示内容为"fill.ind"的内容为空,检查对比以后发现,代码1与代码2中引用的分组数据分别为“data_NR$Label” 与“data_NR$label”,前面为手动输入有误, 将"Label"改为小写"label"问题解决。
再次运行代码1与代码2,结果分别为图4与图5:
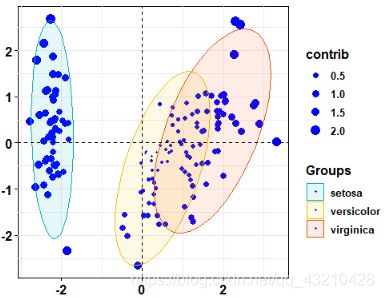
图4 代码1的运行结果
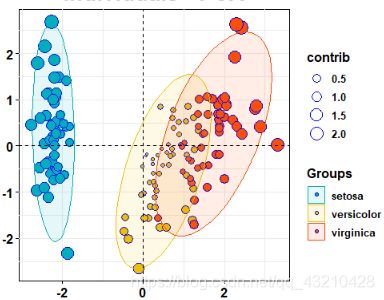
图5 代码2的运行结果
从上图可知,图1的三组结果均为蓝色,图2的三组则根据分组显示不同颜色,检查代码发现pointshape的设置值不一样,代码1中pointshape = 20,而代码2中pointshape = 21。
利用以下代码查询R包“ggpubr”中的所有形状组合,
ggpubr::show_point_shapes()
结果如图6所示,由图6可知,每个数字编码都代表一种形状,21表示的为空心,20表示的为实心,导致上述运行结果不一致,可根据个人需要多尝试几个数字,通过观察出图效果选择适合的形状。
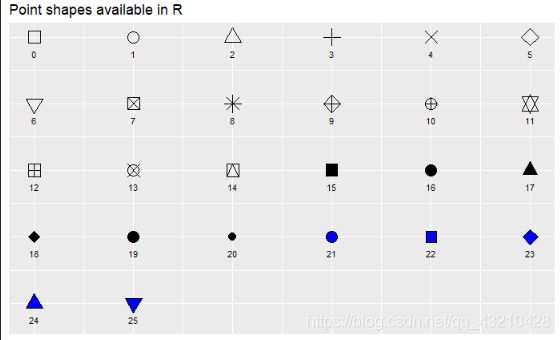
图6 R包中可用的点的形状
参考内容:
- Unexpected behavior of fviz_pca_biplot___color(连续变量/分组变量)的设置问题
- K-Means Clustering in R: Algorithm and Practical Examples
- PCA - Principal Component Analysis Essentials