【论文精读】Pairwise learning for medical image segmentation
Published in: Medical Image Analysis 2020
论文:https://www.sciencedirect.com/science/article/abs/pii/S1361841520302401
代码:https://github.com/renzhenwang/pairwise_segmentation
目录
Published in: Medical Image Analysis 2020
摘要
一、主要亮点
二、问题提出
三、网络结构
1. CFCN
2. ![]()
四、实验部分
1. 实验表格
2.ASSD损失函数
总结
完整网络框架代码
摘要
本文提出了一种共轭完全卷积网络conjugate fully convolutional network (CFCN),其中输入成对样本来捕获丰富的上下文表示,并通过融合模块相互引导。为了避免由少量训练样本引入的类内异质性和边界模糊带来的过拟合问题,我们建议明确利用标签空间的先验信息,称为代理监督。我们进一步将CFCN扩展为紧耦共轭全卷积网络compact conjugate fully convolutional network(![]() ),与CFCN相比,它只需要一个头部来拟合代理监督,而不需要额外的两个解码器分支来拟合输入对的地面真值。
),与CFCN相比,它只需要一个头部来拟合代理监督,而不需要额外的两个解码器分支来拟合输入对的地面真值。
先了解一下孪生神经网络(Siamese network)
度量学习:metric learning 是通过特征变换得到特征子空间,通过使用度量学习,让类似的目标距离更近(PULL),不同的目标距离更远(push),也就是说,度量学习需要得到目标的某些核心特征(特点),比如区分两个人,2只眼睛1个鼻子-这是共性,柳叶弯眉樱桃口-这是特点。
一、主要亮点
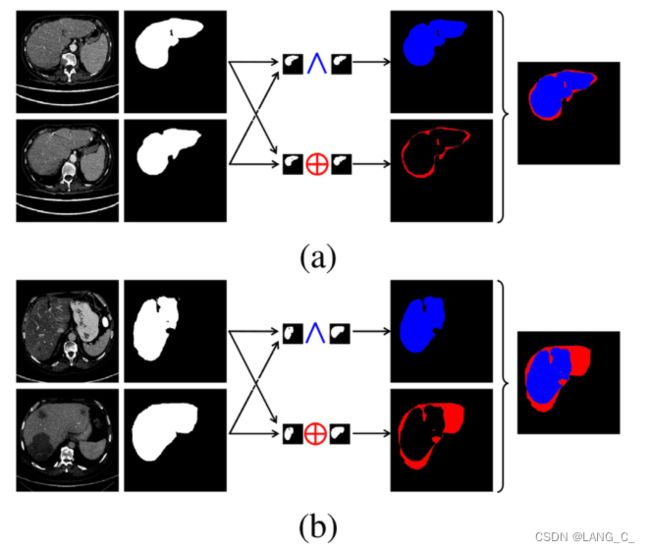
- 输入的是成对切片,训练阶段,将一个切片与不同的患者内/患者间切片进行配对,每个像素的标签由其自身和配对切片中的另一个像素(位于同一位置)确定。
- 提出共轭完全卷积网络,在三个支路上做监督学习,解决类内异质性和边界模糊的问题。
二、问题提出
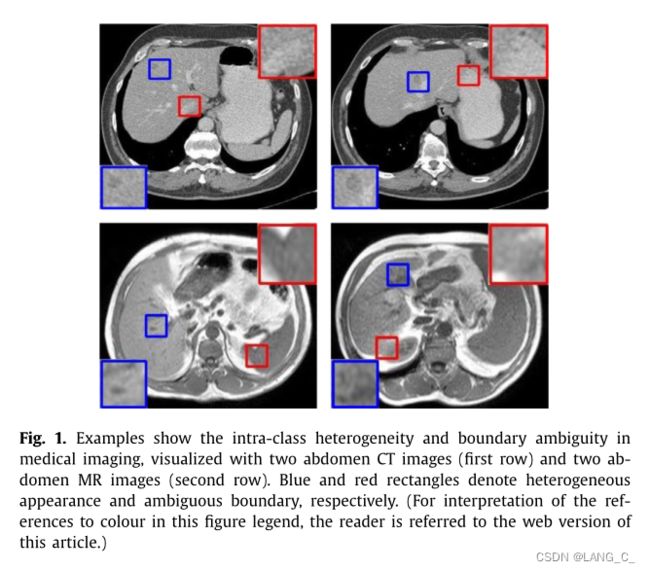 如图所示,通过两个腹部CT图像(第一排)和两个腹部MR图像(第二排)的例子显示了医学成像中的类内异质性和边界模糊。蓝色矩形和红色矩形分别表示外观不均匀和边界模糊
如图所示,通过两个腹部CT图像(第一排)和两个腹部MR图像(第二排)的例子显示了医学成像中的类内异质性和边界模糊。蓝色矩形和红色矩形分别表示外观不均匀和边界模糊
针对医学图像分割中训练数据有限、类内异构和边界模糊的问题
三、网络结构
1. CFCN
[MICCAI2019]Pairwise Semantic Segmentation via Conjugate Fully Convolutional Network
本文主要参考上述文献延伸得到的,网络框架如下
2. 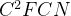
四、实验部分
1. 实验表格
2.ASSD损失函数
Average symmetric surface distance博客参考
Metric评价指标-图像分割之平均表面距离(Mean surface distance )
总结
本文提出了一种新的基于有限训练样本的医学图像分割框架。特别地,专注于解决由类内异质性和边界模糊引起的具有挑战性的问题。扩展前期工作,从两个方面改进了框架:首先,将CFCN扩展为一个一般的双学习框架,其中代理监督作为网络的全局约束,以适应标签空间的固有先验知识。除了在LiTS数据集上的二值分割外,进一步将CFCN扩展到多类别分割,用于基准数据集CHAOS上的多器官分割,结果表明,我们的CFCN可以在所有比较方法中取得最先进的结果。
其次,将CFCN扩展为一个紧凑的架构C2FCN,它可以在测试阶段用可以忽略不计的附加参数和计算开销来改进任何现成的分割网络。具体来说,采用DeepLabv3+作为基线,得到的C2FCN在LiTS数据集上取得了较好的结果(见表1),在Sub-CHAOS数据集上取得了优于CFCN的结果。然而,在训练过程中,C2FCN的参数数量和计算开销都大大低于CFCN。更重要的是,本文提出的C2FCN在训练阶段仅用一个头部学习代理监督中隐含的逻辑关系,在测试阶段通过学习的逻辑关系推断分割概率。与CFCN相比,C2FCN中不需要两个解码器来显式拟合输入对的ground truth,说明通过深度模型学习一般关系是可行的和有潜力的。
值得一提的是,本文采用了提出的两两分割框架来解决类内异质性和边界模糊所带来的挑战。然而,通过明确地设计![]() 函数,可以利用更多的先验信息。未来的另一个研究方向是将所提出的CFCN和C2FCN应用于更多的分割场景,特别是管状结构的自动分割,如血管或小肠。通常在三维空间中折叠,且形状先验很难通过患者内相邻切片进行建模。此外,将方法扩展到三维模型将是作者未来的研究方向。
函数,可以利用更多的先验信息。未来的另一个研究方向是将所提出的CFCN和C2FCN应用于更多的分割场景,特别是管状结构的自动分割,如血管或小肠。通常在三维空间中折叠,且形状先验很难通过患者内相邻切片进行建模。此外,将方法扩展到三维模型将是作者未来的研究方向。
综上所述,本文提出了一种新的针对有限训练样本的医学图像分割框架。为了解决医学影像中类内异质性和边界模糊的问题,提出了一种代理监督,将标签空间中的先验信息显式编码,并在训练阶段将其作为网络的全局约束。特别是,我们提出了一种新的分割通过C2FCN范式,在网络旨在学习输入对之间的逻辑关系,而不是直接适合地面真理传统FCNs一样的目标,和分割测试样本的概率是推断的逻辑关系。实验结果表明,在训练数据量有限的情况下,所提出的两两分割方法能够显著提高分割精度。
完整网络框架代码
# -*- coding: utf-8 -*-
import torch
import torch.nn as nn
import torch.nn.functional as F
from models.sync_batchnorm.batchnorm import SynchronizedBatchNorm2d
from models.aspp import build_aspp
from models.decoder import build_decoder
from models.backbone import build_backbone
from models.fusion import build_fusion
from models.attention_fusion import build_attention_fusion
class PairwiseDeepLab(nn.Module):
def __init__(self, backbone='resnet18', in_channels=3, output_stride=16,
num_classes=1, aux_classes=3, sync_bn=True, freeze_bn=False,
pretrained=False, fusion_type='fusion', is_concat=False, **kwargs):
super(PairwiseDeepLab, self).__init__()
if backbone == 'drn':
output_stride = 8
if sync_bn == True:
BatchNorm = SynchronizedBatchNorm2d
else:
BatchNorm = nn.BatchNorm2d
self.backbone = build_backbone(backbone, in_channels, output_stride, BatchNorm, pretrained)
## branch1
self.aspp = build_aspp(backbone, output_stride, BatchNorm)
self.decoder = build_decoder(num_classes, backbone, BatchNorm)
## branch2
# self.br2_aspp = build_aspp(backbone, output_stride, BatchNorm)
# self.br2_decoder = build_decoder(num_classes, backbone, BatchNorm)
## fusion
self.fusion_type = fusion_type
if self.fusion_type == 'attention_fusion':
print('fusion_type is attention_fusion')
self.fusion = build_attention_fusion(aux_classes, backbone, BatchNorm, is_concat=is_concat)
elif self.fusion_type == 'fusion':
print('init fusion_type')
self.fusion = build_fusion(aux_classes, backbone, BatchNorm, is_concat=is_concat)
else:
raise NotImplementedError
if freeze_bn:
self.freeze_bn()
def forward(self, x1, x2):
## branch1
br1_x, low_level_feat1 = self.backbone(x1)
# print(br1_x.shape, low_level_feat1.shape)
br1_x = self.aspp(br1_x)
br1_out = self.decoder(br1_x, low_level_feat1)
br1_out = F.interpolate(br1_out, size=x1.size()[2:], mode='bilinear', align_corners=True)
# br1_out = br1_out.permute(0, 2, 3, 1).contiguous()
## branch2
br2_x, low_level_feat2 = self.backbone(x2)
br2_x = self.aspp(br2_x)
br2_out = self.decoder(br2_x, low_level_feat2)
br2_out = F.interpolate(br2_out, size=x2.size()[2:], mode='bilinear', align_corners=True)
# br2_out = br2_out.permute(0, 2, 3, 1).contiguous()
## fusion
fusion_x = self.fusion(br1_x, low_level_feat1, br2_x, low_level_feat2)
fusion_x = F.interpolate(fusion_x, size=x2.size()[2:], mode='bilinear', align_corners=True)
# fusion_x = fusion_x.permute(0, 2, 3, 1).contiguous()
return br1_out, br2_out, fusion_x
def freeze_bn(self):
for m in self.modules():
if isinstance(m, SynchronizedBatchNorm2d):
m.eval()
elif isinstance(m, nn.BatchNorm2d):
m.eval()
def get_1x_lr_params(self):
modules = [self.backbone]
for i in range(len(modules)):
for m in modules[i].named_modules():
if isinstance(m[1], nn.Conv2d) or isinstance(m[1], SynchronizedBatchNorm2d) \
or isinstance(m[1], nn.BatchNorm2d):
for p in m[1].parameters():
if p.requires_grad:
yield p
def get_10x_lr_params(self):
modules = [self.aspp, self.decoder, self.fusion]
for i in range(len(modules)):
for m in modules[i].named_modules():
if isinstance(m[1], nn.Conv2d) or isinstance(m[1], SynchronizedBatchNorm2d) \
or isinstance(m[1], nn.BatchNorm2d):
for p in m[1].parameters():
if p.requires_grad:
yield p
import time
start = time.time()
if __name__ == "__main__":
model = PairwiseDeepLab(backbone='resnet18', output_stride=16, in_channels=5,
pretrained=False, fusion_type='attention_fusion')
model.eval()
input = torch.rand(1, 5, 256, 256)
output = model(input, input[:])
print(output[0].size(), output[1].size(), output[2].size())
print("Total paramerters: {}".format(sum(x.numel() for x in model.parameters())))
end = time.time()
print(end-start)