生物信息学中引入NLP的相关思想
这是鄙人写的第一篇博客,还有许许多多的问题,希望大家多多指正
目录
- 生物信息学与NLP是什么?
-
- 什么是生物信息学?
- 什么是NLP?
- 生物信息学和NLP有何关联?
-
- 生物信息学和NLP的相似部分
- 生物信息学研究过程(以EPI预测为例)
- 在生物信息学中引入NLP思想
- 参考文献
生物信息学与NLP是什么?
什么是生物信息学?
生物信息学(Bioinformatics)是研究生物信息的采集、处理、存储、传播,分析和解释等各方面的学科,也是随着生命科学和计算机科学的迅猛发展,生命科学和计算机科学相结合形成的一门新学科。它通过综合利用生物学,计算机科学和信息技术而揭示大量而复杂的生物数据所赋有的生物学奥秘。 ————百度百科(生物信息学)
其实用通俗的话来说,生物信息学就是用特定的计算机算法(Computer Algorithms)来解决“大数据”的生物问题。例如,大家都知道DNA是双螺旋结构,并且由且只A、C、G、T四种嘌呤组成,那么我们就可以用一个字符串来储存一个DNA序列信息。众所周知,DNA通过调控蛋白质的转录从而来调控生命体的一切生命活动,对生物遗传变异等具有最关键的作用,那么,如果能够很好的理解DNA序列信息就能对生物领域做出极大的贡献。就比如,生物信息学中比较常见的几个方向:预测蛋白质与疾病之间的联系、预测mRNA与蛋白质之间的联系、细胞分类、预测增强子与启动子之间的联系等,这些研究既推动了计算机领域的发展又推动了生物领域的发展,甚至于前两年肆虐全球的COVID-19也有许多学者在通过计算机手段研究其生物信息从而对解决这场瘟疫起了十分大的推动作用。
什么是NLP?
Natural language processing (NLP) is a branch of artificial intelligence (AI) that enables computers to comprehend, generate, and manipulate human language. Natural language processing has the ability to interrogate the data with natural language text or voice. This is also called “language in.” Most consumers have probably interacted with NLP without realizing it. For instance, NLP is the core technology behind virtual assistants, such as the Oracle Digital Assistant (ODA), Siri, Cortana, or Alexa. When we ask questions of these virtual assistants, NLP is what enables them to not only understand the user’s request, but to also respond in natural language. NLP applies both to written text and speech, and can be applied to all human languages. Other examples of tools powered by NLP include web search, email spam filtering, automatic translation of text or speech, document summarization, sentiment analysis, and grammar/spell checking. For example, some email programs can automatically suggest an appropriate reply to a message based on its content—these programs use NLP to read, analyze, and respond to your message.
————What is Natural Language Processing?(OCI)
自然语言处理(Nature Language Processing,NLP)于20世纪中旬被提出,当时主要应用于机器翻译方面,之后随着机器学习的发展,NLP逐渐开始了以统计学为基础的机器学习发展,再随着近几年深度学习的风靡,NLP也发展的极为迅速,NLP在深度学习神经网络应用极为契合。一开始只是使用简单的深度学习来获取特征来满足一些统计学的需求,之后通过建模、大模型端对端的训练在翻译、文献阅读、搜索甚至于AI智能问答的方面得到了极大的进展。
简单的来说,NLP就是对人类所使用的自然语言进行处理,将其用计算机语言表示出来并处理获得一些信息从而将这些信息用于某些特定场景。例如文本分类,就是将获得的文本进行语义标注,用大量数据进行模型训练,从而使用模型对文本进行分类。
生物信息学和NLP有何关联?
生物信息学和NLP的相似部分
其实两者从对数据的处理、模型训练再到测试结果都是十分相似的,在计算机内部,两者都是对字符串进行处理,只不过将原始数据转化为字符串的过程不同而已,也就是数据预处理的过程不同,但是处理结束后将数据转化为数学矩阵进行机器学习或者深度学习方法训练的流程是相似的。
生物信息学研究过程(以EPI预测为例)
生物信息学中有许多的方向,但是要说到与NLP联系最为紧密的便是EPI预测了
什么是EPI呢?
EPI全称为Enhancer-Promoter Interactions,即增强子与启动子之间的联系。增强子是DNA序列上一个片段,它可以对基因转录等起一定的增强作用。而启动子也是DNA序列上的某一个片段,它的作用是启动某段基因的转录等,一般位于基因的起始位点。两者都对基因转录调控起着关键性作用,启动子一般位于目标基因附近,但是增强子却有可能离目标基因片段十分远,甚至有可能跨染色体,由于DNA的空间折叠等从而影响到目标基因。而且启动子与增强子并不是一对一的联系,它们是一对多和多对一的。
由此可见,我们难以去判断哪两个增强子和启动子是否有联系
传统的生物实验用大量的人力物力去研究,花费大量的时间去确定两者之间是否有联系,而生物信息学自然要以计算性的方法为主。
如今计算性的方法主要有两种:一是以基因组信号为标注来训练模型,另一种则是仅仅使用基因序列信息来预测。前者更多的是使用传统机器学习的方法来预测,并且如何选择更好的基因组信号则需要更好的生物知识,后者则是使用常见的深度学习框架来实现,这与NLP方向十分相似。
接下来我们主要介绍以基因序列信息来预测EPI
Singh et al. (2016)首次提出了以序列信息来进行EPI预测1,由于基因序列信息为以A、C、G、T组成的字符串,我们需要将其转化为数学矩阵进行相应的深度学习模型搭建。传统的方法是使用one-hot编码。如图:

我们一般增强子取4000bp,启动子取3000bp,如果单纯的对增强子或启动子矩阵(4 x 4000或4 x 3000)进行模型训练效果并不好。而且,如果构建语义库就会产生一个十分严重的问题——维度爆炸。我们并不是只使用固定的这4000个bp,如果构建语义库我们需要所有情况,如果只简单实用one-hot编码来构建语义库1个bp只有 4 1 4^1 41 = 4四种情况(A、C、G、T),2个bp就是 4 2 4^2 42 = 16,,3个bp就是 4 3 4^3 43 = 64,4000个bp就是 4 4000 4^{4000} 44000已经无法计算了。那么,我们就需要用的生物信息学中常见的编码方式了——k-mer编码。
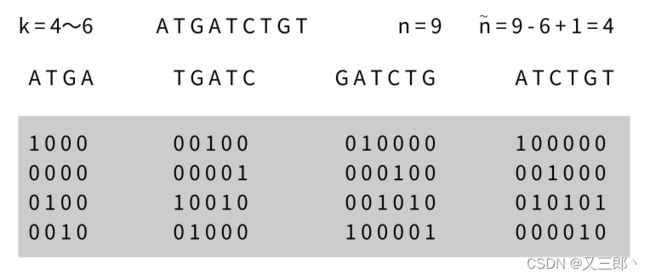
k-mer编码就是将每k个bp DNA分为一个部分,比如当k=6时,每6个bp为一部分,然后往后挪一个bp,那么一个增强子就被分为了3995(4000-6+1)个片段,然后再将这3995个片段链接起来。如上图的ATGATCTGT就被分为了ATGATC、TGATCT、GATCTG、ATCTGT四个片段了,再将其矩阵链接起来。如图:

在设计和训练模型中我们一般不使用固定的k值而是使用多个值并且满足离散均匀分布(若随机变量有n个不同的值,其中每个值出现的概率相同),当使用k-mer时,若k=4~6,可能会出现ATGA、TGATC,GATCTG,ATCTGT那么一共就有 4 4 4^4 44 + 4 5 4^5 45 + 4 6 4^6 46 = 5376种情况。如图(其中 n ~ \widetilde{n} n 为统计学中平均值):
这样,维度爆炸的问题就解决了,但是又有一个很棘手的问题,那就是使用k-mer编码每个片段之间都是相同的距离,这使得所有mer之间失去了相关性。
之后,NLP中一个最常见的思想被融入了,产生了一个强大的预训练模型——dna2vector
2017 Ng P2提出了这个模型。上面我们提到了,使用one-hot编码+k-mer编码构建语义库需要86016个4 x 6的矩阵,但这样会失去每个片段之间的相关性,那么,dna2vec模型就基于word2vec提出了一个方法来获得所有片段之间的相关性。
word2vec是NLP领域十分常见的模型,网上相关的文章十分多,我了解的也不是十分透彻,这里贴一个大佬的文章,有兴趣的小伙伴可以去钻研钻研:https://blog.csdn.net/v_JULY_v/article/details/102708459
dna2vec是基于word2vec提出的,word2vec是有两个模型的:CBOW(Continuous Bag-of-Word)和Skip-gramm。dna2vec主要是基于Skip-gram模型。
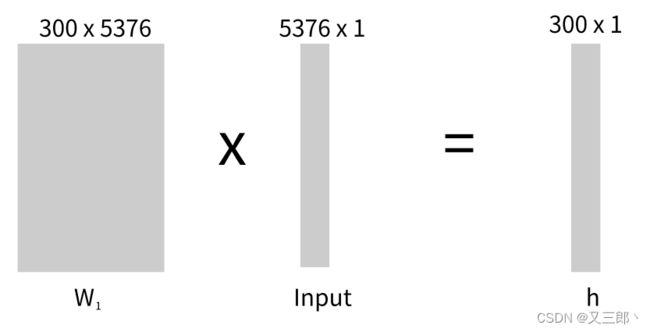
以上,我们构建语义库的时候就不用使用二维矩阵了,之前使用one-hot编码导致矩阵中有大量的0,我们可以换个思想来存储DNA序列信息,例如,当k=6时,我们有 4 6 4^6 46 = 4096种情况,分别是AAAAAA、AAAAAC、AAAAAG、······、TTTTTG、TTTTTT,那么我们可以使用一个维度为4096的向量来保存其中一种情况,比如AAAAAA:1 0 0 0 0 0····0 0 0,AAAAAAC:0 1 0 0 0 0 ···· 0 0 0,TTTTTT:0 0 0 0 ·····0 0 1,如果k=4~6,我们可以使用一个维度为5376的向量来表示。如图:

其中,我们的权重矩阵大小就可以设计为5376 x 300,那么,如果我们输入增强子序列(4000bp),通过dna2vec, n ~ \widetilde{n} n = 4000-6+1 = 3995,我们会获得3995个维度为5376的词向量,然后通过权重矩阵运算和隐藏层获得3995个处理过的向量并将其连接起来成为一个3995 x 300的矩阵。如图(其中W为权重矩阵, W 2 W_2 W2为 W 1 W_1 W1的转置矩阵,h为隐藏层):

至此,dna2vec对数据的预处理结束了,之后便可以通过深度学习神经网络框架进行模型训练,比如EPIVAN3便是先经过dna2vec对数据进行嵌入,然后经过1D CNN + Bi-GRU + Attention网络训练,最后获得预测结果。如图:
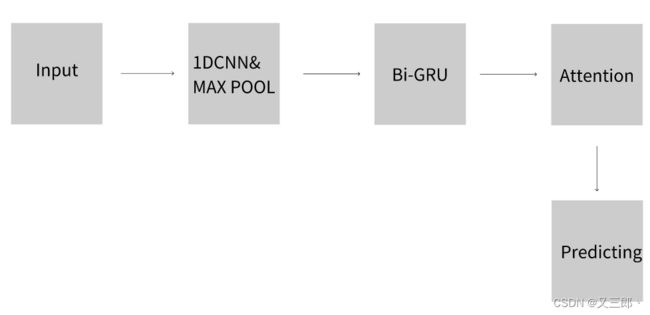
最终的预测是个二分类问题,使用逻辑回归Relu激活函数预测增强子与启动子是否有关联。
在生物信息学中引入NLP思想
NLP领域比较常见的一个应用场景是标签的多分类,其工作流程与EPI预测相似,也是先通过模型预训练分词、构建词典、嵌入词向量,然后搭建训练模型,最终使用softmax函数将结果归一化从而获得分类结果。
其实不仅NLP领域与生物信息学联系密切,在许多无监督机器学习,比如细胞分蔟等,CV图像识别,比如对CT照片进行分析预测药物与疾病效果等都有着十分密切的联系,所以,生物信息学已经在计算机领域有着举足轻重的地位了,以后的研究不能将其仅仅作为生物领域的研究,更多的要将计算机领域的思想融入进去,两者结合,两个领域同步发展!
参考文献
Singh S, Yang Y, Póczos B, et al. Predicting enhancer-promoter interaction from genomic sequence with deep neural networks[J]. Quantitative Biology, 2019, 7: 122-137. ↩︎
Ng P. dna2vec: Consistent vector representations of variable-length k-mers[J]. arXiv preprint arXiv:1701.06279, 2017. ↩︎
Hong Z, Zeng X, Wei L, et al. Identifying enhancer–promoter interactions with neural network based on pre-trained DNA vectors and attention mechanism[J]. Bioinformatics, 2020, 36(4): 1037-1043. ↩︎