转座子的简介
1. 转座子的概述
芭芭拉·麦克林托克(Barbara McClintock,1902年6月16日-1992年9月2日),美国著名女性细胞遗传学家,她首次发现并重点研究了玉米转座子
一个转座子由基因组的一个位置移动到另一个位置的过程称为转座。促进转座的酶称转座酶。转座子常常编码它自己的转座酶,并且每次移动时携带转座必需的基因一起在基因组内跃迁。
1.1 转座作用具有什么遗传学效应呢?
(1)转座插入可能导致插入突变;
(2)转座也可能产生新的基因;
(3)或者染色体畸变;
(4)转座也是引起生物进化的素材;
(5)但有时候转座也可能没有任何影响,这主要取决于转座子的功能和其插入的位点。
1.2 真核生物DNA转座子
如hAT 超家族,CACTA超家族,微小反向重复转座子(MITEs)。果蝇的P 型父系与M 型母系的"杂种不育"也是转座子引起的,比较复杂
1.3 转座子结构
转座子转座机制如下:插入序列两端有20-40个核苷酸的反向重复顺序(下图红色区域GGGGTCTG)。转座发生必须具有编码转座酶的基因。受体DNA上的靶序列转座后在转座子两侧形成正向重复序列(下图绿色区域AATTC)
1.4 转座子类型
根据转座中间体也将转座子分为I型转座子和II型转座子,它们的区别在于I型转座中间体是RNA,该型转座子会先被转录为RNA,然后该RNA被逆转录,再次成为DNA,才被插入到目标位点中。因此转座一次相当于复制粘贴一次基因。II型转座中间体是DNA,就是它本身。转座的过程是不复制自身的,转座一次相当于剪切后粘贴
2. 转座子的分布
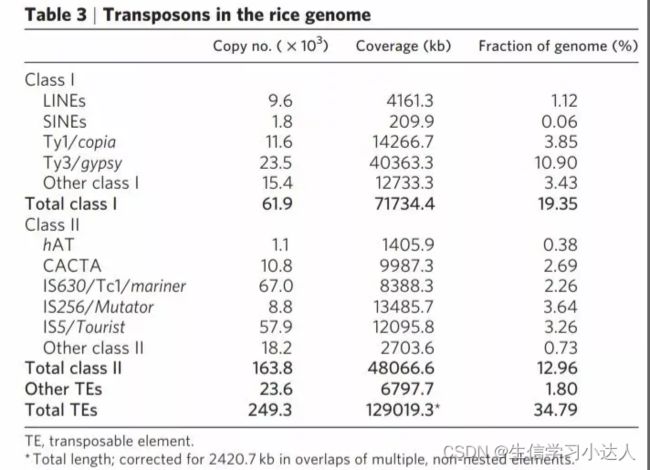
2005年,Nature 发表了水稻的基因组。水稻中转座子序列至少占据了整个基因组的 35%,并且水稻基因组中几乎含有所有已知的转座子家族。具体分析后发现,水稻8号染色体和12号染色体的转座子含量最高,分别为38.0%和38.3%;1号、2号以及3号染色体的转座子含量相对较低,分别为31.0%、29.8%和29.0%。
水稻基因组中II型转座子的含量较多,约是I型转座子的两倍。但是,I型转座子的核苷酸数量要远远大于II型转座子,因为I型转座子通常比较大,而II型转座子一般都较小。
水稻中I型转座子主要集中在基因稀少的区域和异染色质区域,比如着丝粒和中心体附近;不过,仍有一些I型转座子和II型转座子分布在基因密集和重组率较高的染色体区域。
总体来看,水稻转座子的分布和转座子的大小有一定的相关性:较小的转座子主要分布在基因相对密集以及重组率高的区域,而较大的转座子则集中在着丝粒和中心体附近。
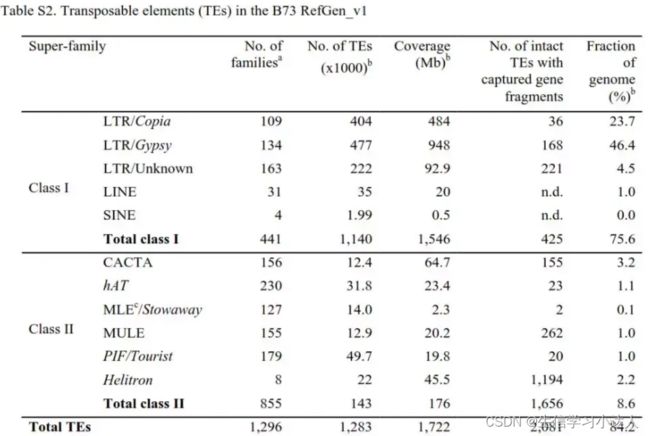
2009年,玉米的基因组也在Science上公布。由于转座子最早是在玉米中发现的,许多转座子家族也都是首先在玉米中鉴定到的,因此玉米中转座子的含量及分布规律自然更加吸引人们的注意。让人们吃惊的是,转座子这种一度被认为是“垃圾DNA”的物质,竟然占据了玉米基因组的近85%。和拟南芥以及水稻不同,玉米中许多转座子都分布在基因密集以及染色体重组率较高的染色体末端区域,这暗示着玉米中转座子在调控基因功能方面可能非常活跃。
3.转座子的鉴定和分类
真核生物转座子鉴定、分类和注释的 3 个步骤:(1)重复序列库的构建; (2)重复序列的校正和分类; (3)基因组注释。详情参考此 文章来源
根据转座介质的不同, 转座子可以分为两大 类:第一类是逆转录转座子, 通过RNA媒介的逆转 录进行转座; 第二类是DNA转座子, 它们以DNA为 媒介进行转座[53]。
其中逆转录转座子根据不同的转座机制又可以分为不同的亚类 [54], 长末端重复 (Long terminal repeat, LTR)逆转录转座子和非长末端重复(Non-long terminal repeat, Non-LTR)逆转录转座子。
前者LTR-TE在类病毒颗粒中由mRNA介质逆转录而 来, 以双链DNA分子插入到整合位点, 而Non-LTR-TE后者由 mRNA 逆转录而来, 直接整合到插入位点。
自主的 Non-LTR也称为长散在核元件 (Long interspersed nuclear elements, LINEs), 非自主的Non-LTR称为短散在核元件(Short interspersed nuclear elements, SINEs), LINEs负责SINEs的转座[55]。LINEs和SINEs的 起源进化关系目前还不清楚[56]。
DNA转座子以“剪切和粘贴”的机制进行转座, 此类元件通过交错双链裂解(Staggered doubled stranded cleavage)从染色体上切除下来, 重新插入到基因组的其他位置[54]。 DNA转座子根据不同的结构特征又进一步分为末端反向重复(Terminal inverted repeats, TIRs)、微型反向重复转座元件(Miniature inverted repeat transposableelements, MITEs)和Helitrons转座子等。转座子不同类别的结构特征, 可以参考相应的综述文献, 如[7] 等。
参考文献:
1.Arabidopsis Genome Initiative. (2000). Analysis of the genome sequence of the flowering plant Arabidopsis thaliana. nature, 408(6814), 796.
2.Sasaki, T. (2005). The map-based sequence of the rice genome. Nature, 436(7052), 793.
3.Schnable, P. S., Ware, D., Fulton, R. S., Stein, J. C., Wei, F., Pasternak, S., ... & Minx, P. (2009). The B73 maize genome: complexity, diversity, and dynamics. Science, 326(5956), 1112-1115.
神奇的转座子:植物中转座子的分布 - 知乎
真核生物转座子鉴定和分类计算方法 (chinagene.cn)